欢迎关注”生信修炼手册”!
评估SNP遗传力有两种方法LDSC和GREML, 本文介绍下GREML评估遗传力的方法。在GCTA软件中,其核心就是如下所示的线性混合模型

其中y表示表型,b表示固定效应协变量的系数,u表示随机效应自变量的系数, 这里的随机效应指的就是所有SNP位点对表型的效应,e表示随机误差。其中u和e服从如下所示的正态分布


表型方差可以用如下公式表示

在GCTA中,W的计算公式如下
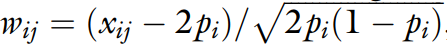
在之前的文章中,我们介绍了GCTA对于样本遗传相似度的定义,公式如下
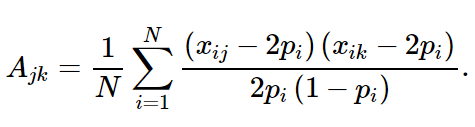
可以推导出如下关系
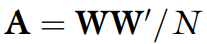
定义所有SNP位点的方差如下

则表型变异V的公式变换如下

定义SNP遗传力的计算公式如下

此时只需要通过REML算法对线性混合模型的参数进行估计,然后求解上述方差,即可计算出了SNP遗传力了。软件实际操作的代码如下

首先构建GRM矩阵,下游分析都是需要输入GRM矩阵的,在实际分析中,还可以根据遗传相似度进行过滤,去除亲缘关系较近的样本,上述代码为了方便演示,没有做过滤。
在进行REML分析时,有两种选择,第一种是默认用法,没有协变量的校正,第二种添加了协变量的校正,注意这里的协变量都是属于固定效应协变量,协变量可以是离散型,也可以是连续型。
上述代码中使用了两个协变量,第一个为covar参数指定的离散型协变量,第二个为qcovar参数指定的连续型协变量,样本的PCA分析的前10主成分作为协变量,即使用群体分层这个因素作为协变量。
输出结果后缀为hsq, 内容示意如下

Variance表示方差,SE表示标准误,其中V(G)/Vp即为SNP遗传力,下面几行为LRT检验,即似然比检验的结果,其中LRT = 2(logL - logL0), df表示自由度,Pval表示检验的p值,n表示样本个数。
·end·
—如果喜欢,快分享给你的朋友们吧—
往期精彩
GWAS meta分析
基因型填充
CNV分析
 使用GCTA进行SNP遗传力GREML评估
使用GCTA进行SNP遗传力GREML评估





 本文介绍了使用GCTA软件通过GREML方法评估SNP遗传力的流程。在GCTA中,遗传力评估基于线性混合模型,通过对样本遗传相似度矩阵(GRM)进行分析,利用REML算法估算模型参数,从而计算出SNP遗传力。文中还提到了如何构建GRM矩阵,以及在实际分析中可能涉及的样本过滤和协变量校正。
本文介绍了使用GCTA软件通过GREML方法评估SNP遗传力的流程。在GCTA中,遗传力评估基于线性混合模型,通过对样本遗传相似度矩阵(GRM)进行分析,利用REML算法估算模型参数,从而计算出SNP遗传力。文中还提到了如何构建GRM矩阵,以及在实际分析中可能涉及的样本过滤和协变量校正。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 5370
5370

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








