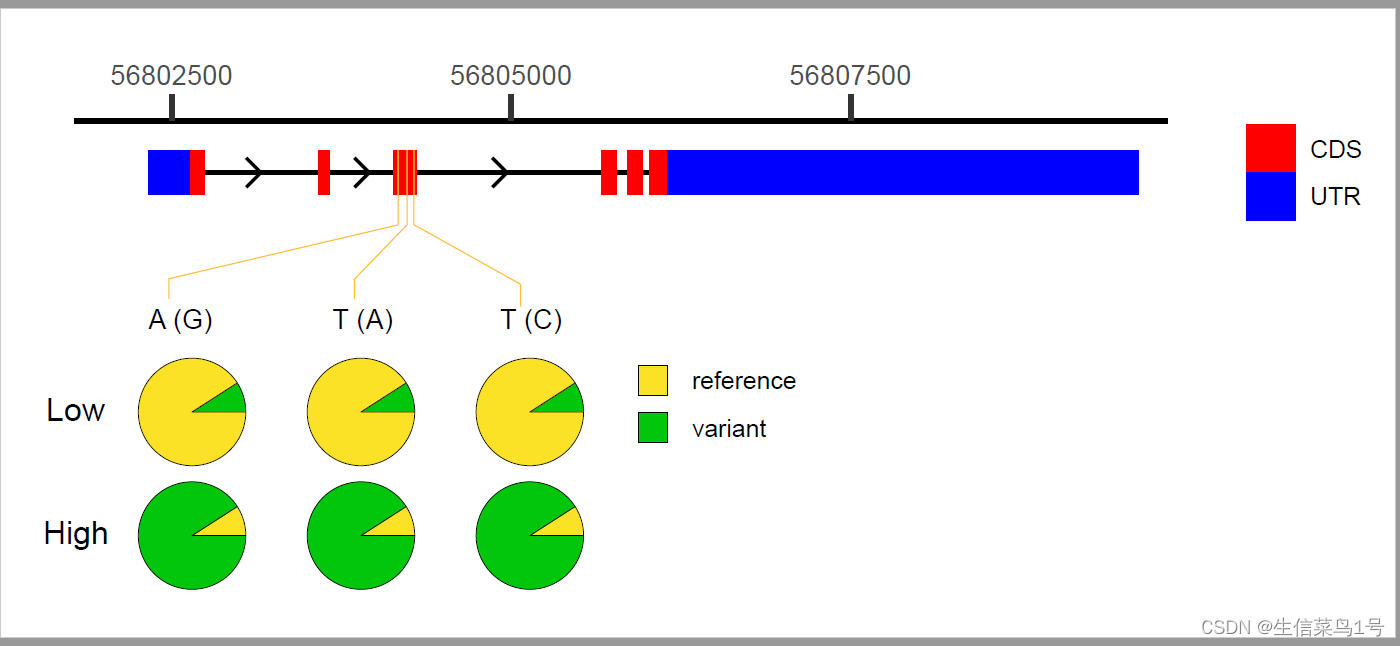
在平时研究中我们找到某个基因,需要把这个基因结构可视化出来,ggbio就可以进行基因结构图的绘制。
在绘制之前我们需要得到基因的具体结构信息,所以我们需要下载对应物种对应版本的基因组gtf文件。NCBI,UCSC,Gencode等数据库都能下载。具体文件格式如下,包含基因各个结构的具体位置。

rm(list=ls())
options(stringsAsFactors = F)
#安装包
#BiocManager::install('ggbio')
#调用
library(ggh4x)
library(ggplot2)
library(ggbio)
library(GenomicRanges)
library(readxl)
data=read_xlsx('chr4.xlsx')
我们需要自己把感兴趣的基因的CDS和UTF给取出来。也就是gtf文件的1,3,4,5列,当然有的基因因为选择剪切的缘故可能有多个转录本,我们自行选择想要的转录本的结构就好。注意各个结构范围不要有重叠的地方,多个结构要对应一个转录本。

#构建GRanges对象,包括染色体,start,end这三个必要的选项,以及用于区分基因的group列,用于区分基因结构的region列。
data1<-GRanges("chr4",IRanges(start =data$Start,end=data$End,group=data$Gene,region=data$Region))
pdf(file = "1.pdf",width = 10,height = 4)
autoplot(data1,aes(group=group,fill=region,color=region),
geom="alignment")+
theme_bw()+
#x轴范围,根据基因的范围自行确定
scale_x_continuous(limits = c(56700000,56880000),
breaks = c(seq(56700000,56880000,by=60000)),
position = "top",expand = c(0,0))+
theme(panel.border = element_blank(),
panel.grid = element_blank(),
axis.line.x = element_line(),
axis.ticks.length = unit(0.2,'cm'))+
#颜色的选择
scale_fill_manual(values = c('red','blue'))+
scale_color_manual(values = c('red','blue'))+
theme(aspect.ratio = 0.1)+
scale_y_continuous(limits = c(0,length(table(data$Gene))+1))+
theme(axis.text.y = element_blank(),
axis.ticks.y = element_blank())+
coord_cartesian(clip="off")
dev.off()

得到的图片直接在AI中美化就好。























 810
810

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








