Hifiasm 使用教程
介绍
Hifiasm 是一个快速的单倍型解析 de novo 组装软件,最初设计用于 PacBio HiFi 读取。其最新版本可以通过利用超长的 Oxford Nanopore 读取支持端粒到端粒的组装。Hifiasm 可以生成单样本端粒到端粒的组装,结合了 HiFi、超长和 Hi-C 读取,可以说是最好的组装软件之一。对于 trio-binning 组装来说,它是最好的单倍型解析组装软件之一,适用于父本短读取。对于人类基因组来说,hifiasm 可以在一天内完成端粒到端粒的组装。
安装
# 安装 hifiasm (需要有g++和zlib)
git clone https://github.com/chhylp123/hifiasm
cd hifiasm && make
# 用conda安装
conda install -c bioconda hifiasm
# 直接使用集群module
module load hifiasm/0.19.9
# 用测试数据运行 (用-f0来处理较小的数据集)
wget https://github.com/chhylp123/hifiasm/releases/download/v0.7/chr11-2M.fa.gz
./hifiasm -o test -t4 -f0 chr11-2M.fa.gz 2> test.log
awk '/^S/{print ">"$2;print $3}' test.bp.p_ctg.gfa > test.p_ctg.fa # 在FASTA获得主要的contigs
软件参数
$ hifiasm
Usage: hifiasm [options] <in_1.fq> <in_2.fq> <...>
Options:
Input/Output:
-o STR prefix of output files [hifiasm.asm]
-i ignore saved read correction and overlaps
-t INT number of threads [1]
-z INT length of adapters that should be removed [0]
--version show version number
Overlap/Error correction:
-k INT k-mer length (must be <64) [51]
-w INT minimizer window size [51]
-f INT number of bits for bloom filter; 0 to disable [37]
-D FLOAT drop k-mers occurring >FLOAT*coverage times [5.0]
-N INT consider up to max(-D*coverage,-N) overlaps for each oriented read [100]
-r INT round of correction [3]
Assembly:
-a INT round of assembly cleaning [4]
-m INT pop bubbles of <INT in size in contig graphs [10000000]
-p INT pop bubbles of <INT in size in unitig graphs [0]
-n INT remove tip unitigs composed of <=INT reads [3]
-x FLOAT max overlap drop ratio [0.8]
-y FLOAT min overlap drop ratio [0.2]
-u disable post join contigs step which may improve N50
--lowQ INT
output contig regions with >=INT% inconsistency in BED format; 0 to disable [70]
--b-cov INT
break contigs at positions with <INT-fold coverage; work with '--m-rate'; 0 to disable [0]
--h-cov INT
break contigs at positions with >INT-fold coverage; work with '--m-rate'; -1 to disable [-1]
--m-rate FLOAT
break contigs at positions with <=FLOAT*coverage exact overlaps;
only work with '--b-cov' or '--h-cov'[0.75]
--primary output a primary assembly and an alternate assembly
Trio-partition:
-1 FILE hap1/paternal k-mer dump generated by "yak count" []
-2 FILE hap2/maternal k-mer dump generated by "yak count" []
-c INT lower bound of the binned k-mer's frequency [2]
-d INT upper bound of the binned k-mer's frequency [5]
-3 FILE list of hap1/paternal read names []
-4 FILE list of hap2/maternal read names []
--t-occ INT
force remove unitigs with >INT unexpected haplotype-specific reads;
ignore graph topology; [60]
Purge-dups:
-l INT purge level. 0: no purging; 1: light; 2/3: aggressive [0 for trio; 3 for unzip]
-s FLOAT similarity threshold for duplicate haplotigs [0.75 for -l1/-l2, 0.55 for -l3]
-O INT min number of overlapped reads for duplicate haplotigs [1]
--purge-cov INT
coverage upper bound of Purge-dups [auto]
--n-hap INT
number of haplotypes [2]
Hi-C-partition:
--h1 FILEs file names of Hi-C R1 [r1_1.fq,r1_2.fq,...]
--h2 FILEs file names of Hi-C R2 [r2_1.fq,r2_2.fq,...]
--seed INT RNG seed [11]
--n-weight INT
rounds of reweighting Hi-C links [3]
--n-perturb INT
rounds of perturbation [10000]
--f-perturb FLOAT
fraction to flip for perturbation [0.1]
格式转换
# bam --> fasta
samtools view *.bam | awk '{print ">"$1"\n"$10}' > fasta
#补充一下其他格式的转换
## sam ---> fasta
cat *.sam | awk '{print ">"$1"\n"$10}' > *.fasta
## fasta ---> sam
bowtie2 -1 *_1.fa -2 *_2.fa -p 16 -x prefix -S *.sam
## sam --> bam
# -@:线程 -b:输出格式为BAM -S:自动检测输入格式 -o:输出文件
samtools view -@ 16 -b -S final.sam -o final.bam
## bam --> sam
samtools view *.bam -O SAM > *.sam
用途
# 组装近交/同源基因组(-l0 禁用重复清除功能)
hifiasm -o CHM13.asm -t 32 -l0 CHM13-HiFi.fa.gz 2> CHM13.asm.log
# 利用内置的重复清除功能组装杂合基因组
hifiasm -o HG002.asm -t 32 HG002-file1.fq.gz HG002-file2.fq.gz
# 用两个FASTQ文件中的成对端短reads进行 Hi-C 相位分析
hifiasm -o HG002.asm --h1 read1.fq.gz --h2 read2.fq.gz HG002-HiFi.fq.gz
# 双亲二代测序组件 (需要文件 https://github.com/lh3/yak)
yak count -b37 -t16 -o pat.yak <(cat pat_1.fq.gz pat_2.fq.gz) <(cat pat_1.fq.gz pat_2.fq.gz)
yak count -b37 -t16 -o mat.yak <(cat mat_1.fq.gz mat_2.fq.gz) <(cat mat_1.fq.gz mat_2.fq.gz)
hifiasm -o HG002.asm -t 32 -1 pat.yak -2 mat.yak HG002-HiFi.fa.gz
# 使用 HiFi、超长(ont)和 Hi-C reads进行端粒到端粒的单倍体组装
hifiasm -o HG002.asm --h1 read1.fq.gz --h2 read2.fq.gz --ul ul.fq.gz HG002-HiFi.fq.gz
注意事项
- no need polish
- 无需合并多个输入文件
- 绝大多数二倍体基因组,只需要组装2n中的n,所以参数一般给 -l 2 -n 4
只有Hifi数据
- 无需额外的数据类型组装 HiFi reads
hifiasm -o NA12878.asm -t 32 NA12878.fq.gz#其中NA12878.fq.gz提供输入reads,-t设置使用中的CPU数,-o输出文件的前缀名
# no need haplotype
hifiasm --primary -o NA12878.asm -t 32 NA12878.fq.gz
# -l:0:没有对组装去冗余,组装结果包括全部组装出来的contig,可能包含多个单倍体基因组;2/3:会对组装出来的基因组进行去冗余,对于二倍体,得到的结果基本上是全基因组一半的大小
# -n: 一般给3或者4,默认3,表示组装的contig中,unitigs支持大于3或4才保留,该参数会将支持度比较低的contig去掉
使用ONT数据
- Hifiasm 可以集成超长 ONT 读取来生成端粒到端粒的组装:
# only ONT
hifiasm -o NA12878.asm -t32 --ul ul.fq.gz HiFi-reads.fq.gz
# + Hi-C
hifiasm -o NA12878.asm -t32 --ul ul.fq.gz --h1 read1.fq.gz --h2 read2.fq.gz HiFi-reads.fq.gz
# + parental
hifiasm -o NA12878.asm -t32 --ul ul.fq.gz -1 pat.yak -2 mat.yak HiFi-reads.fq.gz
使用trio binning组件
- 当有父本的短读取可用时,hifiasm 还可以通过 trio binning 生成一对单倍型解析的组装。要进行这样的组装,您首先需要使用 yak 对 k-mer 进行计数,然后再进行组装。
yak count -k31 -b37 -t16 -o pat.yak paternal.fq.gz
yak count -k31 -b37 -t16 -o mat.yak maternal.fq.gz
hifiasm -o NA12878.asm -t 32 -1 pat.yak -2 mat.yak NA12878.fq.gz
也可以通过merqury来确定 k-mer的大小
使用HI-C数据
- 利用成对的端到端 Hi-C reads 生成一对单倍型解析的组装。
hifiasm -o NA12878.asm -t 32 --h1 read1.fq.gz --h2 read2.fq.gz HiFi-reads.fq.gz
# --h1 --h2 HiC 数据
# -t 线程数
# -o 输出文件前缀
结果
hifiasm 输出结果有哪些?
一般来说,用hifiasm组装基因组,纯合材料用- l0,非纯系材料,参数-l3,分出两个单倍型,是默认参数,所以默认可以不设置。
两个模式大体输出结果如下图:
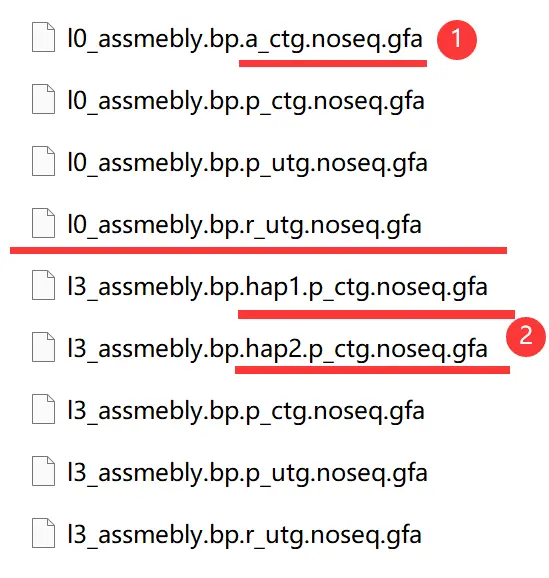
可以看出来,区别在于前者多输出了一个a_ctg而后者则多输出了hap1.p_ctg和hap2.p_ctg
逻辑上,看过文献应该比较容易理解

理解共同的输出文件
r_utg
r 代表 raw,也就是最初组装出来的原始结果。其中 utg 表示 unitig,或理解为初步组装且没有拆分气泡或者冲突的结果。

p_utg
p 代表 primary,基本上是在 raw 的基础上去除掉一些覆盖率低的连接(或叫气泡)。看起来简洁了不少,其实是少了 60000 条边(当然图太大,看不太出区别…不过确实是小了四分之一)

或许高杂合材料里面,覆盖率低的区域,也可能是另一个单倍型区域?用于后续HiC挂载,可能也要考虑进去。在 p_utg 和 p_ctg 上的选择,或需要考量。
p_ctg
p 代表 primary,ctg 代表了拆分结果。

逻辑上 p_ctg 包含了全部单倍型结果(含 hap1 和 hap2)。事实上,这个文件在l0和l3的表现不相同,可以从文件大小看出区别。个人感觉,l0下 p_ctg 约等于 canu 软件的组装结果;而l3模式下,p_ctg 比较接近于主要的一套单倍型结果,大体是hap1和hap2中表现最好的每个contig的hap的组合。
a_ctg
a 代表 alternative,大体是拆分出来 p_ctg 之后剩下的就放在 alternative。
hap1/hap2 ctg
亦即两个单倍型的拆分结果。
假如有 HiC 数据
结果类似。phased的效果会好很多。
对于三重组装,hifiasm会生成以下文件:
prefix.r_utg.gfa(Haplotype-resolved raw unitig graph in GFA format):保存了所有的单倍型信息。
prefix.hap1.p_ctg.gfa(Phased paternal/haplotype1 contig graph):保留了阶段性父系/单倍型1组装。
prefix.hap2.p_ctg.gfa(Phased maternal/haplotype2 contig graph):保留了阶段性母系/单倍型2组装。
- 获取组装结果
# get fasta
awk '/^S/{print ">"$2;print $3}' test.p_ctg.gfa > test.p_ctg.fa
##参考
1.Cheng, H., Concepcion, G.T., Feng, X., Zhang, H., Li H. (2021) Haplotype-resolved de novo assembly using phased assembly graphs with hifiasm. Nat Methods, 18:170-175. https://doi.org/10.1038/s41592-020-01056-5
2.Cheng, H., Jarvis, E.D., Fedrigo, O., Koepfli, K.P., Urban, L., Gemmell, N.J., Li, H. (2022) Haplotype-resolved assembly of diploid genomes without parental data. Nature Biotechnology, 40:1332–1335. https://doi.org/10.1038/s41587-022-01261-x
3.使用hifiasm组装hifi基因组的方法介绍 作者:生信技术 http://t.csdnimg.cn/UBXEm
4.基因组装 | hifiasm 输出结果文件细究 作者:生信石头基因组装 | hifiasm 输出结果文件细究 - 简书 (jianshu.com)
5.单倍体组装工具Hifiasm简介及基本运行命令(一) 作者:吕强强学生信单倍体组装工具Hifiasm简介及基本运行命令(一) - 简书 (jianshu.com)
6.基因组组装:Hifiasm 使用教程 作者:科学冷冻工厂 基因组组装:Hifiasm 使用教程-腾讯云开发者社区-腾讯云 (tencent.com)
























 6384
6384

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








