一、写在前面
总有小伙伴购买服务器(足够支持你完成硕博生涯的生信环境)时不知道哪个版本的配置适合自己,这里我们对CellChat细胞通讯运行进行了内存和时间占用的压力测试。往期测试可见:服务器压力测试
我们最多测试了30w个细胞,占用内存峰值接近60GB,显然比monocle占据的内存小的多,考虑到系统本身以及环境中的其它变量,推荐大家至少准备128GB内存为宜。若采取并行计算等操作,占据的内存数量会更多!
本教程基于Linux中的Rstudio环境(足够支持你完成硕博生涯的生信环境)演示,计算资源不足的同学可参考:
生信分析为什么要使用服务器?
足够支持你完成硕博生涯的生信环境
配置一个心仪的工作站(硬件+环境配置)
独享服务器,生信分析不求人
二、准备工作
数据来源于GEO数据库中的GSE131907和GSE222318两个数据集。
此次教程整合了前面两个数据集,两个数据集整合后将矩阵重新划分为五个矩阵,分别对应了30万、20万、10万、5万和2.5万细胞数。
前期数据处理包括了数据整合、数据的下采样,具体的代码运行过程可以参考30w单细胞数据会吃掉多少内存?
因此本流程接下来可以直接读取之前保存好的rds文件,进行Cellchat的时间和内存测试。
suppressMessages({
library(Seurat)
library(CellChat)
library(peakRAM)
library(patchwork)
})
obj_30w <- readRDS("../11_monocle//obj_30w.rds")
obj_2.5w <- subset(obj_30w,downsample=370)
obj_5w <- subset(obj_30w,downsample=770)
obj_10w <- subset(obj_30w,downsample=1900)
obj_20w <- subset(obj_30w,downsample=5000)三、时间和内存测试
Cellchat是一款常用于单细胞数据细胞通讯分析的软件,对于软件不熟悉的同学可以从《细胞通讯》CellChat学习手册 系统学起。《单细胞转录组测序数据分析》——《细胞通讯》视频学习教程在B站同步更新,涵盖细胞通讯绪论以及cellchat分析代码实操。接下来我们就进入真正的时间和内存测试环节。
for (obj in c(obj_2.5w,obj_5w,obj_10w,obj_20w,obj_30w)){
print(obj)
print(
#用于统计内存和时间的函数
peakRAM({
#提取表达矩阵
data.input <- GetAssayData(obj, layer = "data")
#提取meta.data
meta <- obj@meta.data
meta$seurat_clusters <- paste0("g",meta$seurat_clusters)
#构建cellchat对象
cellchat <- createCellChat(object = data.input, meta = meta,group.by = "seurat_clusters")
#如果上述没有指定meta=meta,可以如下手动加入
cellchat <- addMeta(cellchat, meta = meta)
cellchat <- setIdent(cellchat, ident.use = "seurat_clusters")
levels(cellchat@idents)
groupSize <- as.numeric(table(cellchat@idents))
#使用人类数据库,如果分析物种是小鼠可以设置为CellChatDB.mouse
CellChatDB <- CellChatDB.human
#取出相应分类用作分析数据库
CellChatDB.use <- subsetDB(CellChatDB, search = "Secreted Signaling")
#将数据库内容载入cellchat对象中
cellchat@DB <- CellChatDB.use
#表达量预处理,取出表达数据
cellchat <- subsetData(cellchat,features = NULL)
#寻找高表达的基因
cellchat <- identifyOverExpressedGenes(cellchat)#
#寻找高表达的通路
cellchat <- identifyOverExpressedInteractions(cellchat)
#注意projectData这个函数已经不能用了,投影到PPI请修改成下面函数,参考https://github.com/jinworks/CellChat/issues/185
# cellchat <- projectData(cellchat, PPI.human)
cellchat <- smoothData(cellchat, adj = PPI.human)
#默认计算方式为type = "truncatedMean",
#默认cutoff的值为20%,即表达比例在25%以下的基因会被认为是0, trim = 0.1可以调整比例阈值,较耗时,2.5w细胞约细胞约10min
cellchat <- computeCommunProb(cellchat, raw.use = T)
#去掉通讯数量很少的细胞
cellchat <- filterCommunication(cellchat, min.cells = 10)
cellchat <- computeCommunProbPathway(cellchat) #每对配受体的预测结果存在net中,每条通路的预测结果存在netp中
cellchat <- aggregateNet(cellchat)
})
)
print(cellchat)
}
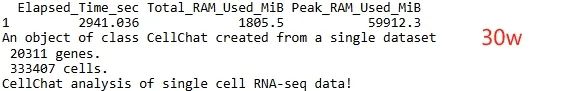
四、总结
sum <- data.frame(matrix(NA, nrow = 5, ncol = 2))
colnames(sum) <- c("time","peak_mem")
row.names(sum) <- c("2.5w","5w","10w","20w","30w")
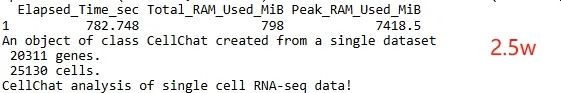
sum["2.5w",] <- c(13,7418)
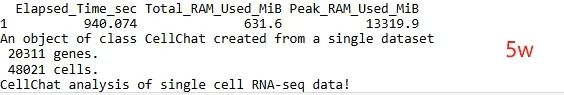
sum["5w",] <- c(16,13319)
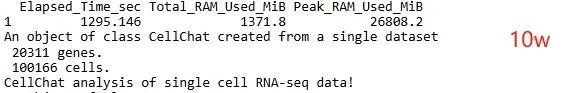
sum["10w",] <- c(22,26808)
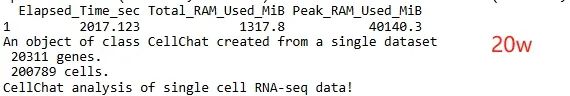
sum["20w",] <- c(34,40140)
sum["30w",] <- c(49,59912)
sum$Category <- rownames(sum)
sum$Category <- factor(sum$Category, levels=sum$Category)
ggplot(sum, aes(x = Category , y = time, fill= Category)) + geom_bar(stat = "identity")+
labs(x = "Category", y = "Time(min)", title = "Cellchat Time Compare") +
theme_minimal() +
theme(axis.text.x = element_text(angle = 45, hjust = 1, size = 12),
axis.text.y = element_text(size = 12),
axis.title = element_text(size = 14, face = "bold"),
plot.title = element_text(size = 16, face = "bold", hjust = 0.5),
legend.title = element_text(size = 12, face = "bold"),
legend.text = element_text(size = 10)) 
从细胞数和计算时间统计结果的柱形图可以看出,随着细胞数目的增加,计算时间呈现逐渐递增的趋势。
ggplot(sum, aes(x = Category, y = peak_mem, fill= Category)) + geom_bar(stat = "identity")+labs(x = "Category", y = "peak_mem(MB)", title = "Cellchat Peak Mem Compare") +theme_minimal() +theme(axis.text.x = element_text(angle = 45, hjust = 1, size = 12),axis.text.y = element_text(size = 12),axis.title = element_text(size = 14, face = "bold"),plot.title = element_text(size = 16, face = "bold", hjust = 0.5),legend.title = element_text(size = 12, face = "bold"),legend.text = element_text(size = 10)

从细胞数和峰值内存的统计结果的柱形图可以看出,随着细胞数目的增加,峰值内存也呈现逐渐递增的趋势。这个和细胞数与时间统计柱形图对比来看,二者趋势是一致的。对于后续想要自己进行cellchat分析的同学可以参考这里的时间和内存消耗。如果手头数据集细胞数目比较多的情况,还是推荐使用服务器进行分析,避免超过电脑负荷生信分析为什么要使用服务器?。
bj_20w <- subset(obj_30w,downsample=5000)























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








