零基础入门转录组分析——数据处理(自测序数据)
通常有的小伙伴会选择将自己的样本送到不同的测序公司进行测序,会出现以下几种情况:
- 有的公司返回来的数据文件中是已经注释好的count(行为基因symbol,列为样本名),这种情况的可以直接用于后续的分析。
- 但是有的公司返回来的数据行是基因ID,列为样本,这样的数据就需要自己将基因ID转换成symbol。
- 再甚至有些小伙伴的导师会觉得公司定量结果不可靠,会要求重新跑一遍质控,定量等流程(重新定量可参考之前的相关教程零基础入门转录组分析——第五章(表达定量))
本章教程会重点关注第二种和第三种情况,介绍如何将自测序的基因ID转换成对应基因symbol,用于后续分析
本项目以之前教程中定量结果作为展示
物种:C57BL/6J小鼠
实验分组:药物处理组,对照组,每组6只鼠
R版本:4.2.2
要用到的R包:tidyverse
1. 原始数据集
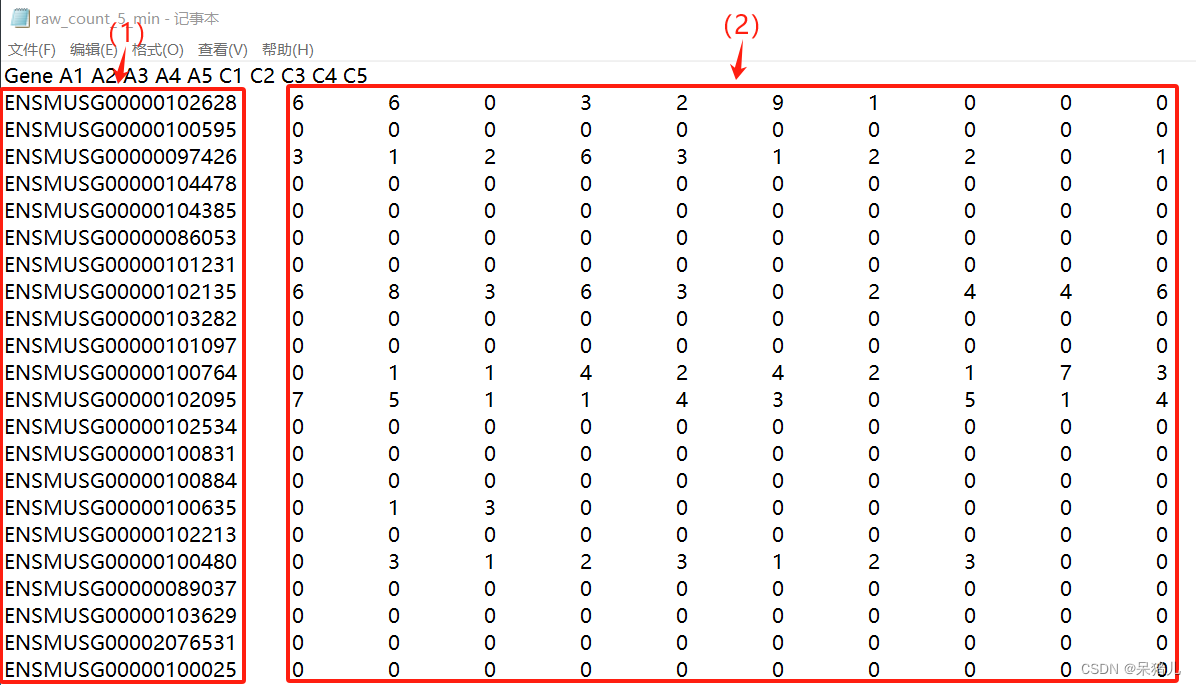
首先来看一下之前定量后的原始count长啥样零基础入门转录组分析——第五章(表达定量),如下图所示:(每一行是一个基因ID,每一列是一个样本)
(1)基因的ensemble_ID
(2)是基因所对应的样本原始count
2. 数据处理(Rstudio)
首先将这个原始的表达矩阵导入到Rstudio中,并设置工作路径
rm(list = ls()) # 删除工作空间中所有的对象
setwd('/XX/XX/XX') # 设置工作路径
if(!dir.exists('./00_rawdata')){
dir.create('./00_rawdata')
} # 判断该工作路径下是否存在名为00_rawdata的文件夹,如果不存在则创建,如果存在则pass
setwd('./00_rawdata/') # 设置路径到刚才新建的00_rawdata下
导入上面的那个表达矩阵表,并设置第一列列名为ID
train_data <- read.table('./raw_count_5_min', quote="\"", comment.char="")
colnames(train_data) <- train_data[1, ]
train_data <- train_data[-1, ]
colnames(train_data)[1] <- 'ID'
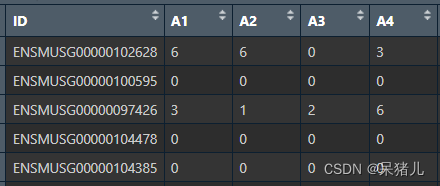
如下图所示,第一列为基因的ensemble_ID,之后每一列都是样本对应的原始count(接下来要做的就是将这个ensemble_ID转成常见的基因symbol)
接下来要导入一个比较重要的文件——gencode.vM33.annotation.gtf
从bing上搜索“小鼠gencode.vM33.annotation.gtf ”,检索到的第一个就是小鼠的基因注释文件
注:这里的注释文件和零基础入门转录组分析——数据处理(TCGA数据库)是不一样,因为物种是小鼠,TCGA那个用的人类基因注释文件
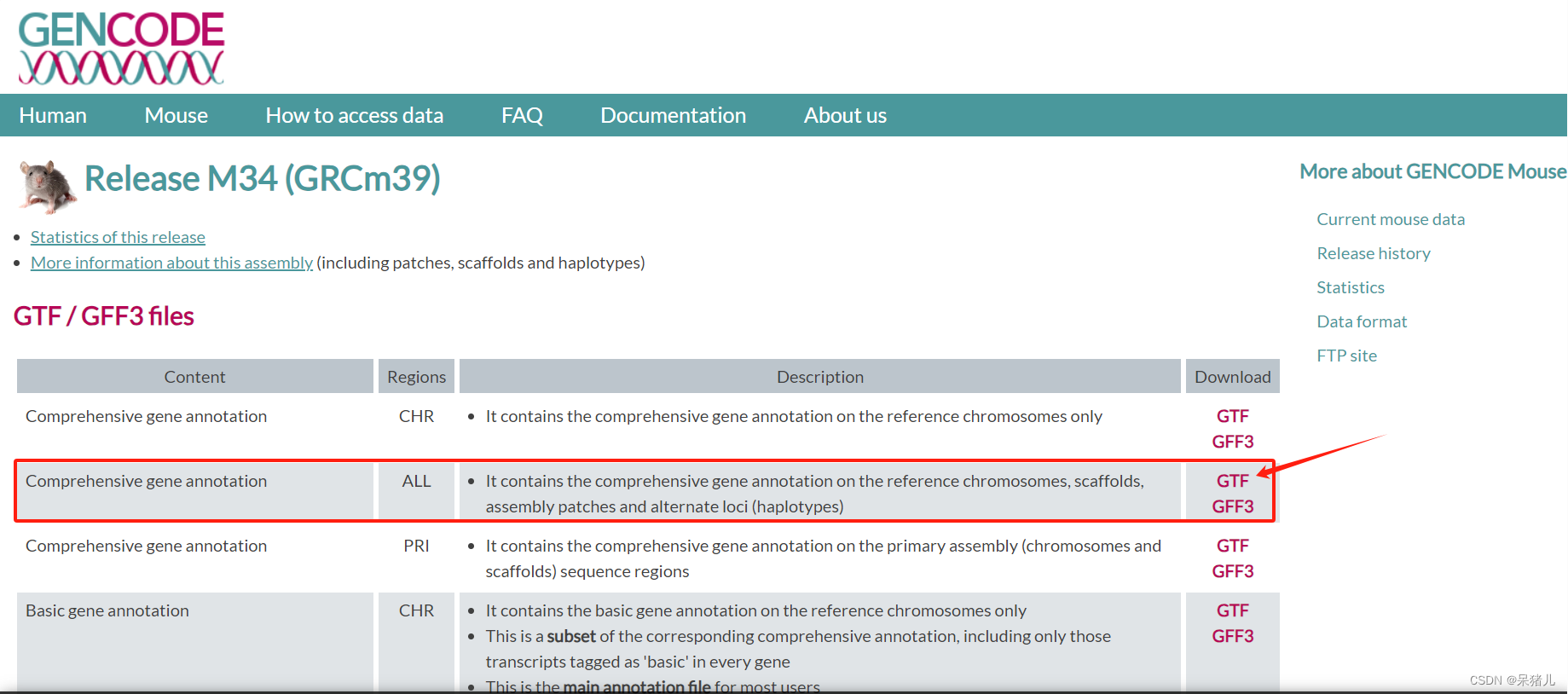
进入GENCODE的gencode.vM33.annotation页面,可以看到有很多gtf文件,关于gtf文件的相关信息,可以参考第三列Description,这里我们选择第二个(它包含了全部的基因注释信息,比较全面)。
下载后是个.gz的压缩包,解压后同样上传到Rstudio的00_rawdata中,运行如下代码导入:
gene_annotation <- import('./gencode.vM33.annotation.gtf')
gene_annotation <- as.data.frame(gene_annotation)#将文件转换为数据框格式
gene_annotation <- gene_annotation[gene_annotation$type == 'gene', ] # 筛选为gene的类型
gene_annotation <- gene_annotation[gene_annotation$gene_type == 'protein_coding', ]
gene_annotation <- dplyr::select(gene_annotation, gene_id, gene_name, seqnames, start, end, width, strand, gene_type)
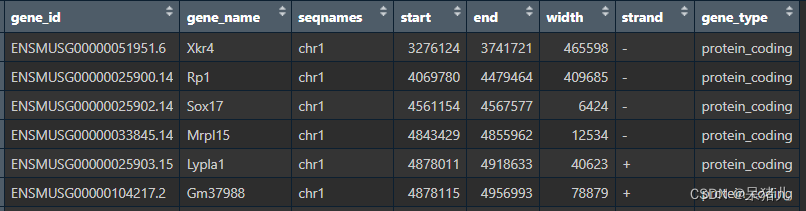
看看最后处理好的gene_annotation长啥样,点击Rstudio右上角的gene_annotation 如下图所示:
- gene_id——基因的ensemble_ID
- gene_name——基因名
- seqnames——基因位于哪条染色体
- start——该基因于染色体上的起始位置
- end——该基因于染色体上的终止位置
- width——该基因的长度(注意:凭借这个就可以计算FPKM)
- strand——链的方向,+表示正链,-表示负链(关于链的方向的描述信息可以自行查找资料,在这里跟教程无关不做过多介绍)
- gene_type——基因的类型,有的是miRNA,有的是lincRNA…(这里选择的是protein_coding,即编码蛋白)
注意一点:gene_annotation中gene_id这一列中有小数点,但是之前的表达矩阵中基因ID是没有小数点的。
因此要去除小数点后选择第一列和第二列(让表达矩阵和基因注释文件匹配)
gene_annotation <- gene_annotation %>% separate_wider_delim(gene_id, ".", names = c("ID", "var1")) %>% dplyr::select(-var1) # 以.分隔gene_id列
gene_symbol <- gene_annotation[,(1:2)] # 筛选gene_annotation文件中的第一列和第二列
colnames(gene_symbol) <- c("ID", "symbol") # 将gene_symbol文件的列名改成ID和symbol
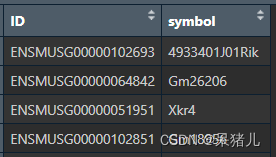
gene_symbol如下图所示,第一列是ensemble_ID,第二列是基因symbol
准备好前面的count数据和基因注释文件后,运行下面代码,每句代码后都有注释信息,可以挨个运行,比对处理前后的变化
data_count <- train_data
data_count$ID <- as.character(data_count$ID) # 将data_count文件中ID列从数值类型数据转化为字符串类型数据
gene_symbol$ID <- as.character(gene_symbol$ID) # 将gene_symbol文件中ID列从数值类型数据转化为字符串类型数据
data_count <- inner_join(data_count, gene_symbol, by = "ID") # 将data_count文件和gene_symbol文件根据ID列进行合并(这样就能获得ID对应的基因名,但是都排在最后)
data_count <- dplyr::select(data_count, -ID) # 删除ID列
data_count <- dplyr::select(data_count, symbol, everything()) # 重新排列,将最后一列的基因名放到第一列(如果不加everything只会选择symbol一列)
data_count <- mutate(data_count, rowMean=rowMeans(data_count[grep('count',names(data_count))])) # 添加一列为表达量的平均值
data_count <- arrange(data_count, desc(rowMean)) # 把表达量的平均值从大到小排序
data_count <- distinct(data_count, symbol, .keep_all = T) # distinct函数被用于去重,.keep_all参数表示去重后返回数据框的所有列向量
data_count <- dplyr::select(data_count, -rowMean) # 去除rowMean这一列
data_count <- tibble::column_to_rownames(data_count, var = "symbol") ## 把第一列变成行名并删除
dim(data_count)
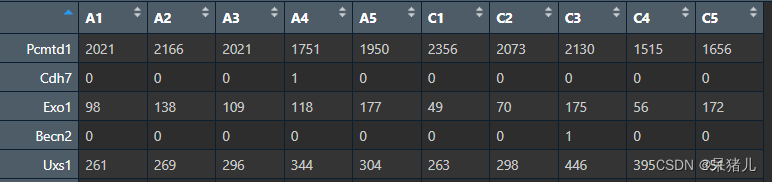
上面的代码说白了就是一个去重加基因注释,结果如下图所示,行为基因symbol,列为样本名,表格中的整数就是原始的count。
将处理好的count表达矩阵保存即可:
write.csv(data_count, file = paste0('dat.count.csv'))
到这里原始count表达矩阵就已经处理好了,分组情况是按照自己的样本进行分组的,分组信息表可参考零基础入门转录组分析——数据处理(TCGA数据库)
接下来就是对raw_count做标准化处理,将其转换成FPKM形式。
3. 数据标准化(Rstudio)
关于FPKM的解释,可以参考RPKM、FPKM 和 TPM,解释清楚这篇文章(简单说就是由于存在测序深度不同的情况,所以原始count并不能真实反映基因表达量,有的基因长度较长,匹配到的read会非常多,因此会根据基因长度做个校正)
首先要从前面gene_annotation文件中获取基因的长度
## FPKM
colnames(gene_annotation) # 查看gene_annotation列名
gene_length <- dplyr::select(gene_annotation, gene_name, width) # 选择gene_name和width两列
colnames(gene_length) <- c('symbol', 'length') # 列名重新定义为symbol和length
gene_length <- gene_length[gene_length$symbol %in% rownames(data_count), ] # 将gene_length的基因symbol与count表达矩阵中的基因symbol匹配
gene_length <- gene_length[!duplicated(gene_length$symbol), ] # 去重
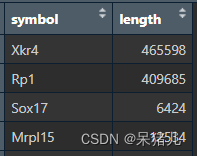
gene_length 如下图所示:第一列为基因symbol,第二列为基因长度
定义一个名为countToFpkm 的计算函数:
countToFpkm <- function(counts, effLen){
N <- sum(counts)
exp( log(counts) + log(1e9) - log(effLen) - log(N) )
}
通过apply函数和之前自定义的countToFpkm 函数对表达矩阵(dat)进行FPKM转换
dat <- data_count[rownames(data_count) %in% gene_length$symbol, ]
dat <- as.data.frame(lapply(dat, as.numeric))
rownames(dat) <- rownames(data_count)
expr_fpkm <- apply(dat, 2, countToFpkm, effLen = gene_length$length)
expr_fpkm就是经过FPKM转换的数据集(但是目前还不能直接用,是因为除了差异分析之外,基本所有的分析点用到的数据集都是log2()处理的,这是因为纯fpkm值有的会很大,不同分析点在计算的时候可能会由于个别值特别大会导致较大的误差,因此要通过log2处理。)
接下来通过GEO官方的判定公式,如果logC的值为TRUE则对expr_fpkm数据集进行log2处理,如果为FALSE则不处理
qx <- as.numeric(quantile(expr_fpkm, c(0., 0.25, 0.5, 0.75, 0.99, 1.0), na.rm=T))
LogC <- (qx[5] > 100) ||
(qx[6]-qx[1] > 50 && qx[2] > 0) ||
(qx[2] > 0 && qx[2] < 1 && qx[4] > 1 && qx[4] < 2)
if (LogC) {
expr_fpkm[which(expr_fpkm <= 0)] <- NaN
expr_fpkm <- log2(expr_fpkm)
print("log2 transform finished")
}else{
print("log2 transform not needed")
}
注:这里需要特别注意一点的是这个判定函数并不是万能的,有些数据集在这一步判定的时候为FALSE,但是里面的fpkm值都很大,像这种特殊情况的就不能用判定了,可以直接对其做log2处理
最后将处理好的数据集保存即可
expr_fpkm <- data.frame(expr_fpkm)
write.csv(expr_fpkm, file = paste0('dat.fpkm.csv'))
补充内容:
- 为什么要进行log2处理:一般数据集的表达值范围都是非常大的,从0到25000不等。将这些数据绘制密度分布后,一般呈现右偏,即大部分信号都是在左侧,右侧拖个长长的尾巴(偏态分布),不利于研究,而经过log2转化后,数据更加集中,更加接近正态分布,更方便套用正态分布那一套理论进行分析。
以上就是自测序数据处理的所有过程,如果有什么需要补充或不懂的地方,大家可以私聊我或者在下方评论。
如果觉得本教程对你有所帮助,点赞关注不迷路!!!
- 目录部分跳转链接:零基础入门生信数据分析——导读
























 5万+
5万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










