本文介绍利用 rdkit 处理多肽链的末端修饰,主要是需要根据文字描述的 N 端及 C 端修饰利用 ReplaceSubstructs 自动处理,难点在于子结构搜索。
首先读入多肽链,下面是示例:
smi='CC[C@H](C)[C@H](NC(=O)[C@H](CO)NC(=O)CN)C(=O)NCC(=O)N[C@@H](C)C(=O)N[C@@H](C)C(=O)N[C@@H](CO)C(=O)N[C@@H](CCSC)C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](Cc1ccccc1)C(=O)O'
mol=Chem.MolFromSmiles(smi)
以序列描述也可以,同时可以将序列转化成 SMILES:
def transform(seq):
if (pd.isnull(seq)):
return None
else:
try:
mol = Chem.MolFromSequence(seq)
smi = Chem.MolToSmiles(mol)
return smi
except:
print("转换失败",seq)
return '###{}'.format(seq)
将 seq 存储在 dataframe 中,利用下面的方法即可自动转换:
df['SMILES']=df['SEQUENCE'].map(transform)
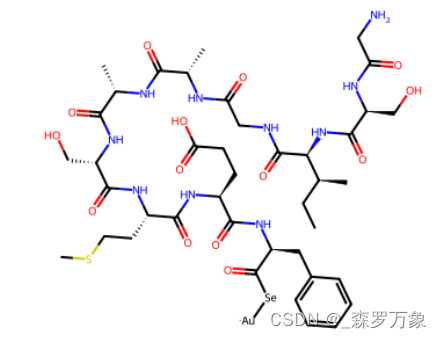
假设 C 端修饰是将羧基的OH替换为 Au 及 Se (当然没有这种修饰,这里只是假设):


自动替换子结构需要先精准定位到肽链的 C 末端,再进行子结构替换:
patt=Chem.MolFromSmarts('[$([OH]C(=O)CN)]') #这里是关键
rep=Chem.MolFromSmiles('[Se][Au]')
Chem.MolToSmiles(AllChem.ReplaceSubstructs(mol,patt,rep)[0],canonical=True)
运行上面代码即可得到修饰之后的多肽链 SMILES
同理 N 端修饰类似处理,以 NH2 做以下修饰为例:

patt1=Chem.MolFromSmarts('[$([NH2]CC(=O))]') #N末端未成环
patt2=Chem.MolFromSmarts('C1CCCN1') #N末端成环
rep1=Chem.MolFromSmiles('[NH]C(=O)(CCCCCCCCCCNC(=O)COc1ccc(Cl)cc1Cl)')
mol=AllChem.ReplaceSubstructs(Chem.MolFromSmiles(seqList[i]),patt1,rep1)
Chem.MolToSmiles(mol[0],canonical=True)
运行上面代码即可得到多肽 N 端做如上修饰后的 SMILES








 本文介绍如何使用RDKit处理多肽链的N端和C端修饰,包括通过文字描述自动处理末端修饰的方法。重点讲解了如何精确定位肽链末端并进行子结构替换。
本文介绍如何使用RDKit处理多肽链的N端和C端修饰,包括通过文字描述自动处理末端修饰的方法。重点讲解了如何精确定位肽链末端并进行子结构替换。

















 799
799

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










