这里不直接用ggplot2画,因为要做差异分析,所以直接用ggpubr
step0.数据读取
load("Type.Rdata")
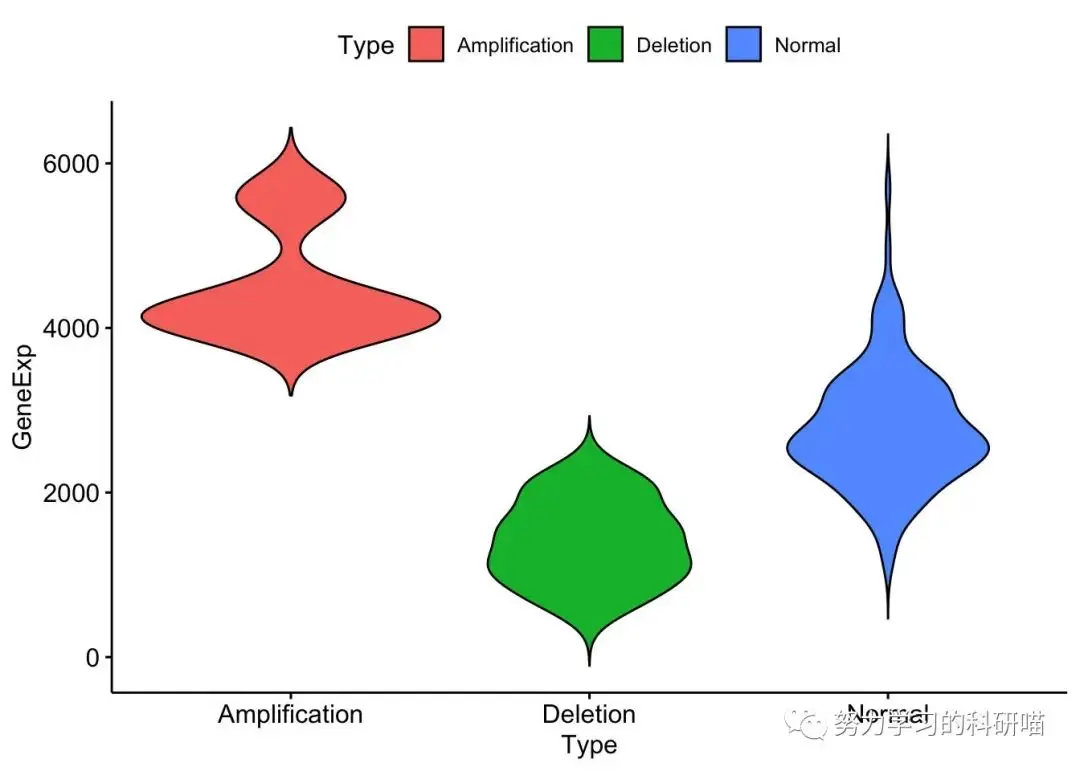
首先外面是一个小提琴图,中间一个箱式图,然后两两之间做差异分析,而且排列要从低到高,并且要用*来表示p值的显著程度
step1.映射
x轴是类型,y轴是表达量,填充的颜色按照type来区分,所以直接引用ggviolin
library(ggpubr)
ggviolin(Type, x="Type", y="GeneExp", fill = "Type")
step2.调色
柳叶刀配色
ggviolin(Type, x="Type", y="GeneExp", fill = "Type",
palette = c('lancet'))

step3.添加箱式图(可选)
需要给它打上白色,覆盖下面的小提琴图配色
ggviolin(Type, x="Type", y="GeneExp", fill = "Type",
palette = c('lancet'),
add = "boxplot",
add.params = list(fill="white"))

step4.排序(order函数)
比如要看泛肿瘤里面各种肿瘤里面特定基因的表达量之间有没差异,就可以用这个小提琴图来进行展示,这时候order就可以排序了
type "Amplification","Normal","Deletion")
ggviolin(Type, x="Type", y="GeneExp", fill = "Type",
palette = c('lancet'),
add = "boxplot",
add.params = list(fill="white"),
order=type)

step5.对比
三组两两对比 :先定义一下三组之间的关系,再用一个对比函数,stat_compare_means设置两两比较
my_comparisons=list(c("Deletion","Normal"),
c("Normal","Amplification"),
c("Amplification","Deletion"))
#my_comparisons
#[[1]]
#[1] "Deletion" "Normal"
#
#[[2]]
#[1] "Normal" "Amplification"
#
#[[3]]
#[1] "Amplification" "Deletion"
type "Amplification","Normal","Deletion")
ggviolin(Type, x="Type", y="GeneExp", fill = "Type",
palette = c('lancet'),
add = "boxplot",
add.params = list(fill="white"),
order=type)+
stat_compare_means(comparisons = my_comparisons)

基础知识,多多学习







 本文介绍了如何使用R语言中的ggpubr包进行数据可视化,包括绘制小提琴图和箱式图,对基因表达量进行类型间的差异分析,并通过柳叶刀配色和统计对比功能。步骤详细讲解了数据加载、映射、排序以及三组间对比的实现过程。
本文介绍了如何使用R语言中的ggpubr包进行数据可视化,包括绘制小提琴图和箱式图,对基因表达量进行类型间的差异分析,并通过柳叶刀配色和统计对比功能。步骤详细讲解了数据加载、映射、排序以及三组间对比的实现过程。
















 565
565

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








