《生物信息学》(即网络版备课笔记《生物信息学札记》第四版)自 2017 年出版以来,受到广大读者的喜爱,反响热烈,编者深感欣慰。华中科技大学一位同行告诉我,《生物信息学札记》当年对他影响很大,其中动态规划算法就是读了札记后才看懂的。
本书被不少院校和研究机构作为教学用书,例如,中国科学院遗传与发育生物学研究所将本书列为研究生课程的主要参考书,华中农业大学信息学院将本书作为生物信息学专业本科生的主要教材,等等。这使我倍感压力,也给了我更大的动力来修订好这本教材。生物信息学是一个快速发展的学科,技术变革很快,加之上一版中存在一些疏漏和不足(感谢许多同行和读者给我的来信),深感有些内容已惨不忍睹,亟待更新。

第二版的修订工作自 2019 年 5 月启动。在此之前,我们刚刚完成了《植物基因组学》的编写。因此我们可以合理安排《生物信息学》(第二版)和《植物基因组学》的有关内容,使它们相辅相成,便于读者阅读和学习。在第二版中,我对有关算法原理的描述过程给予了更多关注。入门教材不应写得太简略,否则卡在某处可能许多年都无法理解。经过近两年的努力,总算拿出了目前的版本。
第二版主要在以下4个方面进行了修改和完善:
1
调整了全书组织框架,全书现共 15 章、3 个附录。由于高通量测序数据已成为常态数据,是目前生物信息学分析的主体,已没有必要将高通量数据分析单独成篇进行介绍。同时,将第一版“生物信息学外延与交叉篇”中有关交叉学科整合到生物信息学学科中的部分纳入整个生物信息学框架内集中介绍更为合适;第一版“生物信息学资源与实践篇”各章内容(实验除外)在第二版中均列为附录内容。这样的调整使全书逻辑更加清楚,内容更加紧凑,便于阅读。
2
增设“生物信息学统计与算法基础”一章,强化了贝叶斯统计和机器学习在生物信息学中的应用相关内容。其中,相关统计和算法内容都结合具体生物信息学应用案例进行讲述;第一版中原有的统计和算法内容,部分整合到该章内。该章与“生物信息学计算机基础”一章构成了本书两大基础支撑。
3
为了紧跟生物信息学应用前沿领域,增设“新类型组学数据分析与利用”一章,专门介绍三维基因组、单细胞组学等新领域中的生物信息学分析方法前沿。
4
为了有利于学习学科发展历史和增强教材趣味性,第二版补充了生物信息学学科相关人物和重要历史事件介绍。每章最后都增加了一篇以代表性人物为主线的“历史与人物”趣味短文。历史有利于读者理解学科发展脉络,大师会为学生树立榜样,激励他们更好地学习相关知识。
此外,为了配合生物信息学教学需求,编者在实验室主页(http://ibi.zju.edu.cn/bioinplant/)提供了本书所有彩图的下载链接,同时提供了编者的教学课件、教学大纲和思政教学切入点等材料,供“生物信息学”课程授课教师参考。
樊龙江
2021 年 2 月 6 日
于浙江大学紫金港校区启真湖畔
致 敬 经 典
本书讲述的部分人物

编辑推荐
生物信息学是一门年轻而迅猛发展的学科,现已发展成为生命科学极具吸引力的重要前沿领域。20世纪末,我国生物信息学处于起步阶段,学习资料很少,浙江大学樊龙江教授整理了“生物信息学”课程的备课笔记,取名《生物信息学札记》,放在实验室主页上供读者参考。由于网络传播的作用,该札记成为许多生物信息学初学者的必读材料,在国内形成一定影响。《生物信息学》(第二版)正是在该札记的框架基础上,结合樊龙江教授多年的教学心得和研究成果,补充大量新材料编写而成。
本书内容全面、体系完整,既包括生物信息学统计与算法基础和计算机基础等基本知识,也涵盖当下火热的组学大数据及其分析等前沿内容,书中配有丰富的生物信息学实际应用案例,还可以扫二维码查看精美的英文彩色插图和大量知识拓展材料,并可在主编实验室主页下载本书教学课件、教学大纲和思政教学切入点等材料。
郝柏林院士、顾孝诚教授、陈润生院士等老一辈科学家,在我国生物信息学引入和普及等方面做出了突出贡献,因此,本书在每章后均安排了一篇以相关领域代表人物为主线的“历史与人物”短文,有助于弘扬经典人物精神、体现课程思政特色。
可以说,《《生物信息学》(第二版)是一本理论联系实际的教材,更是一部致敬经典、具有人文历史情怀的教科书,值得推荐!
主编简介:

樊龙江,浙江大学生物信息学研究所执行所长,IBM生物计算实验室主任,生物信息学专业和作物遗传育种专业博导,宝钢优秀教师奖获得者,浙江省“万人计划”杰出人才,教育部新世纪优秀人才。主要研究领域包括植物基因组与进化、生物信息学算法与工具、植物比较医学与健康等。承担国家自然科学基金重大研究计划项目等国家级课题10余项,在Nature Ecology & Evolution、Genome Biology、PNAS、Bioinformatics等重要刊物上发表论文100余篇。主编规划教材《生物信息学》和《植物基因组学》。
正文抢先看




<< 滑动查看下一张图片 >>
数字资源展示

<< 滑动查看下一张图片 >>

本文摘编自《生物信息学(第二版)》一书,内容有删减,标题为编者所加。

《生物信息学(第二版)》
樊龙江 主编
北京:科学出版社,2021.5
ISBN 978-7-03-068101-0
责任编辑:张静秋
READING
内容简介
本书主要介绍生物信息学基本概念、主要算法和常用工具。全书共15 章,涵盖生物分子数据产生、数据库、序列联配、基因组拼接及其基因预测、系统发生树构建、组学数据(转录、三维、单细胞等)分析、群体遗传分析等内容,同时包括生物信息学统计与算法基础和计算机基础。每章(绪论除外)前均设计了思维导图,帮助读者更好地理解各章知识点和逻辑关系;每章后均安排了一篇以相关领域代表人物为主线的“历史与人物”短文,有助于读者理解学科发展脉络。书后还提供了丰富的生物信息学资源(主流软件工具和数据库),作为学习本书的辅助资料。
本书可作为生物信息学等相关专业本科生和研究生的入门教材,也可供从事生物学、生物信息学及相关专业领域科研工作者阅读。
READING
本书目录
CONTENTS
第二版前言
第一版序
第一版前言
本书使用说明
致敬经典
第1章 绪论 1
第一节 生物信息与生物信息学 1
一、迅速增长的生物信息 1
二、生物信息学概念 2
第二节 生物信息学历史与展望 4
一、发展简史 4
二、应用领域 8
三、学科展望 9
习题 14
历史与人物“bioinformatics”之名的由来 14
第2章 生物信息类型及其产生途径 16
第一节 生物信息类型与测序技术 17
一、生物信息的类型 17
二、第一代测序技术 18
三、第二代测序技术 20
四、第三代测序技术 25
第二节 组学数据及其测定 28
一、基因组 28
二、转录组 30
三、其他组学数据 35
第三节 蛋白质序列及其结构测定 49
一、蛋白质序列与蛋白质互作测定 49
二、蛋白质结构测定 51
习题 52
历史与人物 第一台高通量测序仪与罗斯伯格 53
第3章 分子数据库 54
第一节 分子序列数据库概述 54
一、分子数据库及其记录格式 54
二、数据库序列递交与检索 58
第二节 核苷酸序列相关数据库 61
一、核苷酸初级数据库 61
二、核苷酸二级数据库 65
第三节 蛋白质相关数据库 69
一、蛋白质序列与结构数据库 69
二、蛋白质功能域等其他数据库 73
习题 77
历史与人物 分子数据库与戴霍夫和戈德 78
第4章 两条序列联配及其算法 80
第一节 序列联配与计分矩阵 80
一、序列联配 80
二、计分矩阵 81
第二节 两条序列联配算法 87
一、Needleman-Wunsch算法 87
二、Smith-Waterman算法 91
第三节 BLAST算法及数据库搜索 93
一、BLAST算法 93
二、利用BLAST进行数据库序列搜索 94
三、序列相似性的统计推断 103
习题 105
历史与人物 序列联配算法与三个“man” 106
第5章 多序列联配及功能域分析 109
第一节 多序列联配算法 109
一、多序列全局联配算法 109
二、多序列局部联配算法 111
第二节 蛋白质序列功能域 115
一、功能域概念 115
二、功能域模型 116
第三节 熵与信息量 119
一、熵与不确定性 119
二、多序列联配结果的信息量估计 121
习题 122
历史与人物 基序、ClustalW与杜立特 122
第6章 系统发生树构建 124
第一节 系统发生树概述 124
一、系统发生树的概念 124
二、遗传模型 127
第二节 距离法 129
一、UPGMA法 129
二、Fitch-Margoliash法 131
三、邻接法 134
四、最小进化法 136
第三节 最大似然法 137
一、DNA序列的似然模型 137
二、基于最大似然法建树 138
第四节 其他方法 141
一、最大简约法 141
二、贝叶斯法 143
三、基因组组分矢量法 144
习题 146
历史与人物 邻接法、MEGA与根井正利 146
第7章 基因组调查、拼装与分析 148
第一节 基于字符串的基因组调查分析 148
一、基因组大小估计 149
二、基因组复杂度估计 149
第二节 基因组序列拼接与组装 151
一、基因组测序策略与步骤 151
二、基因组序列拼接算法 154
三、基因组染色体水平组装 163
第三节 基因组序列分析与比较 166
一、基因组序列构成分析 166
二、基因组可视化 174
三、比较基因组学分析 175
第四节 基因组重测序数据分析 181
一、分析流程与变异鉴定方法 181
二、泛基因组分析 185
习题 187
历史与人物 文特尔和帕夫纳的神来之笔 188
第8章 基因预测及其功能和结构注释 190
第一节 蛋白质编码基因预测 190
一、基因预测方法及其流程 190
二、隐马尔可夫模型预测方法 196
第二节 基因功能注释 199
一、基于已知基因和功能域数据 199
二、基于功能分类和代谢途径 200
第三节 蛋白质结构预测 202
一、蛋白质结构概述 202
二、蛋白质二级和三级结构预测 204
三、基因突变与蛋白质三维结构功能分析 209
习题 212
历史与人物 HMM、马尔可夫及其他 212
第9章 非编码RNA鉴定与功能预测 214
第一节 小RNA计算识别与靶基因预测 214
一、miRNA主要特征及计算识别 214
二、siRNA主要特征及计算识别 220
三、小RNA靶基因预测 223
第二节 长非编码RNA鉴定与功能预测 225
一、lncRNA鉴定与功能预测 225
二、circRNA鉴定与功能预测 229
习题 235
历史与人物 首届中国生物信息学终身成就奖 236
第10章 基因转录与调控网络 238
第一节 转录组数据分析 238
一、转录组序列比对和拼接 239
二、基因表达分析 241
三、 基因可变剪接与融合 244
四、基因簇鉴定 249
第二节 甲基化分析 252
一、DNA甲基化 252
二、RNA甲基化 254
第三节 基因调控网络分析 256
一、生物网络 256
二、基因调控网络 259
习题 263
历史与人物 DNA自动测序仪、系统生物学与胡德 264
第11章 宏基因组分析 265
第一节 16S rRNA等基因序列数据 265
一、质控与分析流程 267
二、物种多样性估计 269
三、群落结构分析 272
第二节 全基因组序列数据 275
一、分析流程及其主要工具 275
二、宏基因组拼接与物种注释 279
习题 284
历史与人物 16S rRNA、生命之树与乌斯 284
第12章 新类型组学数据分析与利用 286
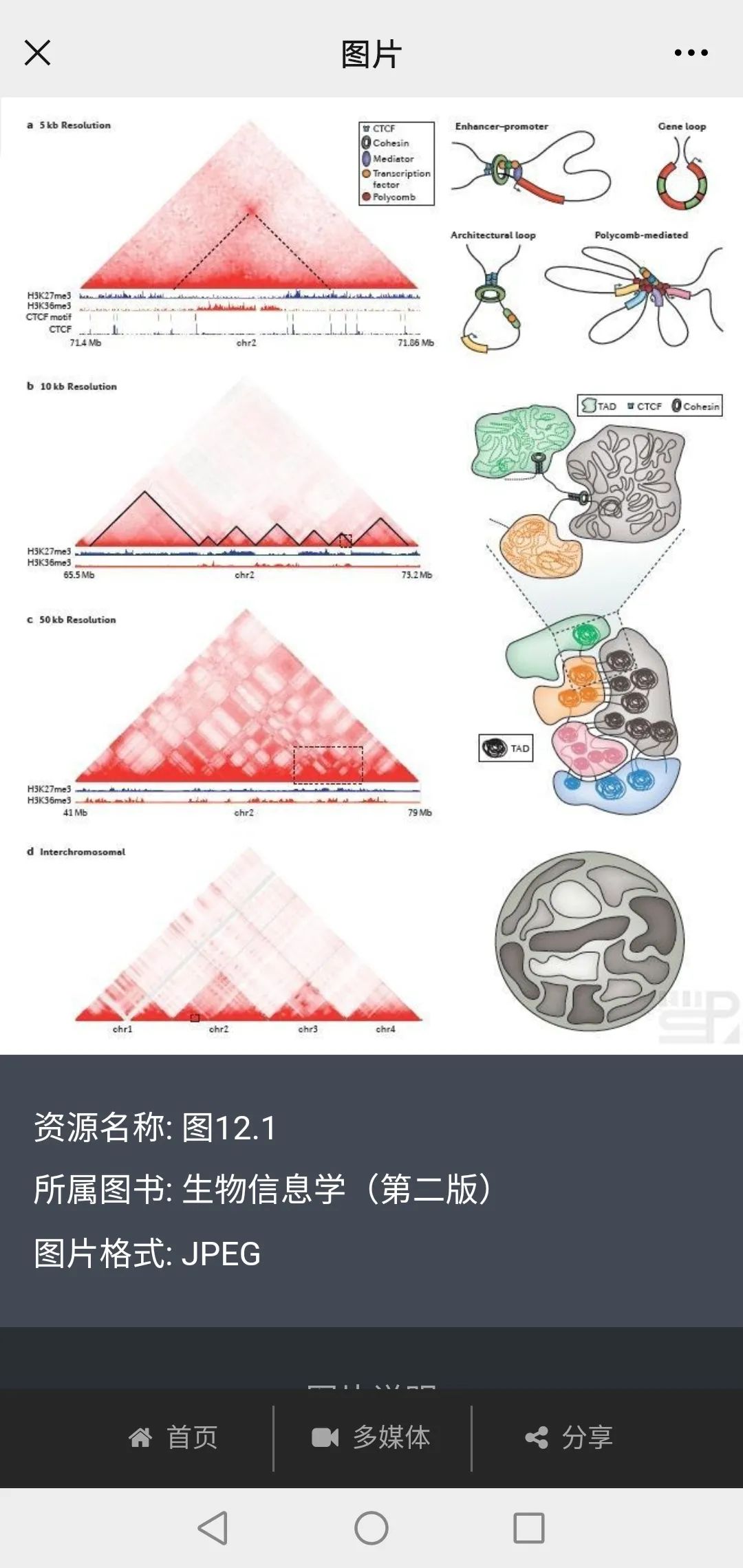
第一节 三维基因组 286
一、三维基因组数据标准化 286
二、染色质三维多级结构鉴定 289
三、三维基因组组装与可视化 292
第二节 单细胞组学数据 295
一、单细胞组学技术概况 295
二、单细胞基因组分析 297
三、单细胞转录组分析 299
第三节 基因组预测与选择 306
一、基因组数据与动植物育种 306
二、复杂性状的基因组预测与选择 311
第四节 其他 315
一、表型组之图像识别 315
二、合成生物学之基因组设计 323
三、翻译组 328
习题 331
历史与人物 深度学习“三剑客” 332
第13章 群体遗传分析 334
第一节 群体遗传多态性与结构分析 334
一、遗传多态性及其估计 335
二、群体遗传结构分析 337
第二节 自然选择的统计检验 339
一、基于种内多态性的检验方法 341
二、基于种间分歧度的检测方法 345
第三节 种群历史的溯祖分析 347
一、溯祖理论与溯祖模拟 347
二、种群进化模型的溯祖测验 349
三、有效群体大小的溯祖估计 352
第四节 数量遗传学分析 355
一、QTL定位 355
二、全基因组关联分析 362
三、混池分离分析 369
习题 372
历史与人物 马莱科特和科克汉姆的“神器” 373
第14章 生物信息学统计与算法基础 375
第一节 贝叶斯统计 376
一、贝叶斯统计概述 376
二、贝叶斯统计与生物信息学 379
三、图论与概率图模型 380
第二节 概率图模型 381
一、隐马尔可夫模型 381
二、贝叶斯网络 388
三、神经网络 391
第三节 机器学习算法 396
一、最大期望算法 396
二、马尔可夫链蒙特卡罗方法 398
三、动态规划 400
四、遗传算法 401
习题 405
历史与人物 贝叶斯之谜 406
第15章 生物信息学计算机基础 407
第一节 Unix/Linux操作系统 408
一、系统特点及其结构 408
二、Linux Shell常用命令 409
第二节 计算机编程语言 411
一、计算机编程语言概述 411
二、Python语言与Biopython简介 413
三、R语言与Bioconductor简介 415
四、MySQL语言 415
第三节 其他 418
一、并行化 418
二、算法与画图 423
习题 426
历史与人物 Python语言与范罗苏姆 427
主要参考文献 429
附录1 生物信息学常用代码和关键词 430
附录2 生物信息学主要数据库与分析工具 435
附录3 生物信息学常用英文术语及释义 440
中文名词索引 441
英文名词索引 444
后记 447
想了解更多内容?

长按识码,即刻购书
科学商城

京东(科学出版社)

当当(科学出版社)


推荐阅读


(本期编辑:王芳 )

一起阅读科学!
科学出版社│微信ID:sciencepress-cspm
专业品质 学术价值
原创好读 科学品味

科学出版社 视频号
硬核有料 视听科学
传播科学,欢迎您点亮★星标,点赞、在看▼























 946
946

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








