FastQC使用与结果详细解读
一 、FastQC用法

- 简单用法
fastqc -o outputfile inputfile
- 重要参数
-o 结果文件输出路径,需要自己事先建立好文件夹,默认输出文件是压缩文件,加--noextract则不压缩
-t 设置所使用的核数
-f 强制指定输入文件格式,默认自动检测
-c 污染物选项,输入的是一个文件,格式是Name[Tab] Sequence,#开头的行是注释,里面是可能的污染序列,如果有这个选项,FastQC会在计算时候评估污染的情况,并在统计的时候进行分析
-q 会进入沉默模式,指定这个选项的时候,程序不会实时报告运行的状况
二、FastQC结果详细解读

输出结果有一个.html问价和一个压缩文件,.html文件用浏览器打开即可得到FastQC Report
-
Summary

绿色:PASS
黄色:WARN(需要查看结果)
红色:FALL -
Basic Statistics:基本信息统计

Filename : 文件名
File type : 文件类型
Encoding : 测序平台的版本和相应的编码版本号,用于计算Phred反推error P时用
Total Sequences : 输入文本的reads数量
Sequence flagged as poor quality : 测序长度
Sequence length : 测序长度
%GC : 表示整体序列的GC含量,由于二代测序GC偏好性高,GC含量会高 -
Per base sequence quality:序列测序质量统计

横轴为read长度,纵轴为质量得分,Q = -10*log(error P),Q20表示1%的错误率,Q30表示0.1%的错误率;
柱状表示该位置所有序列的测序质量的统计,柱状是25%-75%区间质量分布,error bar 是10%-90%区间质量分布,蓝线表示平均数;
一般要求所有位置的10%分位数大于20,即最多允许该位置10%的序列低于Q20。当任何碱基质量低于10,或者任何中位数低于25报告WARN,当任何碱基质量低于5或任何中位数低于20报告FALL。 -
Per tile sequence quality:每个tile的序列质量
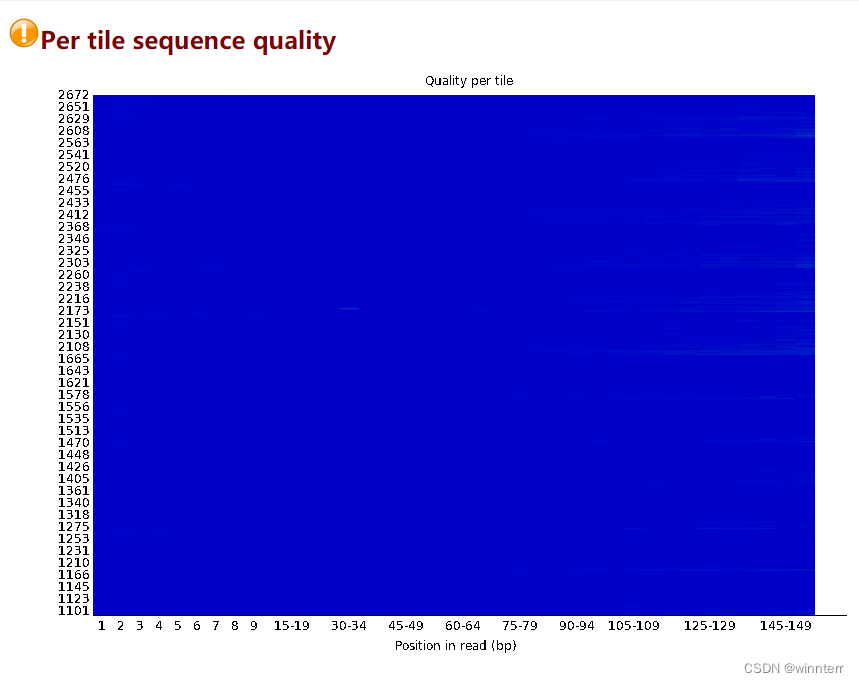
每个tile的测序质量,横坐标表示序列的长度位置从1到N,纵坐标是tile的编号,蓝色表示测序质量很好,颜色越红越不好 -
Per sequence quality scores:序列的测序质量
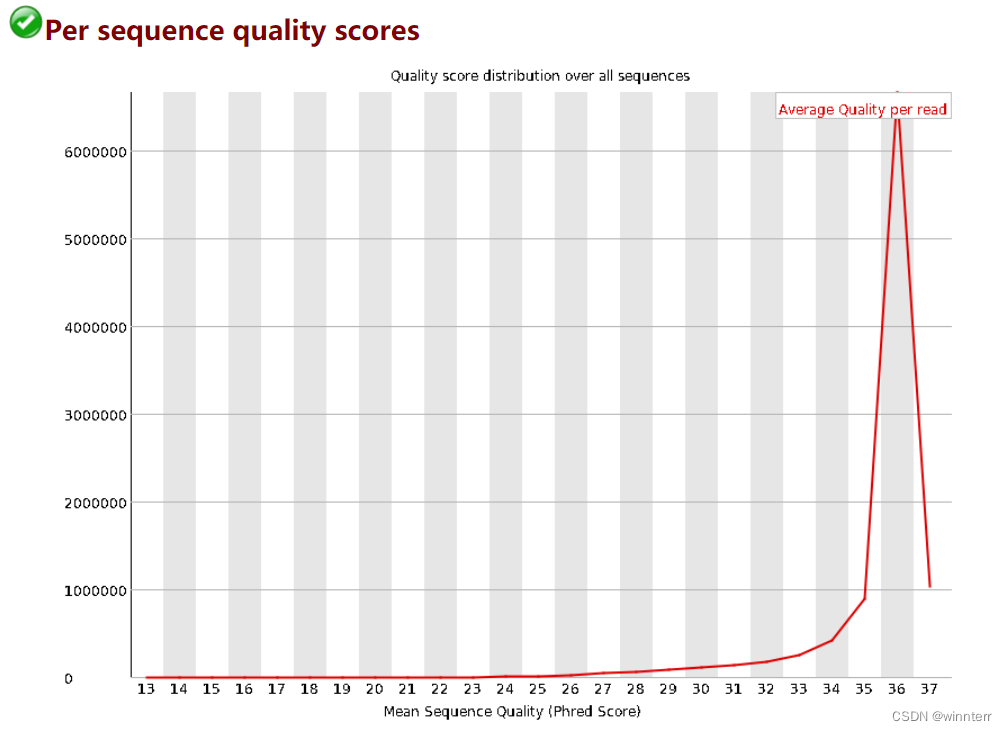
横轴:Q值
纵轴:每个Q值对应的read数
用来查看碱基质量是否存在普遍过低的情况,绝大部分的碱基的Q值的峰值大于20,可判断序列质量较好,在质量较低的坐标位置出现另一个或多个峰,说明测序数据中有一部分序列质量较差,当峰值小于27(错误率20%)时报WARN,当峰值小于20(错误率1%)时报FALL -
Per base sequence content:序列的测序质量

横轴:各碱基位置
纵轴:碱基百分比
四条线四种颜色代表四种碱基在每个位置的平均含量(一个位置会测很多reads,然后求一个平均)
一般来讲,A=T,C=G,但是由于刚开始测序仪状态不稳定,造成前几个碱基有波动。一般不是波动特别大的,像这里cut掉前5bp就好了。另外如果A、T或C、G间出现偏差,只要在1%以内都是可以接受的。 -
Per sequence GC content

横轴:平均GC含量
纵轴:每个GC含量对应的序列含量
蓝线为系统计算得到的理论分布;红线为测量值,二者越接近越好
偏离理论分布的reads超过15%时,报WARN,超过30%时,报FALL
曲线和蓝线不一致可能的情况有:
- 如果出现两个或多个峰值,表明测序数据里可能有其他来源的DNA序列污染,或者有接头的二聚体污染;
- 曲线形状的偏差往往时由于文库的污染或是部分reads构成的子集有偏差(overrepresented reads);
- 形状接近正态分布但偏离理论分布的情况提示我们可能有系统偏差;
-
Per base N content:统计reads每个位置N的比率

N是指一起不能识别ATCG时给出的结果,一般不会出现。但是如果出现并且量还很大,应该就是测序系统或者试剂的问题
任意位置的N的比例超过5%,报WARN,超过20%,报FALL -
Sequence Length Distribution:reads的长度分布

理想情况下,测得的序列长度应该是相等的,实际情况上总有些偏差
当reads长度不一致时报WARN,当有长度为0的read时报FALL -
Sequence Diplication Levels:重复reads的次数统计

横轴:duplication的次数
纵轴:duplicated reads的数目(红线)
以unique reads的总数作为100%,上图的情况中,相当于unique reads数目的15%的reads是观察到两次重复的,大约3%是观察到3次重复的,以此类推
测序深度越深,越容易产生一定程度的重复,但重复程度很高,可能是有偏差的存在(如建库过程中的PCR duplication)。
FastQC中用测序数据的前200000条reads统计其在全部数据中的重复情况
当非unique的reads占总数的比例大于20%时,报WARN,大于50%时,报FALL -
Overrepresented sequences:大量重复序列

如果有某个序列大量出现,就叫做over-represented,FastQC的标准是占全部reads的0.1%以上,为计算方便只取测序数据的前200000条reads进行统计,所以有可能over-represented reads不在里面。而>75bp的reads也是只取50bp。若在运行时加入-c contaminant file,出现的over-represented sequence会从contaminant file里面照匹配的hie(至少20bp且最多一个mismatch)
发现超总数0.1%的reads报WARN,超过总数1%报FALL
- Adapter Content:接头含量

表示序列中两端adapter的情况
软件中内置了四种常用的测序接头序列,fastqc有一个参数-a可以自定义接头序列
此图中使用的illumina universal adapter并未去除,后期再使用cutadapt去接头
-Kmer content:重复短序列
在序列中某些特征的短序列重复出现的次数























 2212
2212

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








