
为了系统展示itol.toolkit(辅助iTOL进行可视化的R包)的多个功能协同使用,我们选择了一些用iTOL制作并已发表的图来复现。

本期的主题是群落组装机制,这是近些年微生物生态领域的大热门。
前情
微生物生态学的主要目标之一是确定微生物群落组装过程中确定性过程(例如变量选择)和随机过程(例如扩散限制)的相对贡献。了解微生物多样性建立和维持的基本机制对于弄清微生物群落与生态系统功能之间的关系以及破译微生物群落对环境变化的反应和反馈至关重要。微生物群落的组成有两个重要而互补的过程:确定性过程(基于生态位理论),其中微生物群落的模式受环境过滤和各种生物相互作用的控制;和随机过程(中性理论),强调概率扩散和生态漂移的作用。
确定性过程包括由非生物和生物因素强加的生态选择,这些因素会影响有机体的适应性。异质选择(HeS)意味着空间或时间环境变化从当地物种库中选择优势物种,产生高β多样性,而同质选择(HoS)由于同质的非生物和生物环境条件而降低了β多样性。随机过程主要包括生态漂移、随机出生、随机死亡和扩散事件。尽管人们普遍认为确定性和随机过程同时运行,但它们在形成细菌群落组装方面的相对重要性各不相同。
基于系统发育bin的 🔗 零模型分析(iCAMP) ,以确定确定性和随机过程对细菌群落组装的潜在贡献。观察到的分类群首先根据它们的系统发育关系分为组(“箱”)。然后计算了bin内beta网络相关性指数(βNRI)和修正的Raup-Crick度量(RC),以评估每个bin中不同生态过程的相对重要性。对于每个箱,βNRI<-1.96的成对比较被认为受同质选择控制,而βNRI>+1.96的成对比较受异质选择控制。接下来,使用分类多样性度量RC与|βNRI|≤1.96划分其他过程。当RC<-0.95时被视为均质扩散过程,而RC>+0.95时为扩散限制过程。|βNRI|≤1.96且|RC|≤0.95代表漂变等过程的影响。具体计算方法可以参考:🔗 R语言iCAMP教程
本篇我们主要关注计算所得的结果如何进行可视化,可以更好地展示信息。
常规方案
在大多数已发表论文中,使用饼状图或堆积柱形图来表示不同过程的占比。





但当需要重点围绕群落组装机制进行讨论时,常常需要关联其他信息,如Bin的物种信息、丰度信息等。这时就很欠缺可视化表达手法。目前较为系统的可视化方法有如下三个:



这三个可视化作品所使用的信息是一致的。只是应用场景稍有不同:第一种当Bin数量巨大时效果较好,第二种当各Bin在系统发育树上分布占比较为均匀时可行,第三种适合常规及较少Bin数量的场景。
本篇我们对更多情况下会用到的第三种可视化方法进行复现。
数据
本节文档使用的数据来自Lei Zheng等2022年发表在Environmental Pollution的论文Comparing with oxygen, nitrate simplifies microbial community assembly and improves function as an electron acceptor in wastewater treatment。这项研究使用基于高通量测序方法的分子生态网络、零模型和功能预测分析,解析了处理生物废水的关键过程中微生物组的特征和组装机制,阐释了电子受体对菌群功能的影响有助于提高废水处理效率。
通过作者的帮助,我们获得了数据,数据存放在:https://github.com/TongZhou2017/itol.toolkit/discussions/13
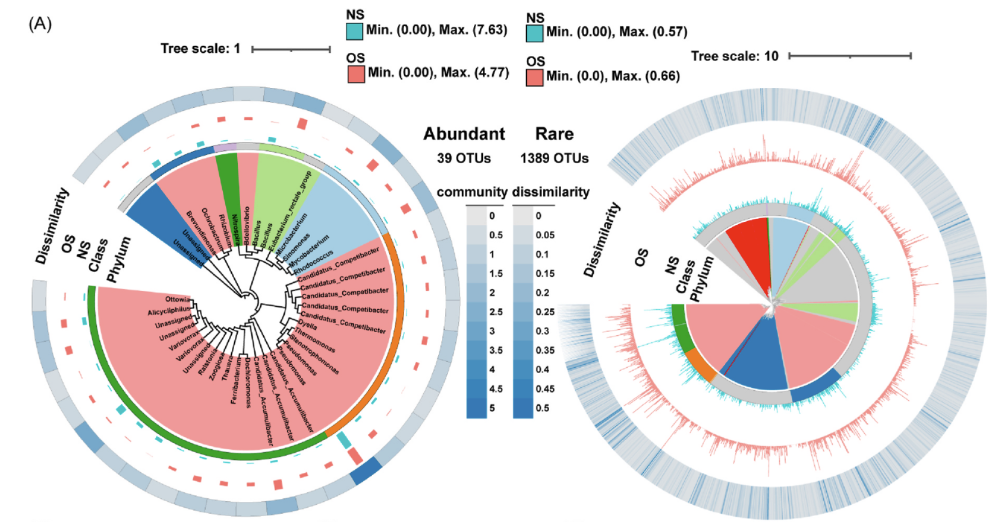
其中Fig3路径下是对丰富和稀有物种组间丰度差异以及门、纲水平下物种组成进行可视化所需的系统发育树及元数据。该部分所需的技能大部分与 🔗 之前的复现重叠,所以会在下面的篇幅简略提及。

我们本篇重点介绍Fig4路径下对iCAMP结果的复现,数据包括了Bin系统发育树以及元数据。
考虑到网络访问问题以及为了方便用户学习,将本文所需的全部代码及对应版本的安装包打包放在公众号后台,留言“itol.toolkit”即可获得。
Fig3 左图
这是一个由高丰度物种构成的小树。其中相对于作者使用热图模版来表示差异度,对于单列数据,我们优化选择了更为匹配的DATASET_GRADIENT模版功能来实现。
键(key)信息的书写使用了rep表示复现系列文档,Zheng2022ep表示论文信息,3al表示图表位置,第一个数字表示推荐拖入顺序,之后的字符串表示所使用的模版类型,如果同文档中使用了多个同类模版,则会在最后追加序号。
# install.packages("BiocManager")
#BiocManager::install("Biostrings")
# install.packages("devtools") # if you have not installed "devtools" package
#devtools::install_github("TongZhou2017/itol.toolkit")
library(itol.toolkit)
library(data.table)
tree_1 <- "./Fig.3/Abundant-tree/abunt-tree.nwk"
hub_1 <- create_hub(tree_1)
data_file_1 <- "./Fig.3/Abundant-tree/metadata.txt"
data_1 <- data.table::fread(data_file_1)
# relabel by genus
unit_1 <- create_unit(data = data_1 %>% select(ID, Genus),
key = "rep_Zheng2022ep_3al_1_labels",
type = "LABELS",
tree = tree_1)
# tree_colors range by phylum
unit_2 <- create_unit(data = data_1 %>% select(ID, Phylum),
key = "rep_Zheng2022ep_3al_2_range",
type = "TREE_COLORS",
subtype = "range",
tree = tree_1)
# color_strip by class
set.seed(123)
unit_3 <- create_unit(data = data_1 %>% select(ID, Class),
key = "rep_Zheng2022ep_3al_3_strip",
type = "DATASET_COLORSTRIP",
color = "wesanderson",
tree = tree_1)
unit_3@common_themes$basic_theme$margin <- 50
# simple_bar by NS
unit_4 <- create_unit(data = data_1 %>% select(ID, NS),
key = "rep_Zheng2022ep_3al_4_simplebar",
type = "DATASET_SIMPLEBAR",
tree = tree_1)
unit_4@specific_themes$basic_plot$size_max <- 100
# simple_bar by OS
unit_5 <- create_unit(data = data_1 %>% select(ID, OS),
key = "rep_Zheng2022ep_3al_5_simplebar",
type = "DATASET_SIMPLEBAR",
tree = tree_1)
unit_5@specific_themes$basic_plot$size_max <- 100
# simple_bar by OS
unit_6 <- create_unit(data = data_1 %>% select(ID, Dissimilarity),
key = "rep_Zheng2022ep_3al_6_gradient",
type = "DATASET_GRADIENT",
tree = tree_1)
#unit_6@specific_themes$heatmap$color$min <- "#0000ff"
#unit_6@specific_themes$heatmap$color$max <- "#ff0000"
hub_1 <- hub_1 +
unit_1 +
unit_2 +
unit_3 +
unit_4 +
unit_5 +
unit_6
write_hub(hub_1,getwd())
Fig3 右图
这是一个由低丰度物种构成的大树。在实际操作过程Chrome浏览器由于已经打开了过多页面,出现崩溃,更换使用Safari浏览器可以稳定渲染结果。举这个例子是为了提醒用户,当树过大或增加的注释信息维度过多时,可能会造成浏览器崩溃,这时可以尝试关闭不用的其他页面或打开一个其他浏览器尝试。
tree_2 <- "./Fig.3/Rare-tree/rare-tree.nwk"
hub_2 <- create_hub(tree_2)
data_file_2 <- "./Fig.3/Rare-tree/metadata.txt"
data_2 <- data.table::fread(data_file_2)
# relabel by genus
unit_7 <- create_unit(data = data_2 %>% select(ID, Genus),
key = "rep_Zheng2022ep_3ar_1_labels",
type = "LABELS",
tree = tree_2)
# tree_colors range by phylum
unit_8 <- create_unit(data = data_2 %>% select(ID, Phylum),
key = "rep_Zheng2022ep_3ar_2_range",
type = "TREE_COLORS",
subtype = "range",
tree = tree_2)
# color_strip by class
set.seed(123)
unit_9 <- create_unit(data = data_2 %>% select(ID, Class),
key = "rep_Zheng2022ep_3ar_3_strip",
type = "DATASET_COLORSTRIP",
color = "wesanderson",
tree = tree_2)
unit_9@common_themes$basic_theme$margin <- 50
# simple_bar by NS
unit_10 <- create_unit(data = data_2 %>% select(ID, NS),
key = "rep_Zheng2022ep_3ar_4_simplebar",
type = "DATASET_SIMPLEBAR",
tree = tree_2)
unit_10@specific_themes$basic_plot$size_max <- 100
# simple_bar by OS
unit_11 <- create_unit(data = data_2 %>% select(ID, OS),
key = "rep_Zheng2022ep_3ar_5_simplebar",
type = "DATASET_SIMPLEBAR",
tree = tree_2)
unit_11@specific_themes$basic_plot$size_max <- 100
# simple_bar by OS
unit_12 <- create_unit(data = data_2 %>% select(ID, Dissimilarity),
key = "rep_Zheng2022ep_3ar_6_gradient",
type = "DATASET_GRADIENT",
tree = tree_2)
#unit_6@specific_themes$heatmap$color$min <- "#0000ff"
#unit_6@specific_themes$heatmap$color$max <- "#ff0000"
hub_2 <- hub_2 +
unit_7 +
unit_8 +
unit_9 +
unit_10 +
unit_11 +
unit_12
write_hub(hub_2,getwd())
Fig4 群落组装图
到了重点。
加载数据,使用Newick格式的系统发育树作为输入,树的分枝为Bin。

同时加载元数据,其中包含了门阶元的分类信息,以及各类群落组装过程的指标数值和丰度信息。
tree_3 <- "./Fig.4/assemblytree.nwk"
hub_3 <- create_hub(tree_3)
data_file_3 <- "./Fig.4/metadata.txt"
data_3 <- data.table::fread(data_file_3)根据门阶元的分类信息对系统发育树进行背景色设置,使用 🔗 TREE_COLORS模版功能的range子类功能,在iTOL控制面板中选择Full全局作用范围,让原本只作用在标签的色块延伸到整个树。
# tree_colors range by phylum
unit_13 <- create_unit(data = data_3 %>% select(ID, Phylum),
key = "rep_Zheng2022ep_4a_1_range",
type = "TREE_COLORS",
subtype = "range",
tree = tree_3)
write_unit(unit_13,
paste0(getwd(),"/rep_Zheng2022ep_4a_2_range.txt"))
根据丰度信息,将高丰度的Bin所在分枝标注#。原文作者后期手动添加,这里优化使用了DATASET_TEXT模版功能实现同样效果。
top_10_NS <- data_3 %>% select(ID,`NS-bar`) %>% arrange(desc(`NS-bar`)) %>% head(n=10) %>% pull(ID)
top_10_OS <- data_3 %>% select(ID,`OS-bar`) %>% arrange(desc(`OS-bar`)) %>% head(n=10) %>% pull(ID)
RA_1 <- data_3 %>% mutate(pct = `OS-bar`+`NS-bar`/sum(data_3$`OS-bar`,data_3$`NS-bar`)) %>% select(ID, pct) %>% arrange(pct) %>% filter(pct > 1) %>% pull(ID)
RA_ids <- unique(c(top_10_NS,top_10_OS,RA_1))
df_text <- data.frame(id = RA_ids, text = rep("#",length(RA_ids)))
unit_14 <- create_unit(data = df_text,
key = "rep_Zheng2022ep_4a_3_text",
type = "DATASET_TEXT",
size_factor = 1,
position = 0.9,
rotation=0,
color = "#000000",
tree = tree_3)
write_unit(unit_14,
paste0(getwd(),"/rep_Zheng2022ep_4a_3_text.txt"))
使用热图展示各过程指标的占比程度。
df_cap_NS <- data_3 %>% select(ID, starts_with("NS"))
df_bar_NS <- df_cap_NS %>% select(ID,ends_with("bar"))
df_cap_NS <- df_cap_NS %>% select(-ends_with("bar"))
unit_15 <- create_unit(data = df_cap_NS,
key = "rep_Zheng2022ep_4a_4_heatmap",
type = "DATASET_HEATMAP",
tree = tree_3)
write_unit(unit_15,
paste0(getwd(),"/rep_Zheng2022ep_4a_4_heatmap.txt"))
使用简单柱形图展示各Bin丰度。
# simple_bar by OS
df_bar_NS$`NS-bar` <- round(df_bar_NS$`NS-bar`,3)
unit_16 <- create_unit(data = df_bar_NS,
key = "rep_Zheng2022ep_4a_6_simplebar",
type = "DATASET_SIMPLEBAR",
tree = tree_3)
unit_16@specific_themes$basic_plot$size_max <- 100
write_unit(unit_16,
paste0(getwd(),"/rep_Zheng2022ep_4a_6_simplebar.txt"))
同样的方法对另一组过程指标数据进行热图可视化。
df_cap_OS <- data_3 %>% select(ID, starts_with("OS"))
df_bar_OS <- df_cap_OS %>% select(ID,ends_with("bar"))
df_cap_OS <- df_cap_OS %>% select(-ends_with("bar"))
unit_17 <- create_unit(data = df_cap_OS,
key = "rep_Zheng2022ep_4a_7_heatmap",
type = "DATASET_HEATMAP",
tree = tree_3)
write_unit(unit_17,
paste0(getwd(),"/rep_Zheng2022ep_4a_7_heatmap.txt"))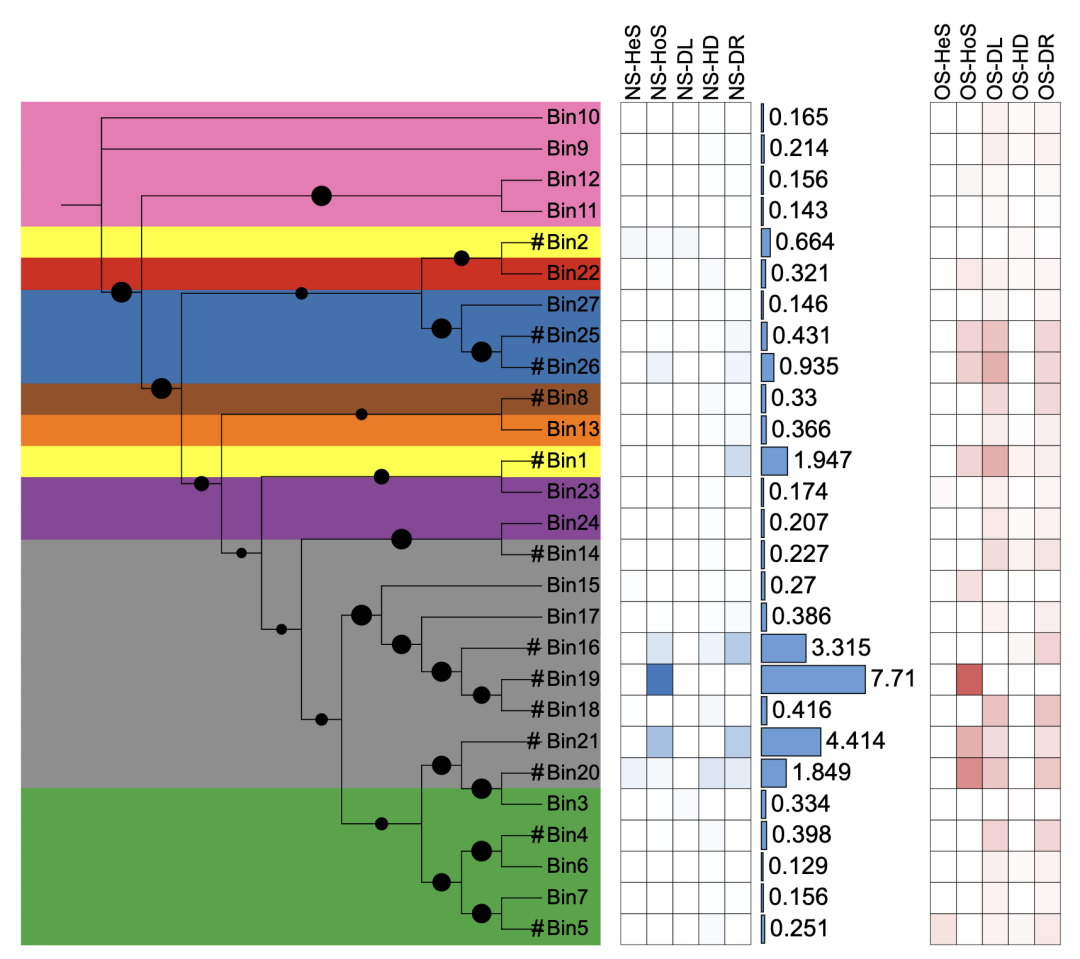
同样的方法对另一组丰度数据进行简单柱形图可视化。
# simple_bar by OS
df_bar_OS$`OS-bar` <- round(df_bar_OS$`OS-bar`,3)
unit_18 <- create_unit(data = df_bar_OS,
key = "rep_Zheng2022ep_4a_8_simplebar",
type = "DATASET_SIMPLEBAR",
tree = tree_3)
unit_18@specific_themes$basic_plot$size_max <- 100
write_unit(unit_18,
paste0(getwd(),"/rep_Zheng2022ep_4a_8_simplebar.txt"))
更新
itol.toolkit包的GitHub版更新了3个补丁版本 1.0.3、1.0.4、1.0.5
1.0.3添加了一个新功能:
DATASET_TEXT支持RStudio插件调控HTML样式文本
1.0.4添加了一个新功能:
DATASET_COLORSTRIP功能支持wesanderson配色盘
1.0.5添加了一个新功能:
增加itol.unit和itol.unit的+运算,合并同模版类型数据
社群
加作者微信(id:zitangumu),邀请进入itol.toolkit用户群。
用户群功能:
解决用户使用itol.toolkit过程中遇到的报错
将用户提出的功能需求纳入开发列表
协助用户使用itol.toolkit完成数据分析
文档类型:用户-工作流-rep_Zheng2022ep
链接状态:1
文档版本:1.0.4
文本:周通
编辑:凌浩
审阅:王雪,李明,赵启祥,张思威

END
TTfriends,是一个面向需求定制化开发生物信息分析工具的开发者团队,致力于推动科学前沿进步贡献新工具。欲了解更多详情,欢迎访问TTfirends官方网站:
tongzhou2017.github.io/ttfriends
Copyright © 2022 TTfirends
猜你喜欢
iMeta简介 高引文章 高颜值绘图imageGP 网络分析iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文,跳转最新文章目录阅读























 9604
9604

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








