做了泛基因组需要将结果可视化,这里记录一下可视化方法,其实有一个网站已经帮我们总结好了:https://pangenome.github.io/ ,有的是专门的可视化软件,有的是综合软件,不仅可以可视化还可以分析构建泛基因组,下面列出我用过的感觉不错的三个软件:Bandge,odgi,SequenceTube,各自绘图结果展示如下:
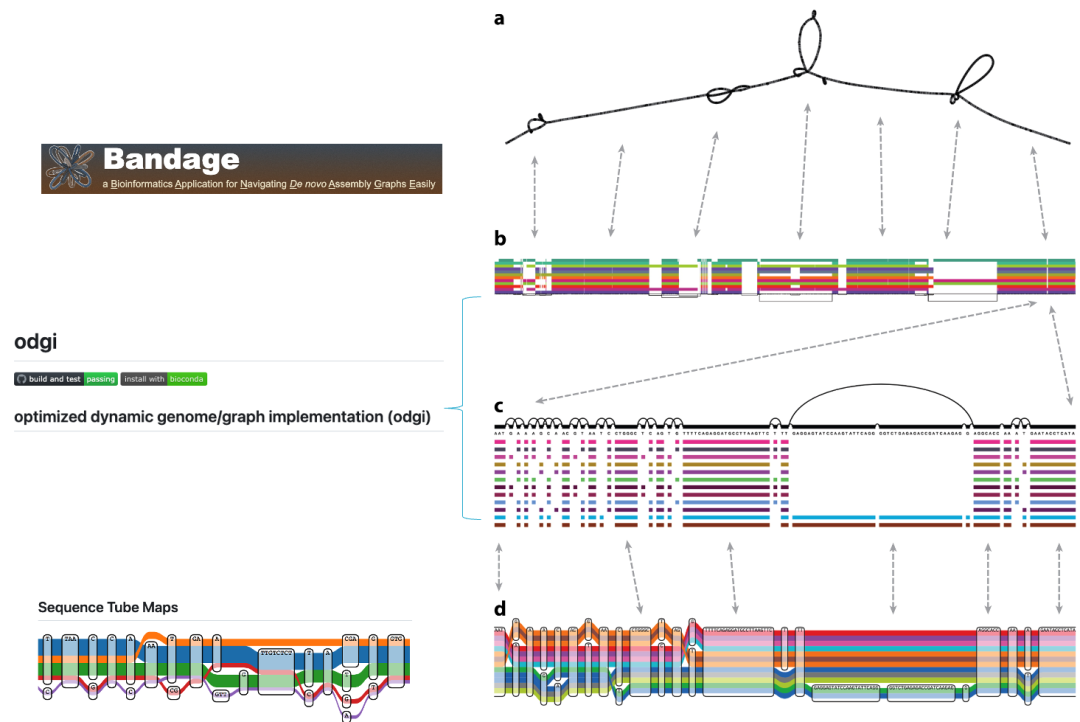
Bandage:https://rrwick.github.io/Bandage/, 图形可视化软件,支持输入GFA格式的文件可视化,在基因组组装中用的比较多,但是也可以展示泛基因组的结果;由于本人多用命令行工具,这个就不做过多介绍了。
odgi:https://github.com/pangenome/odgi,是一个专门用于可视化泛基因组的工具,是一个命令行的工具。主要做泛基因组不同单倍型的展示,结果也是挺好看的。
SequenceTube:https://github.com/vgteam/sequenceTubeMap,也是一个专门用于可视化泛基因组的工具,为网页版本,也提供一个示例网站:https://vgteam.github.io/sequenceTubeMap/。这个工具绘图最炫酷,还支持read比对结果可视化,方便后续特定基因不同单倍型分析,重点介绍。
使用:
1.odgi:
#设置环境变量,安装在export TMPDIR=`pwd`/tmpexport PATH=/share/work/biosoft/cactus/latest/bin/:$PATH#一般我们使用VG构建泛基因组,会有vg文件,可讲vg文件转换成og文件,就可以可视化odgi paths -i chr1.og -L >chr1.paths.txt#整条染色体#odgi viz -i pangenome/pangenome.chroms/chr1.og -o chr1.odgi.png -x 1500#排序odgi sort --optimize -i chr1.og -o chr1.sorted.og#查看vcf 文件找到感兴趣的位点展示:odgi extract -i chr1.sorted.og -n 518 -c 3 -o subregion518.og -d 10000 # ID 33 (-n 33), -d 距离 ,-c 附近的 node数量 -E #节点统计odgi stats -i subregion518.og -S >subregion518.paths.stat.txt#path IDodgi paths -i subregion518.og -L >subregion518.paths.txt#转换 GFA odgi view -i subregion518.og -g > subregion518.gfa #for Bandage view#绘图odgi viz -i subregion518.og -o subregion518.png -x 5000 #-z strand Black is forward, red is reverse.#获取FASTA 文件:odgi paths -i subregion518.og -f > subregion518.paths.fasta结果图如下,只是展示很小一个地方的情况:

2.SequenceTube使用:
这个工具是一个网页工具,默认端口是3000;先把要展示的数据文件链接到他的安装目录中的exampleData中,再启动服务就可以可视化查看感兴趣的位点了:
# 链接文件过去到这个文件夹:
cd /share/work/biosoft/sequenceTubeMap/sequenceTubeMap/exampleData/
# 链接文件 主要文件包括,
ln -s /work/ara_pangenome/pangenome* .
#启动服务可视化。记得启动docker时加上端口映射 -p 3000:3000
cd /share/work/biosoft/sequenceTubeMap/sequenceTubeMap
npm run serve #启动服务启动服务之后,打开电脑网页,输入运行这个服务的IP地址加端口号:http://192.168.101.5:3000/,即可打开网页,选择相应的文件和区域即可展示:官方的参考文档:https://github.com/vgteam/sequenceTubeMap/blob/master/public/help/help.md,按照下方图片中1-8步骤操作即可使用

可视化展示结果:

3.软件的安装
这些软件的安装大家可以看官方安装指导,链接我已经贴过了,如果省事的话我已经构建好镜像,可以使用docker镜像:
#下载镜像
docker pull omicsclass/pan-genome:v1.0
#启动镜像:
docker run --rm -it -v ~/pan-genome/:/work -p 3000:3000 omicsclass/pan-genome:v1.0






 本文介绍了三种用于泛基因组可视化的软件:Bandage、odgi和SequenceTube。Bandage适用于GFA文件,odgi专用于展示单倍型,而SequenceTube提供网页版并支持read比对。作者详细展示了如何使用这些工具进行数据分析和可视化。
本文介绍了三种用于泛基因组可视化的软件:Bandage、odgi和SequenceTube。Bandage适用于GFA文件,odgi专用于展示单倍型,而SequenceTube提供网页版并支持read比对。作者详细展示了如何使用这些工具进行数据分析和可视化。
















 1648
1648

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








