点击蓝字 关注我们
TCellSI:首个全面评估T细胞状态的创新工具

介绍

T细胞在免疫系统中扮演着至关重要的角色,其功能多样性不仅取决于不同的T细胞类型,还取决于其具体的功能状态。虽然当前已有多种计算工具可以预测T细胞的类型,但对于T细胞状态的全面评估仍然存在显著空白。为了解决这一问题,2024年6月21日,四川大学华西医院郭安源教授团队首次开发了一种能够系统地评估T细胞状态的新工具,TCellSI (T Cell State Identifier)。目前TCellSI文章处于审稿阶段。


核心功能

1. 全面评估T细胞状态
TCellSI能够准确评估T细胞的八种不同状态,这些状态包括:
● 静止(Quiescence)
● 调节(Regulating)
● 增殖(Proliferation)
● 辅助(Helper)
● 细胞毒性(Cytotoxicity)
● 初始耗竭(Progenitor exhaustion)
● 终末耗竭(Terminal exhaustion)
● 衰老(Senescence)
这些状态的评估结果被称为T细胞状态评分(TCSS, T Cell State Score)。通过这些评分,研究人员可以深入了解T细胞在免疫反应中的具体功能和角色。
2.高精度和可靠性
TCellSI借助Mann-Whitney U统计量,通过特定的标记基因集和参考谱为样本进行T细胞状态的评估。TCellSI在开发和验证过程中,涵盖了大规模数据,包括来自20个数据集的 379个 T 细胞系、34,730个单细胞、4,477个pseudo-bulk样本、33 种癌症类型的 10,535 名癌症患者、20种组织类型的 7,862个正常样本、免疫治疗队列中的 674个样本,以及 884个病毒感染的非癌症外周血样本。TCellSI不仅能准确描述T细胞状态,而且在免疫环境中显现出高度的预测价值。
3. 实际应用示例
● 癌症研究:TCellSI可以用于不同癌症类型中T细胞状态的评估,帮助研究人员理解肿瘤微环境中的免疫反应。
● 免疫治疗:通过评估患者T细胞的状态,TCellSI能够预测患者对免疫检查点阻断(ICB)治疗的响应,为个性化治疗方案的制定提供数据支持。
● 基础免疫学研究:TCellSI可以帮助研究人员探索T细胞在不同免疫环境中的动态变化,揭示T细胞功能的复杂性。
4. 易用性和可访问性
TCellSI提供了用户友好的R包 (https://github.com/GuoBioinfoLab/TCellSI/)和Webserver (https://guolab.wchscu.cn/TCellSI/),使研究人员可以轻松上传数据、进行分析并查看结果。用户可通过直观的图表和表格下载功能获取详细分析结果。
现有工具的局限性
● 功能范围有限:当前的许多工具如ImmuCellAI、CIBERSORTx、xCell等,主要关注预测T细胞的类型,如效应T细胞、调节性T细胞等。这些工具虽然可以提供T细胞类型的丰富信息,但无法深入了解这些细胞的功能状态。
● 缺乏状态评估:T细胞的功能不仅取决于其类型,更取决于其所处的具体状态。例如,效应T细胞在休眠状态与激活状态下的功能完全不同,但现有工具往往忽视这一点。
● 静态视角:大多数现有工具只能提供T细胞类型的静态快照,无法反映T细胞在不同状态下的动态变化。这种静态视角限制了我们对免疫反应过程的全面理解。
● 缺乏连续性评估:T细胞的状态通常是连续变化的,而不是离散的类别。现有工具将T细胞分类为离散类型,而忽视了它们在不同状态之间的连续变化。

TCellSI R package

安装
# install.packages("devtools")
devtools::install_github("GuoBioinfoLab/TCellSI")加载
library(TCellSI)评估T细胞的八种状态
## 示例表达数据
## 要求是完整的基因表达矩阵(log2转换后的RNA-seq TPM标准化基因表达矩阵)
sample_expression <- TCellSI::exampleSample
sample_expression[1:10, ]## SRR5088825 SRR5088828 SRR5088830 SRR5088833 SRR5088835
## 5_8S_rRNA 0.000000 0.000000 0.0000000 0.0000000 0.000000
## 5S_rRNA 0.000000 0.000000 0.4346853 0.0000000 0.000000
## 7SK 0.000000 0.000000 0.0000000 0.0000000 0.000000
## A1BG 1.907599 3.284418 4.0821248 3.2864715 2.863233
## A1BG-AS1 3.083914 2.021561 4.5169002 2.9347126 2.918992
## A1CF 0.000000 0.000000 0.0000000 0.0000000 0.000000
## A2M 9.812037 9.516751 5.7541683 6.9308912 7.394556
## A2M-AS1 2.107557 1.889085 0.1596072 0.8729146 1.206896
## A2ML1 0.000000 0.000000 0.0000000 0.0000000 0.000000
## A2ML1-AS1 0.000000 0.000000 0.0000000 0.0000000 0.000000## 进行TCSS的计算,TCSS是0-1之间的数值
ResultScores_TCSS <- TCSS_Calculate(sample_expression)## Calculating of Quiescence state## Calculating of Regulating state## Calculating of Proliferation state## Calculating of Helper state## Calculating of Cytotoxicity state## Calculating of Progenitor_exhaustion state## Calculating of Terminal_exhaustion state## Calculating of Senescence stateResultScores_TCSS## SRR5088825 SRR5088828 SRR5088830 SRR5088833 SRR5088835
## Quiescence 0.6422158 0.6153140 0.5105856 0.4520591 0.6951249
## Regulating 0.4404173 0.3974391 0.1670356 0.2615326 0.5454356
## Proliferation 0.5780474 0.5761891 0.3994404 0.4282601 0.6184267
## Helper 0.5647604 0.4063729 0.2121070 0.1735861 0.6547372
## Cytotoxicity 0.6482720 0.5826073 0.1829111 0.2358969 0.5888452
## Progenitor_exhaustion 0.5905679 0.5308304 0.2482848 0.2461412 0.6199635
## Terminal_exhaustion 0.6624943 0.6222188 0.3311532 0.3387099 0.6042142
## Senescence 0.5921543 0.5725185 0.2896508 0.3206251 0.6043355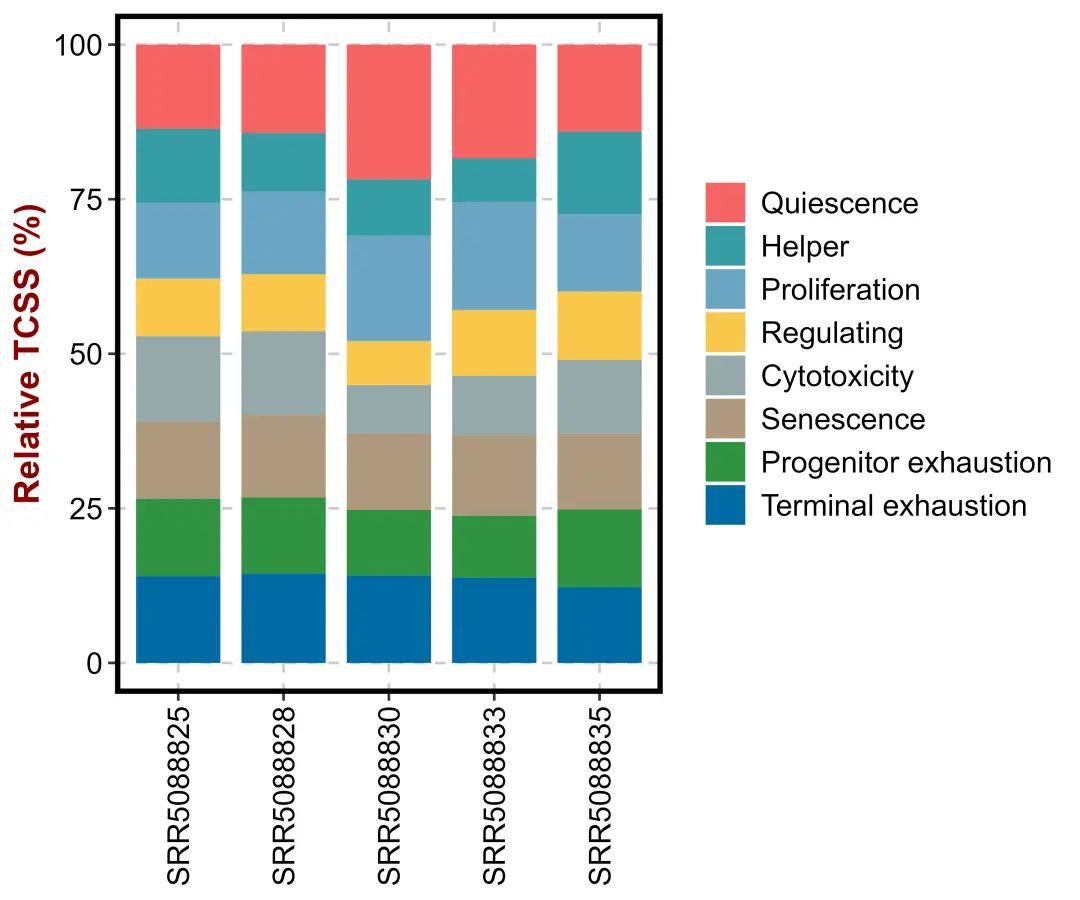
另外TCellSI的原理是通过特定的标记基因集和参考谱为样本进行T细胞状态的评估 那么内核的算法自然也可以应用于用户自定的标记基因集和参考谱进行其他细胞状态的计算(比如B细胞,巨噬细胞等)
下面介绍其他细胞状态的计算
2个需要的输入例子文件 链接:https://pan.baidu.com/s/1gF1ye7804QP6zvMjMYCVyQ 提取码:9v4c –来自百度网盘的分享
## 其他细胞状态的计算
sample_expression <- TCellSI::exampleSample
sample_expression[1:10, ]## SRR5088825 SRR5088828 SRR5088830 SRR5088833 SRR5088835
## 5_8S_rRNA 0.000000 0.000000 0.0000000 0.0000000 0.000000
## 5S_rRNA 0.000000 0.000000 0.4346853 0.0000000 0.000000
## 7SK 0.000000 0.000000 0.0000000 0.0000000 0.000000
## A1BG 1.907599 3.284418 4.0821248 3.2864715 2.863233
## A1BG-AS1 3.083914 2.021561 4.5169002 2.9347126 2.918992
## A1CF 0.000000 0.000000 0.0000000 0.0000000 0.000000
## A2M 9.812037 9.516751 5.7541683 6.9308912 7.394556
## A2M-AS1 2.107557 1.889085 0.1596072 0.8729146 1.206896
## A2ML1 0.000000 0.000000 0.0000000 0.0000000 0.000000
## A2ML1-AS1 0.000000 0.000000 0.0000000 0.0000000 0.000000## 读入自建参考谱
## 不同细胞状态的完整基因表达矩阵(log2转换后的RNA-seq或者scRNA-seq TPM标准化基因表达矩阵)
ref <- data.table::fread("CSS_example_ref.txt", data.table = F) %>% tibble::column_to_rownames("Symbol")
head(ref)## cell_state1 cell_state2 cell_state3
## A1BG 0.945000229 1.0214578020 0.432324502
## A1BG-AS1 0.510302271 0.6813711464 0.277653722
## A1CF 0.008817384 0.0005890896 0.001604985
## A2M 0.656710324 0.4461624408 4.106906902
## A2M-AS1 0.355709471 0.1774503922 1.950855264
## A2ML1 0.013431593 0.0152946350 0.007268041## 读入自定的marker基因集
## 多种细胞状态的marker基因列表
marker_list <- data.table::fread("CSS_example_markers.txt", data.table = F)
marker_list <- lapply(marker_list, function(i) {unique(i[i != ""])})
marker_list## $cell_state1
## [1] "BTG2" "KLF2" "FOXP1" "RUNX1" "MYC"
##
## $cell_state2
## [1] "CXCL13" "ENTPD1" "PRF1" "GZMH" "GZMB" "GZMA"
## [7] "BATF" "LYST" "TIGIT" "LAG3" "HAVCR2" "PDCD1"
## [13] "PRDM1" "RBPJ" "SLAMF7" "CCL4" "TNFSF10" "TBX21"
## [19] "NFATC1" "HIF1A" "TNFRSF4" "CXCR6" "PTPN7" "EWSR1"
## [25] "HLA-DPB1" "HLA-DPA1" "UBXN1" "PSMB9" "LY6E" "CCNDBP1"
## [31] "IRF9" "LCP2"
##
## $cell_state3
## [1] "CD3G" "CD3D" "CD3E" "CD247" "KLRG1" "B3GAT1" "TP53" "MAPK14"
## [9] "MAPK8" "MAPK1" "CDKN2A"##进行自定细胞状态评分(CSS)的计算
ResultScores_CSS <- CSS_Calculate(sample_expression, reference = ref, markers = marker_list)## Calculating of cell_state1 state : ( 1/3 )## Calculating of cell_state2 state : ( 2/3 )## Calculating of cell_state3 state : ( 3/3 )ResultScores_CSS## SRR5088825 SRR5088828 SRR5088830 SRR5088833 SRR5088835
## cell_state1 0.6422158 0.6153140 0.5105856 0.4520591 0.6951249
## cell_state2 0.6618459 0.6222132 0.3307533 0.3391375 0.6049080
## cell_state3 0.5913270 0.5720072 0.2893100 0.3209653 0.6039765
TCellSI Webserver功能及使用方法

下面对TCellSI Webserver的功能和具体使用方法进行系统的介绍。
TCellSI Webserver的功能和具体使用方法

1.主页
TCellSI 可以通过转录组数据评估八种不同的T 细胞状态:静息(Quiescence)、调节(Regulating)、增殖(Proliferation)、辅助(Helper)、毒性(Cytotoxicity)、初始衰竭(Progenitor exhaustion)、终末衰竭(Terminal exhaustion)和衰老(Senescence)。评估的结果被命名了T细胞状态评分(TCSS, T cell state score)。
2.分析(Analysis)界面

首先是文件上传部分,关于输入文件的准备,网站提供了例子文件,用户可以点击Expression Example, Group Example下载查看。
必须准备的就是Expression File,要求是完整的基因表达矩阵(log2转换后的RNA-seq TPM标准化基因表达矩阵),用制表符分隔,文件名以".txt "结尾。第一列是Symbol(第一列的列名请改成“Symbol”),其他列是不同样本的表达:

如果用户想在计算TCSS之后进行不同组别之间的TCSS的差异分析(非必须),那么需要准备进行Group File的准备和上传。第一列是Sample(第一列的列名请改成“Sample”),第二列是Group(第二列的列名请改成“Group”),请注意,Group File中的样本要和上面Expression File中的样本保持一致。
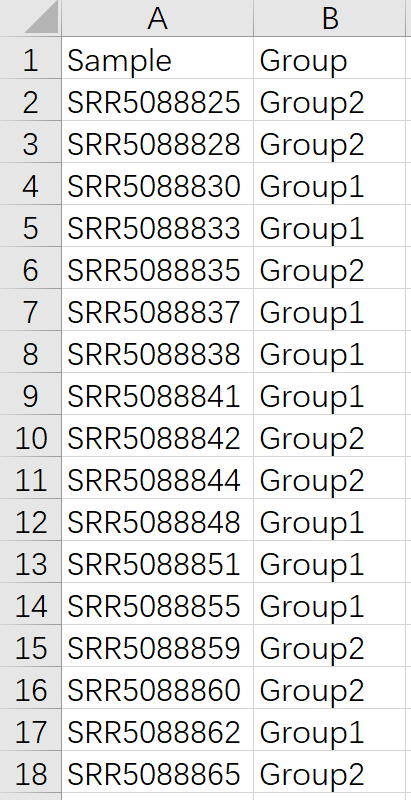

注意:上传文件的时候不要忘记点击“Upload”。


下面进行分析的选择,TCellSI提供了3个不同的分析,分别是:
● 通过转录组数据评估八种不同的 T 细胞状态。
● 评估不同组别中 T 细胞状态的变化。
● 预测对免疫检查点阻断(ICB)疗法的反应。

用户可以通过“Run Example”进行示例文件的计算,进而查看结果:


例子默认运行全部三个分析,页面最下面会展示结果:

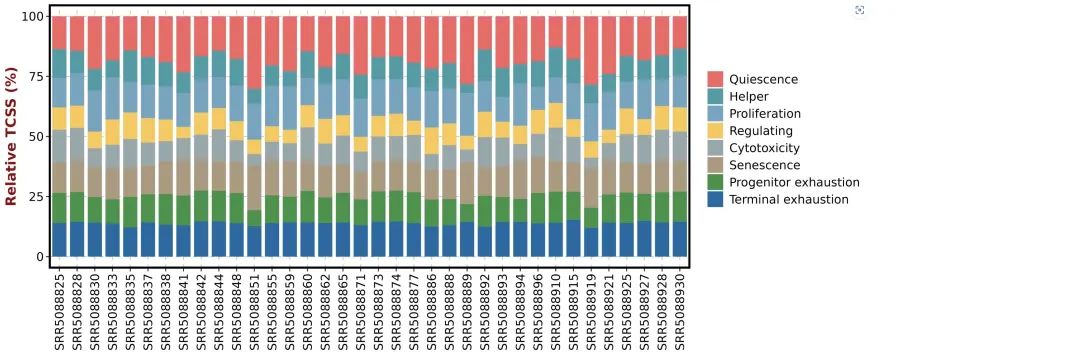
“T cell in samples”返回全部样本的8个T细胞状态评估的结果(TCSS),以及8个TCSS在样本中的相对关系。用户可以进行表格和图片结果的下载。


“T cell in groups” 返回不同组别之间T细胞状态的差异(Wilcoxon test)和分布情况。用户可以进行表格和图片结果的下载。

“ICB response” 返回全部样本的ICB治疗反应的预测结果(这里是通过毒性状态进行预测)。用户可以进行表格的下载。
3.资源(Resource)界面
TCellSI提供3个资源:差异(Difference),预后(Survival),Download(下载)。

差异(Difference)

通过此面板,用户可以分析 TCGA 和 GTEx 中肿瘤与正常组织之间不同 T 细胞状态的差异。T 细胞状态水平用箱式图显示,差异表达的统计学意义用 Wilcoxon 检验评估。用户需要选择感兴趣的癌症类型(以乳腺癌为例),点提交后,会得到差异分析的结果。


预后(Survival)

通过此面板,用户可以生成各种癌症类型中不同 T 细胞状态的Kaplan-Meier图,直观展示Overall Survival差异。每个图中都显示了比较两组生存曲线的对数秩检验的P值。用户需要选择感兴趣的癌症类型(以乳腺癌为例),点提交后,会得到生存分析结果。


Download(下载)

通过此面板,用户可以下载 TCGA 和 GTEx中肿瘤和正常组织的不同 T 细胞状态数据。用户需要选择感兴趣的癌症类型(以乳腺癌为例),点提交后,会得到表格。



4. 文档(Document)界面
提供了TCellSI的帮助文档。

TCellSI 微信交流群

或添加联系人入群:Y1390473501(杨靖敏)或AweKevin0119(张楠)
作者简介

郭安源
● 四川大学华西生物医学大数据研究院副院长、研究员,博士生导师,国家自然科学基金委“优青”(2018)。获2020年教育部高校科学研究优秀成果奖 (自然科学) 二等奖 (第一完成人),连续4年被评为爱思唯尔“中国高被引学者”(2020-2023),H-index=55。
● 研究方向: 肿瘤生物信息学,包括肿瘤多组学数据挖掘,肿瘤免疫及表达调控等的生物信息方法和数据库构建,及其在肿瘤和细胞外囊泡中的应用研究。
● 研究成果: 建立了一系列有一定国际影响的数据库,如AnimalTFDB、hTFtarget、miRNASNP、EVmiRNA和TCRdb等。开发了转录因子和miRNA在复杂疾病中的共调控方法FFLtool,癌症基因集分析平台GSCA和免疫细胞丰度预测方法lmmuCellAl等。开发了肿瘤免疫治疗联合用药的通用预测方法CM-Drug,发现多个免疫治疗联用药物并验证了taltirelin在黑色素瘤中提高anti-PD1治疗疗效的机制。在Nature lmmunology、Science Translational Medicine、Advanced Science、Cancer Research、Nature Communications、Nucleic Acids Research、Briefings in Bioinformatics等期刊发表论文100余篇,总引用15000多次,包括多篇ESI高被引论文或热点论文。
更多推荐
(▼ 点击跳转)
iMeta | 引用7000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
3卷1期
2卷2期封底
2卷4期封底
3卷2期
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(2023 IF 23.7)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、50万用户的社交媒体宣传等。2022年2月正式创刊发行!目前期刊已经被ESCI、PubMed、Scopus等数据库收录。
联系我们
iMeta主页:
http://www.imeta.science
姊妹刊iMetaOmics主页:
http://www.imeta.science/imetaomics/
出版社iMeta主页:
https://onlinelibrary.wiley.com/journal/2770596x
出版社iMetaOmics主页:
https://onlinelibrary.wiley.com/journal/29969514
iMeta投稿:
https://wiley.atyponrex.com/journal/IMT2
iMetaOmics投稿:
https://wiley.atyponrex.com/journal/IMO2
邮箱:
office@imeta.science





































 983
983

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








