点击蓝字 关注我们
揭秘沙门氏菌-宿主-微生物群在免疫与代谢中的相互作用

综 述
● 原文链接DOI: https://doi.org/10.1002/imo2.16
●2024年7月4日,徐州医科大学杨欢团队在iMetaOmics在线发表了题为“Salmonella—Host–microbiota interactions: Immunity and metabolism”的文章。
● 本研究总结了沙门氏菌如何通过免疫和代谢来促进其在宿主体内的扩张和复制,并对这一致病机制与宿主及微生物的关系进行概述,这有助于我们进一步提高对沙门氏菌肠道感染的理解。
● 第一作者:唐冰心
● 通讯作者:杨欢(yangh@xzhmu.edu.cn)
● 合作作者:崔雯雯、李晓
● 主要单位:徐州医科大学医学技术学院、徐州医科大学检验诊断学重点实验室、徐州医科大学
亮 点
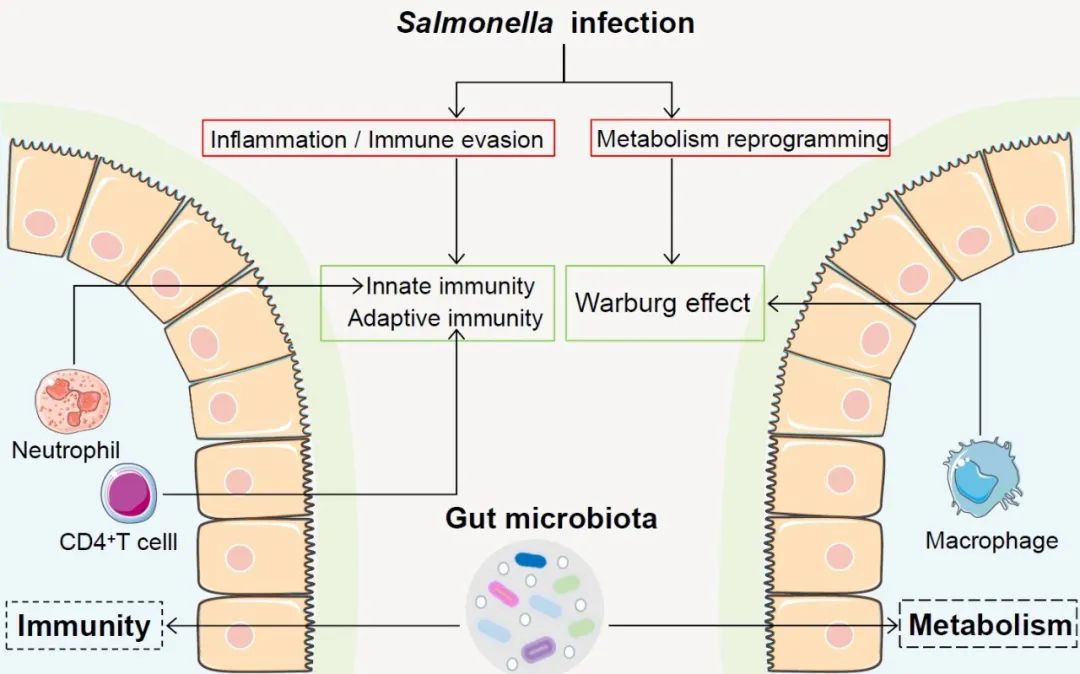
● 沙门氏菌可利用炎症、免疫逃避机制、重新编程宿主能量代谢等来促进其体内感染与增殖;
● 受感染巨噬细胞会重编程其氧化代谢,其特点是被诱发Warburg效应;
● 宿主通过先天免疫和适应性免疫来抵御其感染,其中肠道微生物群也可影响宿主免疫与代谢,以影响沙门氏菌感染。
摘 要
沙门氏菌是一类导致肠道感染的人畜共患病原菌。沙门氏菌通过三型分泌系统(T3SS)注入细菌效应物破坏肠黏膜上皮,进而侵入固有层,利用炎症、免疫逃避机制、重新编程宿主能量代谢等来建立系统性感染。受感染的巨噬细胞被诱发Warburg转移,其特点是糖酵解增加以及TCA循环抑制,宿主通过先天免疫和适应性免疫来抵御其感染。除此之外,肠道微生物可以通过影响宿主免疫与代谢来调节沙门氏菌对宿主感染。在这篇综述中,我们总结最近的进展,主要强调沙门氏菌致病机制、宿主代谢和肠道微生物群之间的关系。
视频解读
Bilibili:https://www.bilibili.com/video/BV1Ub421n7M9/
Youtube:https://youtu.be/hWdu8okKM9s
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/imetaomics/
全文解读
引 言
沙门氏菌是一类导致肠道感染的人畜共患病原菌,主要通过污染食物或水进行传播。沙门氏菌侵入引发肠道炎症会产生电子受体以促进其感染,还可以在受感染巨噬细胞中形成SCV形成免疫逃逸,躲避宿主免疫系统攻击。除此之外,被感染巨噬细胞会重编程其细胞氧化代谢,其中诱发的代谢途径产物积累也为沙门氏菌进一步感染提供能量来源和毒力激活信号,并为沙门氏菌建立系统感染奠定基础。
宿主一般可以通过两方面来抵御沙门氏菌感染,在沙门氏菌感染早期,主要激活先天免疫系统。其中不同的细胞发挥不同的作用,比如中性粒细胞为沙门氏菌早期急性感染产生大量IFN-g建立了良好的基础,以避免机体下一步感染更多的沙门氏菌。适应性免疫激活较慢,但在清除沙门氏菌反应中发挥主要作用,其中主要是CD4+T细胞,而缺乏B细胞或者CD8+T细胞耗竭并不会影响沙门氏菌最后的清除。
沙门氏菌与微生物群相互作用可通过不同的机制。本文主要总结两种不同的机制,包括通过互相竞争营养物质以此来影响宿主细胞免疫反应,还有一点值得注意的是,肠道微生物可以通过影响宿主细胞代谢对沙门氏菌作出反应。本篇综述主要总结了沙门氏菌如何通过免疫和代谢来促进其在宿主体内的扩张和复制,并对这一致病机制与宿主及微生物的关系进行概述,这有助于我们进一步提高对沙门氏菌肠道感染的理解。
沙门氏菌入侵及感染
沙门氏菌利用致病岛SPI编码III型分泌系统侵入机体。在肠上皮细胞中,沙门氏菌致病岛SPI-1编码的III型特殊蛋白质分泌系统T3SS-1可通过转移细菌效应蛋白,如SopE、SopE2、SopB、SipA等来激活Rho-GTP酶破坏肠上皮细胞之间的紧密连接。通过对上皮的入侵,沙门氏菌可在其胞质中进行复制促进其扩增并引发肠道炎症,这促进沙门氏菌对机体感染,一方面,炎症会带来一系列炎症衍生电子受体,如硝酸盐等,这不仅有利于促进沙门氏菌呼吸运动,还促使其利用琥珀酸盐等微生物衍生物。另一方面,炎症有助于扰乱肠道微生物平衡并促进沙门氏菌利用微生物衍生的各种发酵产物,如甲酸盐等。除此之外,沙门氏菌还可利用免疫逃避机制促进其感染,当沙门氏菌被巨噬细胞内吞后,可以在含沙门氏菌液泡(SCV)中进行复制,进一步诱导SCV酸化并利用CadC-YdiV轴感知这一信号,借此关闭鞭毛合成以实现对宿主细胞的免疫逃避。另外,针对宿主自噬防御机制,沙门氏菌也可利用SPI-1效应器SopB来介导自噬的逃逸。总而言之,沙门氏菌针对宿主引发的炎症及免疫防御有其独特的应对策略,这些都为其建立全身的感染奠定了基础。

图1. 沙门氏菌的入侵及其与肠道微生物群的相互作用
沙门氏菌利用其毒力因子SopB、SopE、SopE2和SipA激活Rho-GTPase信号并引发入侵。侵袭后,如中性粒细胞可分泌干扰素g(IFN-g),树突状细胞可分泌B细胞活化因子(BAFF)促进B细胞分泌免疫球蛋白M(IgM)抗体来抵御其感染。此外,炎症产生的电子受体有助于沙门氏菌利用微生物衍生的发酵产物琥珀酸,促进其在宿主中完整的三羧酸(TCA)氧化代谢。炎症也有助于沙门氏菌利用微生物衍生物甲酸盐等。此外,沙门氏菌与共生大肠杆菌等肠道微生物竞争铁、碳源和氧气等营养物质。沙门氏菌还可以通过消除梭菌介导宿主细胞代谢,从而导致宿主细胞从氧化代谢转变为乳酸发酵,从而增加上皮细胞的氧合。然而,当宿主PPAR-g信号被激活时,将减少乳酸的产生并与调节性T细胞相互作用以维持肠道缺氧,逆转这一代谢改变。
宿主细胞代谢
在系统性感染过程中,受感染的巨噬细胞被诱发Warburg转移,其特点是糖酵解增加以及TCA循环抑制。糖酵解过程上调将诱导糖酵解中间物积累,如磷酸甘油酸酯(3PG)、丙酮酸酯和乳酸盐等。其中3PG可作为沙门菌细胞内复制的碳源,使其从巨噬细胞葡萄糖低可用性中解放出来,而丙酮酸酯和乳酸盐则通过双组分系统CreBC激活SPI-2基因,此前有研究指出SseK3作为SPI-2效应器有助于促进糖酵解的上调,诱导巨噬细胞凋亡,同时抑制炎症因子的表达。需要指出的是,此前糖酵解上调被认为是宿主的一种保护作用,通过此途径宿主可以更快获得ATP来支持生物合成以满足细胞增殖代谢需求,但对人巨噬细胞的研究中发现抑制宿主细胞中糖酵解降低了沙门氏菌的生长,这与增加了ROS产量有关。另外,对于糖酵解所累积的乳酸,在小鼠中似乎是为了激活SPI-2做准备,而在人巨噬细胞中并不需要,其中的机制仍需进一步探明。
三羧酸循环受到抑制会导致其代谢中间体的水平显著下降,比如柠檬酸,但另外的琥珀酸、衣康酸盐的水平则是显著增加,这可能是宿主细胞应对沙门氏菌感染的应答方式之一。通过对巨噬细胞中柠檬酸盐研究发现,柠檬酸盐水平下降可促进细胞内脂质生物合成。上升的衣康酸盐可以使TFEB烷基化诱导溶酶体生成促进细菌清除。琥珀酸可以通过上调HIF-1a表达来增加IL-1b的表达,另外,HIF-1a表达抑制可能通过EGFR-HIF-1a轴降低沙门氏菌的生存能力,因为抑制该途径可以增强促炎细胞因子IFN-g、肿瘤坏死因子a(TNF-a)、白细胞介素17(IL-17)的产生,同时减少抗炎因子IL-10的分泌。总的来说,沙门氏菌可以重新规划受感染巨噬细胞的葡萄糖代谢,借此引起巨噬细胞衍生碳源和信号的充分积累,以支持它在细胞内复制和毒性。

图2. 宿主细胞代谢重编程
沙门氏菌感染诱导巨噬细胞的代谢重编程,利用其毒力因子效应子SopE2和SseK3来增强糖酵解过程,其中代谢产物如3-磷酸甘油酸(3PG)可被沙门氏菌用作复制的碳源,丙酮酸和乳酸可被沙门氏菌双组分系统CreBC感知,以激活沙门氏菌致病岛2(SPI-2)的表达。同时,TCA循环在巨噬细胞中受到抑制,代谢中间体衣康酸盐水平的增加可以通过转录因子EB(TFEB)烷基化诱导溶酶体的产生,并对细菌产生清除作用。此外,代谢中间体琥珀酸盐通过激活HIF-1a释放IL-1b来抑制沙门氏菌,但沙门氏菌也可以利用宿主中积累的琥珀酸盐作为毒力激活信号来防御宿主免疫。同时阻断HIF-1a的表达可以促进IFN-g、TNF-a和IL-17的分泌,抑制IL-10的分泌,这对沙门氏菌有益。
宿主对沙门氏菌免疫反应
沙门氏菌侵入肠道时,会有不同的先天免疫细胞对其作出应答。首先,面对沙门氏菌侵入,肠上皮细胞通过NAIP/NLRC4炎症小体进行识别,进而引发上皮层收缩防止沙门氏菌对上皮屏障的破坏。其次,巨噬细胞可通过释放细胞外陷阱ETosis来清除沙门氏菌。树突状细胞产生B细胞激活因子(BAFF)来刺激成熟B细胞扩张并产生针对沙门氏菌的特异性IgM抗体,这对预防沙门氏菌感染是必要的。
对沙门氏菌感染适应性免疫反应特征之一是CD4+T细胞和CD8+T细胞激活。CD4+T细胞可以通过产生IFN-g抵御沙门氏菌持久性感染,但是CD4+T细胞的保护在不同器官中可能会表现出异质性。在慢性感染小鼠中,通过研究发现与脾脏和淋巴结相比,肝脏CD4+T细胞除产生IFN-g外,还能产生IL-10,这抑制了沙门氏菌的清除。CD8+T细胞在沙门氏菌感染期间表现出延迟扩张和收缩,但其在沙门氏菌减毒活疫苗引发原发性感染中发挥了保护作用,其可能为疫苗的设计和研发提供重要线索。
沙门氏菌-微生物相互作用
共生大肠杆菌可以通过竞争铁、碳源来减少沙门氏菌的肠道定殖,碳源减少可提供热量限制(CR),并且以往的研究证实了CR可通过NO来减轻沙门氏菌感染。沙门氏菌可依赖铁载体从宿主中争夺铁,当阻断铁载体时,可有效减少肠道中沙门氏菌定殖。近期有研究指出肠道共生菌Bacteroides thetaiotaomicron可通过分泌铁载体结合脂蛋白XusB,并利用如沙门氏菌分泌的铁载体获取铁,但是这种铁载体与XusB结合物不易被脂蛋白-2所阻断,并且还能为沙门氏菌利用,使病原菌逃避宿主营养免疫。除此之外,氧气也是共生大肠杆菌与沙门氏菌的争夺品,大肠杆菌可通过与沙门氏菌竞争氧气来对沙门氏菌产生定殖抵抗。最新的研究还提出共生大肠杆菌与沙门氏菌在不同空间微栖息地获取关键资源,例如硝酸盐,大肠杆菌可获取上皮源性硝酸盐,但沙门氏菌受到其趋化受体McpB和McpC限制,使其无法利用上皮衍生的硝酸盐,而共生大肠杆菌可以入侵沙门氏菌营养利基与其竞争巨噬细胞来源硝酸盐增强对沙门氏菌定殖抗性。
沙门氏菌利用其毒力因子从肠道相关微生物群落中清除产生丁酸的梭菌,由此产生的上皮氧合增加促使沙门氏菌在肠腔内的细胞色素bd II氧化酶依赖性需氧扩张,这与硝酸盐呼吸驱动的扩张相互协助,以增强传播。有研究也证实产生丁酸的梭状芽孢杆菌的耗竭会引发宿主细胞从氧化代谢转向乳酸发酵,从而支持沙门菌感染,说明沙门氏菌可以通过打破肠道微生物平衡并促进自身氧化代谢这一途径来促进其感染和定殖。使用PPAR-g激动剂会减少宿主乳酸盐的产量,并废除沙门氏菌这一优势,PPAR-g信号激活后降低了沙门氏菌呼吸电子受体利用度并与结肠中调节性T细胞相协同维持结肠缺氧状态,这有助于肠道内微生物产生短链脂肪酸从而维持肠道平衡。利用这一激活信号,有望能从新陈代谢这一途径出发为治疗沙门氏菌所引起疾病提供新的思路。
讨 论
在沙门氏菌感染期间,早期炎症的产生虽然能为机体提供初步保护,但是其中所产生部分衍生物可以促进沙门氏菌呼吸运动,反而促使沙门氏菌从中获益。宿主可以启动先天及适应性免疫来抵御这种感染。但针对这些途径,沙门氏菌也有其独特免疫逃避方式。除此之外,沙门氏菌通过改变宿主代谢来促进其体内增殖,进一步加深了我们对沙门氏菌肠道感染的认识,但是其中分子机制可能还需要未来进一步探索。
此外,沙门氏菌感染会打破肠道中建立的菌群平衡,例如与肠道中共生菌共同争夺营养物质、影响肠道中微生物群落结构,利用肠道微生物代谢物促进自身能量代谢,目前的研究为我们提供了一些沙门氏菌在免疫和代谢方面与宿主和微生物之间相互作用的见解。对于未来,我们仍需要了解它们之间如何通过免疫与代谢相互调节作用的其他机制,这也为我们提供一个重要信号,未来将有望从调节体内代谢来影响机体免疫从而抵御病原菌感染。
代码和数据可用性
这篇稿件没有生成任何代码或数据。附加材料(图形摘要、幻灯片、视频、中文翻译版本和更新材料)可以在在线DOI或iMetaScience网站(http://www.imeta.science/)中找到。
引文格式:
Bingxin Tang, Wenwen Cui, Xiao Li, Huan Yang. 2024. Salmonella—Host–microbiota interactions: Immunity and metabolism. iMetaOmics 1: e16. https://doi.org/10.1002/imo2.16
作者简介

唐冰心(第一作者)
● 重庆医科大学临床检验诊断学专业学术型硕士在读。

崔雯雯
● 硕士研究生,目前工作于徐州市疾病预防控制中心。
● 主要从事致病菌与病毒检测方面工作。

杨欢(通讯作者)
● 医学博士,徐州医科大学副教授,硕士生导师。
● 研究方向为病原微生物感染与宿主免疫代谢相互作用机制,已在Nature communications、mBio、International Journal of Nanomedicine 等期刊发表学术论文30余篇,主持国家自然科学基金项目、江苏省自然基金项目、中国博士后面上课题等项目,相关成果获得2项专利,参编中英文论著2部。

李晓
● 医学博士,讲师。
● 主要研究方向为病原微生物的致病机制以及病原微生物与宿主的相互作用机制。
iMetaOmics
更多资讯
● iMeta姊妹刊iMetaOmics(定位IF>10)欢迎投稿!(2024.2.27)
● iMeta姊妹刊iMetaOmics编委招募 (定位IF>10) (2024.3.2)
● iMeta姊妹刊iMetaOmics电子版和印刷版ISSN申请获批(2024.4.1)
● iMeta姊妹刊iMetaOmics投稿系统正式上线(2024.4.17)
● iMeta姊妹刊iMetaOmics主编正式官宣(2024.4.22)
● 出版社iMetaOmics主页正式上线!(2024.4.28)
● iMetaOmics | 浙江大学宗鑫组揭示两猪种宿主-肠道菌群互作差异
● iMetaOmics | 罗鹏/袁硕峰/苗凯/程全发表STAGER: 生成式人工智能可靠性的标准化测试和评估推荐
更多推荐
(▼ 点击跳转)
iMeta | 引用7000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
3卷1期
2卷2期封底
2卷4期封底
3卷2期
3卷3期
3卷3期封底
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百千华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF > 20)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、前3年免出版费、50万用户的社交媒体宣传等。2022年2月正式创刊发行!发行后相继被Google Scholar、ESCI、PubMed、DOAJ、Scopus等数据库收录!2024年6月获得首个影响因子23.7,位列全球SCI期刊前千分之五(107/21848),微生物学科2/161,仅低于Nature Reviews,同学科研究类期刊全球第一,中国大陆11/514!
“iMetaOmics” 是“iMeta” 子刊,主编由中国科学院北京生命科学研究院赵方庆研究员和香港中文大学于君教授担任,定位IF>10的高水平综合期刊,欢迎投稿!
联系我们
iMeta主页:
http://www.imeta.science
姊妹刊iMetaOmics主页:
http://www.imeta.science/imetaomics/
出版社iMeta主页:
https://onlinelibrary.wiley.com/journal/2770596x
出版社iMetaOmics主页:
https://onlinelibrary.wiley.com/journal/29969514
iMeta投稿:
https://wiley.atyponrex.com/journal/IMT2
iMetaOmics投稿:
https://wiley.atyponrex.com/journal/IMO2
邮箱:
office@imeta.science








































 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








