点击蓝字 关注我们
OmicShare tools: 零代码交互式的在线生物数据分析与可视化工具

研究论文
● 期刊: iMeta (IF 23.7)
● 原文链接DOI: https://doi.org/10.1002/imt2.228
●2024年8月1日,广州基迪奥生物科技有限公司周煌凯、艾鹏和高川等在iMeta在线发表了题为“OmicShare tools: A zero-code interactive online platform for biological data analysis and visualization”的文章。
● 本研究介绍的OmicShare tools是一个用户友好且高效的数据分析与可视化在线工具,包含161款生物信息分析小工具,主要用于生物数据特别是高通量测序数据的分析与可视化。
● 第一作者:母红岩
● 通讯作者:周煌凯(hkzhou@genedenovo.com)、艾鹏(pai@genedenovo.com)、高川(chgao@genedenovo.com)
● 合作作者:陈建周、黄文杰、黄桂、邓美英、洪仕秒
● 主要单位:广州基迪奥生物科技有限公司
亮 点
● OmicShare tools是一个用户友好且功能强大的数据分析与可视化在线平台;
● OmicShare tools具有强大的交互式图形引擎,可对分析结果中的图表进行自定义调整;
● OmicShare tools的图表样式精美,支持导出矢量格式的文件,足以满足科学期刊的发表需求。
摘 要
OmicShare tools是一个用户友好且高效的数据分析与可视化在线工具,包含161款生物信息分析小工具,主要用于生物数据特别是高通量测序数据的分析与可视化。OmicShare tools简单易上手,一般只需保持默认的参数选项即可得到很好的效果。用户提交分析任务后,可在项目总览页面实时查看任务进度。OmicShare tools具有强大的交互式图形引擎,可对分析结果中的图表进行自定义调整。OmicShare tools的图表样式精美,支持导出矢量格式的文件,足以满足科学期刊的发表需求,且已有超过4000篇文章引用。
视频解读
Bilibili:https://www.bilibili.com/video/BV1Xf421v7zV/
Youtube:https://youtu.be/mD2MgR5X7Pc
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
全文解读
引 言
随着高通量测序技术的快速发展,生物数据呈指数级增长,如何高效分析大数据和挖掘潜在的生物信息对生物学家来说是一个巨大的挑。一般来说,从获得基础数据到最终发表研究论文,通常需要数据统计、绘制统计图表和图形美化三个步骤。为此,许多生物信息学平台工具,如Majorbio Cloud,SangerBox,ImageGP,Evenn,TBtools,complexHeatmap,ggVennDiagram等,逐渐涌现出来。而这些平台大多是为特定的组学研究开发的,特别是宏基因组和转录组,而一些工具是为了特殊问题提供了专门解决方案。但这些平台或工具的默认图形输出通常需要使用图形编辑软件(如Adobe Illustrator或Photoshop)进行大量调整,以达到适合出版标准。
此外,大多生物信息数据的分析与可视化工具都缺少用户友好的界面,无法通过“点点鼠标”就可以完成数据分析与图表绘制。为了让即使没有编程基础的生物学研究者也能轻松进行数据分析和可视化,并一步完成图形美化,我们开发了OmicShare tools(通过https://www.omicshare.com/tools/ 进行访问)。OmicShare tools是一个零代码的交互式数据分析和可视化平台,能够帮助科研人员灵活地进行数据个性化分析。
结 果
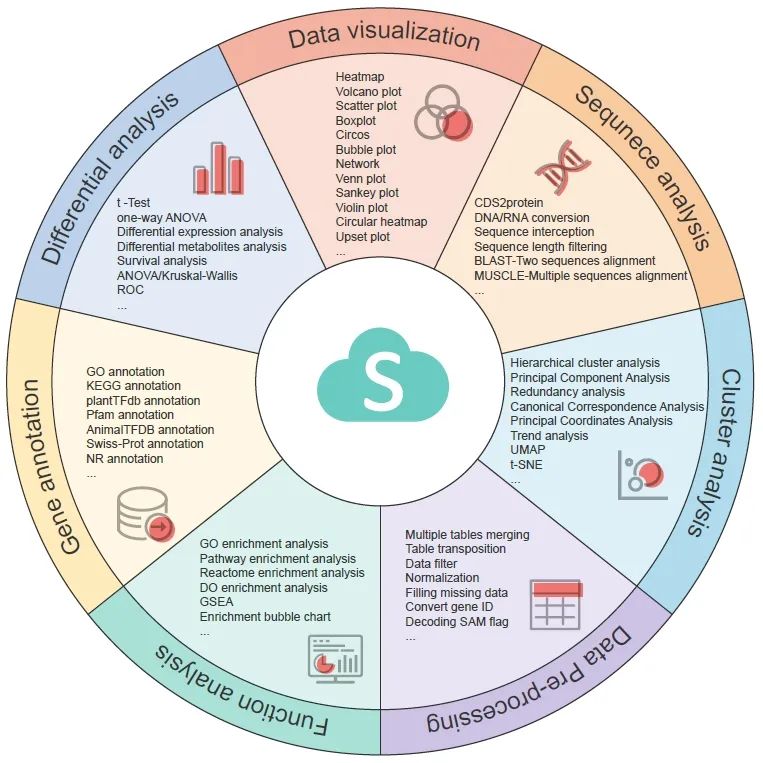
OmicShare tools数量多且功能丰富
OmicShare tools目前包含161款生物信息分析工具。这些工具的功能包括核酸和蛋白序列分析、基因功能注释、差异检验分析、聚类分析、个性化图表绘制等,涵盖了常见的组学数据分析与可视化方法(Figure 1 A)。例如,使用OmicShare tools可以进行基于 GO(Gene Ontology)、KEGG(Kyoto Encyclopedia of Genes and Genomes)、DO(Disease Ontology)、Reactome的基因功能富集分析,也可以进行常规的统计检验,如相关性分析、方差分析、主成分分析、生存分析等,还可以进行发表级图表的绘制。
在这161款工具中,具有数据可视化功能的有129个,除了常见科研图表的绘制,还可以绘制更加新颖、更加个性化的科研图表。比如,我们可以使用OmicShare tools绘制GO富集分析分组柱状图、相关性网络热图、GSEA组合图、添加置信区间的PCA散点图、多比较组散点图、山峦图等(Figure 1 B-K)。

图1. OmicShare tools的功能概述和输出的案例图形。
(A) OmicShare tools的分类,(B) GO富集条形图,(C)GSEA,(D)网络热图,(E)KEGG富集气泡图,(F)山峦图,(G) PCA 散点图,(H) 富集圈图,(I) 多组差异散点图,(J) 韦恩图,(K) 差异富集气泡图。
另外,一些综合性OmicShare tools的结果中的图表也比较丰富。以KEGG富集分析工具的结果为例,除了常见的气泡图(Figure 3 E)、条形图(Figure 3 B),还有更加个性化的圈图、组合气泡图、网络图(Figure 3 H,K)等。此外,为了满足不同工具的格式要求,OmicShare tools还提供了很多数据格式转换工具。比如,数据归一化/标准化工具可用于数据的标准化处理,基因ID转换工具可用于常见物种的不同基因ID类型之间的相互转换,表格格式转换工具可实现“长数据”和“宽数据”表格的相互转换等。
OmicShare tools具有强大的交互式图形引擎
OmicShare tools的使用方法很简单。用户只需根据分析目的,选择具有相应功能的小工具,然后通过图形操作界面上传数据文件,设置分析参数,即可提交分析任务(Figure 2 A)。整个分析过程中,最重要的是如何整理符合格式要求的数据文件。
OmicShare tools每个工具都包含对应的示例文件。用户可以直接下载示例文件进行工具测试或者参考示例文件的格式进行数据整理。对于参数的设置方法,可参考工具对应的说明文档(Figure 2 B),像大多数的生物信息分析工具一样,一般只需保持默认的参数选项即可得到很好的结果。
OmicShare tools具有完善的任务状态监测和报错机制。提交分析任务后,用户可在项目总览页面实时查看任务进度(Figure 2 C)。任务完成后,可以直接预览分析结果或者下载分析结果文件。如果任务出错,也可以查看出错原因。任务出错时,除了查验工具页面的说明文档,用户遇到问题也可以联系在线客服。此外,提交任务时,工具会自动对上传的数据文件进行格式检查,如果文件格式不符合要求,工具无法成功提交任务并提示出错原因。OmicShare tools具有很高的兼容性,支持多种类型的文件格式,比如FASTA格式的序列文件、制表符或逗号分隔的文本文件、Excel格式的二进制文件等。

图2. OmicShare tools的主要图形界面
(A) 数据文件提交界面,(B) 工具说明与案例,(C) 任务进程,(D) 图形绘制设置窗口。
OmicShare tools支持高质量图表生成
OmicShare tools具有强大的交互式图形引擎。为了满足用户实时地对图表样式进行调整,我们自主研发了基于JavaScript的交互式的图形绘制引擎。首先,OmicShare tools的图形引擎具有丰富的图形设置参数,支持用户在线实时调整图表的图形元素,例如调整图表的坐标轴、字体样式、主题、配色等(Figure 2 D)。其次,OmicShare tools内置了丰富的配色方案供用户直接选用,包含23种渐变颜色(Gradient colors)和15种分类色板(color palettes),如果不满意,用户甚至还可以自定义配色方案。以动态网络图韦恩图工具为例,通过图形参数选项窗口,可以直接调整网络节点的形状、大小、颜色、透明度、标签样式等,也可以调整网络边的线条配色、粗细、曲率、样式等。
图形引擎中的设置参数可以进行保存。之后重新打开工具界面或者切换之前任务,可快速重现之前调整后的效果。一般来说,OmicShare tools默认的图表样式可满足用户的投稿需求,用户无需付出太多时间成本就可获得自己想要的效果。
OmicShare tools导出的图片可直接用于文章发表。我们对OmicShare tools的图表样式和配色进行了精心设计,足以满足科学期刊的发表需求。另外,用户也可以对图表中的图形元素进行自定义调整,对默认的图表样式进行调整。OmicShare除了支持导出JPG、PNG、BMP、TIF格式的位图,也支持导出PDF、SVG格式的矢量图。此外,用户还可以使用Adobe illustrator、Inkscape、CorelDRAW等软件对矢量图进行个性化调整。
方 法
OmicShare tools作为一个在线工具网站,使用Javascript、HTML和boostrap进行前端开发,而高级web框架ThinkPHP则用于后端的数据预处理和统计分析。一部分工具是基于R和Python编程语言进行分析和绘图的,包括ggplot2、complexHeatmap、edgeR、DESeq2等辅助包;其他工具则是根据用户数据,完全使用自研前端绘图插件进行绘图的。
讨 论
OmicShare tools是一个零代码、交互式、用户友好的在线数据分析与可视化工具。OmicShare tools支持导出多种可用于出版的图形文件格式。上述特点不仅降低了用户的软件操作的学习成本,而且输出结果可以直接用于论文发表。很多优秀的科研工作者使用OmicShare tools 进行数据分析与图表绘制,例如,热图的绘制、GO富集分析与KEGG富集分析、主成分分析(PCA)、韦恩图的绘等。OmicShare tools于2016年4月发布后引用SCI论文数超过4000篇。
OmicShare tools目前仍在不断地进行优化和更新。虽然OmicShare拥有强大的交互式图形引擎,但一些工具的图形参数并不全面,缺乏详细的分类,仍需进一步优化和调整。OmicShare tools未来的版本可能会包含更多的数据类型和分析方法,例如单细胞测序数据、临床医学数据等。OmicShare tools也将持续对工具参数选项和交互设计进行优化,使得更多非生信专业的用户也能轻松进行数据挖掘。
引文格式:
Mu, Hongyan, Jianzhou, Chen, Wenjie Huang, Gui Huang, Meiying Deng, Shimiao Hong, Peng Ai, Chuan Gao, and Huangkai Zhou. 2024. “OmicShare tools: a Zero‐Code Interactive Online Platform for Biological Data Analysis and Visualization.” iMeta e228. https://doi.org/10.1002/imt2.228
作者简介

母红岩(第一作者)
● 华中农业大学植物病理学硕士,现为广州基迪奥生物科技有限公司运营组成员。
● 目前主要负责组学生信资料整理与撰写,以及OmicShare tools云工具的开发、优化工作。

周煌凯(通讯作者)
● 博士,“广东省科技咨询专家库专家”,广州基迪奥生物科技有限公司技术总监。
● 从事基因测序服务十余年,在基因组学、单细胞组学及转录组学数据挖掘方面有相当丰富的经验。已在Nature Cell Biology、Nature Communications、PNAS、Cell Metabolism、Genome Biology、iMeta等期刊发表学术论文28篇,主持广东省科技计划项目、广州市科技局重点部署项目子课题等,相关成果获得发明专利21项,30余项软件著作权。

高川(通讯作者)
● “广州市产业发展和创新人才”,广州基迪奥生物科技有限公司总经理。
● 在大数据领域深耕10余年,主持参与广东省、广州市政府科技项目等,相关成果获得23项发明专利、30余项软件著作权。

艾鹏(通讯作者)
● 广州基迪奥生物科技有限公司生信分析部总监。
● 从事生物信息数据挖掘十余年,擅长组学数据分析流程、定制化化分析流程的开发与优化。主持参与多项广州市政府科技项目,相关成果获得6项发明专利、20余项软件著作权。
更多推荐
(▼ 点击跳转)
iMeta | 引用13000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
3卷1期
2卷2期封底
2卷4期封底
3卷2期
3卷3期
3卷3期封底
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百千华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表所有领域高影响力的研究、方法和综述,重点关注微生物组、生物信息、大数据和多组学等。目标是发表前10%(IF > 20)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、前3年免出版费、50万用户的社交媒体宣传等。2022年2月正式创刊发行!发行后相继被Google Scholar、ESCI、PubMed、DOAJ、Scopus等数据库收录!2024年6月获得首个影响因子23.7,位列全球SCI期刊前千分之五(107/21848),微生物学科2/161,仅低于Nature Reviews,同学科研究类期刊全球第一,中国大陆11/514!
“iMetaOmics” 是“iMeta” 子刊,主编由中国科学院北京生命科学研究院赵方庆研究员和香港中文大学于君教授担任,是定位IF>10的高水平综合期刊,欢迎投稿!
联系我们
iMeta主页:
http://www.imeta.science
姊妹刊iMetaOmics主页:
http://www.imeta.science/imetaomics/
出版社iMeta主页:
https://onlinelibrary.wiley.com/journal/2770596x
出版社iMetaOmics主页:
https://onlinelibrary.wiley.com/journal/29969514
iMeta投稿:
https://wiley.atyponrex.com/journal/IMT2
iMetaOmics投稿:
https://wiley.atyponrex.com/journal/IMO2
邮箱:
office@imeta.science







































 2747
2747

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








