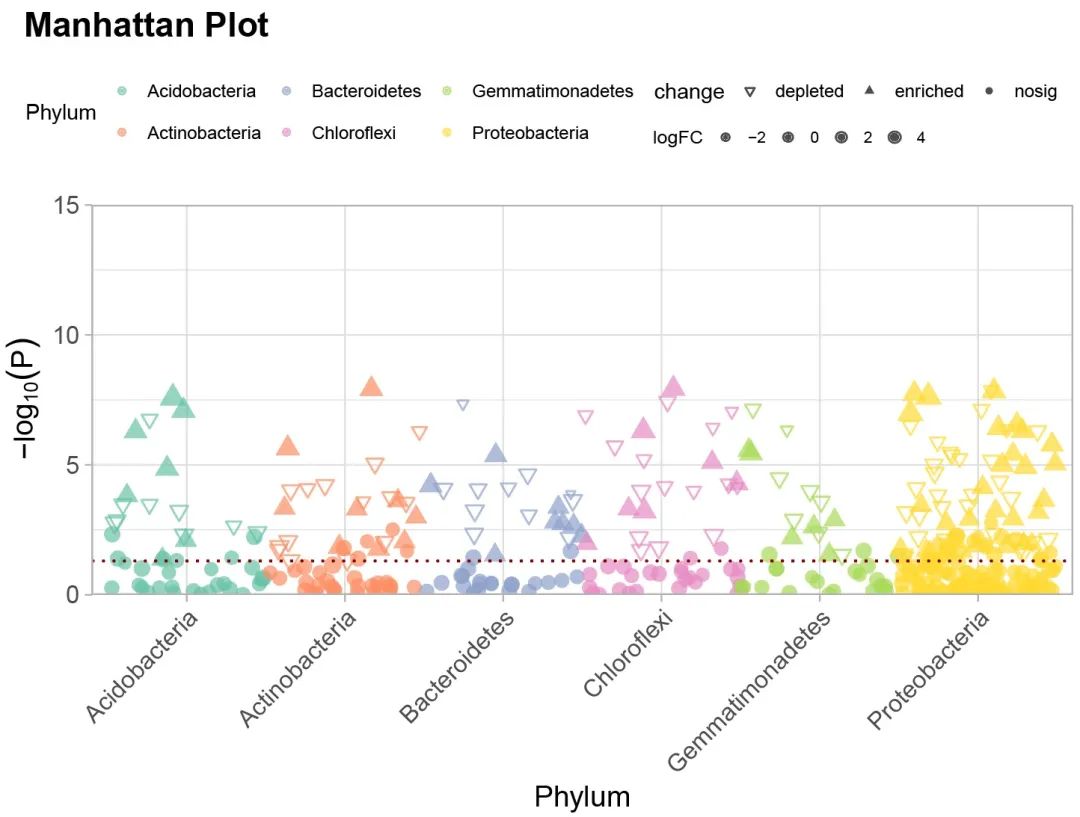
曼哈顿图简介
曼哈顿图(Manhattan Plot)是一种散点图,通常用于显示具有大量非零的波动数据点,以及更高幅度值的分布,常常用在全基因组关联研究(GWAS)中。它的图像因与曼哈顿的天际线相似而得名。在微生物分析中,曼哈顿图可以用来展示差异OTU/ASV的上下调和差异OTU/ASV归属情况。曼哈顿图在其他研究中的应用包括:1.展示全基因组数据,直观的展示整个基因组中的统计显著性,通常以染色体为单位来展示数据点; 2.识别显著性区域,通过颜色或高度的变化,可以快速识别出具有统计显著性的区域,这些区域可能与某些疾病或性状相关;3.比较不同研究,可以用来比较不同研究或不同群体之间的统计显著性差异;3.可视化P值分布,使用负对数尺度(-log10)来表示P值,这使得观察者可以很容易识别出P值的分布情况;4.软色体位置的可视化,通常沿着X轴展示染色体的位置,使得观察者可以直观地看到每个显著性信号在基因组中的位置;5.辅助假设生成,通过观察曼哈顿上的模式,研究人员可以生成新的科学假设,比如那些基因或区域可能与研究的性状有关联。
标签:#微生物组数据分析 #MicrobiomeStatPlot #曼哈顿图 #R语言可视化
作者:First draft(初稿):Defeng Bai(白德凤);Proofreading(校对):Ma Chuang(马闯) and Jiani Xun(荀佳妮);Text tutorial(文字教程):Defeng Bai(白德凤)
源代码及测试数据链接:
https://github.com/YongxinLiu/MicrobiomeStatPlot/ 项目中目录 3.Visualization_and_interpretation/ManhattanPlot
或公众号后台回复“MicrobiomeStatPlot”领取
曼哈顿图案例1
这是中国科学院遗传发育所白洋课题组2019年发表于Nature Biotechnology上的一篇论文用到的曼哈顿图。论文题目为:NRT1.1B is associated with root microbiota composition and nitrogen use in field-grown rice. https://doi.org/10.1038/s41587-019-0104-4.
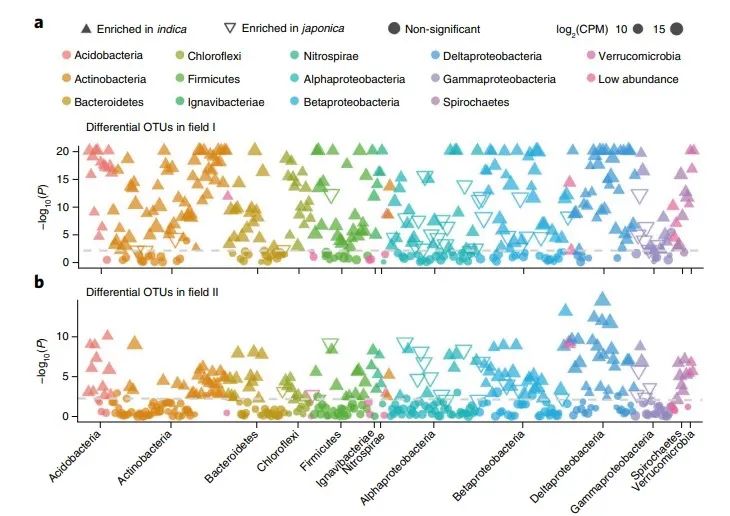
图 3 | 籼稻和粳稻根部微生物群差异细菌的分类和功能特征
曼哈顿图显示田块 I (a) 和田块 II (b) 中富含籼稻或粳稻的 OTU。每个点或三角形代表一个 OTU。富含籼稻或粳稻的 OTU 分别用实心或空心三角形表示(FDR 调整 P<0.05,Wilcoxon 秩和检验)。OTU 按分类顺序排列,并根据门或变形菌纲着色。CPM,每百万计数。
总结:该图利用曼哈顿图展示了在籼稻或粳稻中富集的OTU。
结果
根系微生物组和氮利用。接下来我们在OTU水平上研究了籼稻和粳稻亚种根系微生物组的差异。首先,我们使用曼哈顿图分析了OTU的富集情况(图3a、b和补充表6)。在两个田块中,籼稻中富集的OTU属于广泛的细菌门,包括酸杆菌门、变形菌门、放线菌门、拟杆菌门、绿弯菌门、厚壁菌门和疣微菌门(FDR调整后的P<0.05,Wilcoxon秩和检验;图3a、b和补充表6)。粳稻根系保留了富集属于变形菌门、拟杆菌门和厚壁菌门的OTU的能力(FDR调整后的P<0.05,Wilcoxon秩和检验;图3a、b和补充表6)
正文描述:通过曼哈顿图展示了OTU水平上籼稻和粳稻亚种根系微生物组的差异。使用曼哈顿图分析了OTU的富集情况。
曼哈顿图案例2
这是约翰霍普金斯大学彭博公共卫生学院Nilanjan Chatterjee团队2022年发表于Nature Genetics上的一篇论文。论文的题目是:Plasma proteome analyses in individuals of European and African ancestry identify cis-pQTLs and models for proteome-wide association studies. https://doi.org/10.1038/s41588-022-01051-w.
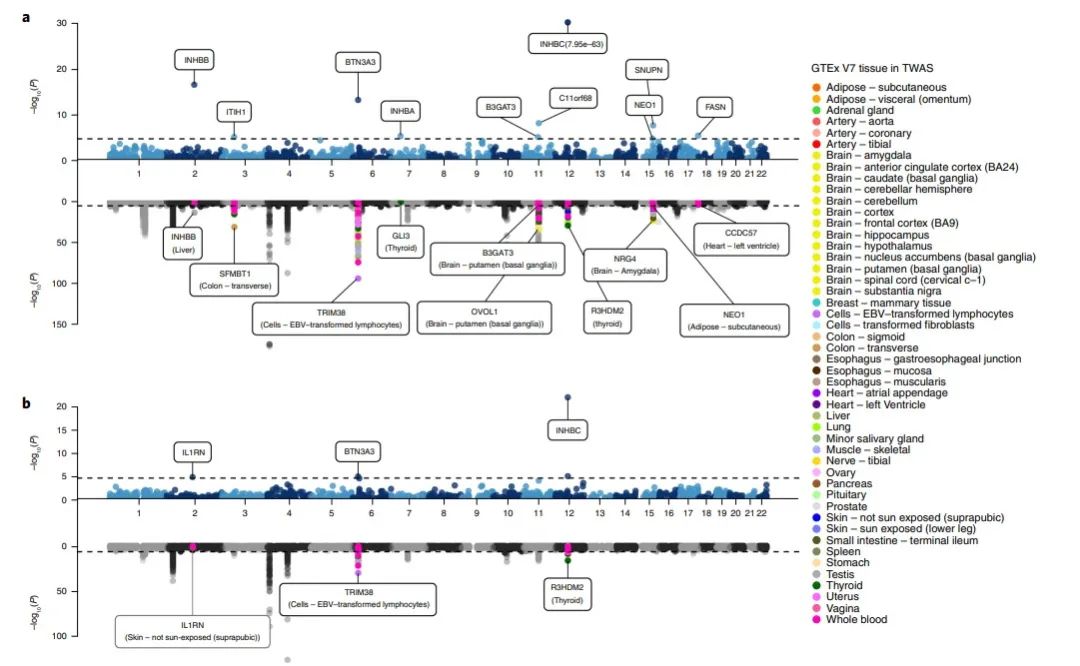
图 4 | 血清尿酸水平和痛风的 PWAS 和 TWAS 分析的迈阿密图
a、b 尿酸 (a) 和痛风 (b) 的 PWAS (上) 和 TWAS (下) 的迈阿密图。每个点代表表型与顺式遗传调节的血浆蛋白或基因表达水平之间关联的双侧 z 检验的 P 值,按 x 轴上的基因组位置和 y 轴上的关联强度的 −log10(P 值) 排序。黑色水平虚线是 Bonferroni 校正后对总插补模型数量的显着性阈值(P 值 = 3.7 × 10−5(PWAS)和 TWAS 的 2.1 × 10−7)。为了更好地显示,a 中的尿酸 PWAS 和 TWAS 在 y 轴上以 −log10(P 值) = 30 和 −log10(P 值) = 150 截断。重要 PWAS 基因附近的 TWAS 基因(±500 kb)按 GTEx 组织着色。最显著的附近 TWAS 基因标有其基因名称和相应的组织。IL1RN 的 TWAS 未达到 TWAS 显著性阈值,因此标记为灰色。所有主要 TWAS 分析均基于使用 GTEx V7 数据开发的既定模型进行,并使用 GTEx V8 提供的初步模型进一步验证已确定的顶级基因/组织组合的结果(补充表 16)。为了减小图形的








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 1291
1291

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








