点击蓝字 关注我们
产气荚膜梭菌对人类肠道的基因组适应性

iMeta主页:http://www.imeta.science
研究论文
● 原文链接DOI: https://doi.org/10.1002/imo2.38
●2024年10月25日,四川大学王红宁课题组与多家单位合作在iMetaOmics在线发表了题为“Genomic adaptation of Clostridium perfringens to human intestine”的文章。
● 本研究使用大规模配基因组数据,系统分析了不同地区、宿主来源的产气荚膜梭菌遗传进化关系、食源致病基因cpe基因背景变化和产气荚膜梭菌基因组的宿主适应性。
● 第一作者:吴克、王娟
● 通讯作者:王红宁(whongning@163.com)、张嵘(zhang-rong@zju.edu.cn)、殷喆(jerry9yin@163.com)
● 合作作者:严泽琳、竺艳艳、王少林、付博、孙城涛、李瑞超、Edward M Fox、Séamus Fanning、白莉、汪洋、唐艺芝
● 主要单位:四川大学生命科学学院、西北农林科技大学动物医学院、国家食品安全风险评估中心、浙江大学医学院附属第二医院、中国农业大学动物医学院、扬州大学比较医学研究院、军事科学院军事医学研究院
亮 点

● 本研究对中国人源产气荚膜梭菌基因组进行了研究;
● 在个体之间存在产气荚膜梭菌的区域和跨区域传播;
● cpe基因首次在pCW3样和pCP13样质粒融合质粒上发现;
● 产气荚膜梭菌的基因组特征和其宿主物种相关联。
摘 要
产气荚膜梭菌(Clostridium perfringens)与人类的食源性感染紧密相关,可导致显著的公共卫生安全危害。然而目前缺少人群中产气荚膜梭菌基因组学研究;因此本研究对中国某省立医院699个个体中分离的产气荚膜梭菌菌株进行了基因组学分析。并在此基础上进一步对不同地区人和动物源的产气荚膜梭菌菌株进行了进化关系和泛基因组分析。结果表明不同地区的人类间存在产气荚膜梭菌的潜在传播,并且在此过程中伴随着普遍的小基因片段转移。值得注意的是,与食物中毒密切相关的毒素基因cpe首次在一个融合质粒上鉴定出来,揭示了产气荚膜梭菌中pCW3样和pCP13样质粒的融合以及cpe基因背景的改变。此外,本研究还观察到产气荚膜梭菌的基因组特征与宿主物种相关;某些毒素基因、如 pfoA 和 colA、可能影响产气荚膜梭菌的宿主选择性。通过这项全面的基因组学分析,我们提供了关于 pCW3 样和 pCP13样质粒的融合、cpe 的遗传背景、产气荚膜梭菌的传播以及毒素基因与产气荚膜梭菌宿主相关性之间联系的新见解。有助于拓展我们对产气荚膜梭菌的理解。
视频解读
Bilibili:https://www.bilibili.com/video/BV1F9m4YBEpD/
Youtube:https://youtu.be/AQUj3IciN30
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/imetaomics/
全文解读
引 言
产气荚膜梭菌是引起人和动物肠道感染的主要食源性病原体之一。产气荚膜梭菌的致病性主要源自其产生的蛋白毒素和酶。目前研究人员已从产气荚膜梭菌中鉴定出超过20种毒素;这些毒素可导致人和动物出现从组织毒性感染(如气性坏疽)到肠道感染(如肠炎)等多种疾病。根据其对α-毒素(plc编码)、β-毒素(cpb编码)、ε-毒素(etx编码)、ι-毒素(iap/ibp编码)、产气荚膜梭菌肠毒素(cpe编码)和坏死性肠炎B毒素(netB编码)这六种主要毒素的产生能力,产气荚膜梭菌被分为A-G七种毒素型。产气荚膜梭菌可广泛分布于环境和食物中;其耐热性芽孢可以在未完全烹饪的食物中增殖进而导致潜在感染。机体摄入相应食物后,病原菌株迅速增殖并分泌大量蛋白毒素,导致肠道感染和食物中毒。产气荚膜梭菌被认为与人类的抗生素相关腹泻和坏死性小肠结肠炎等疾病紧密相关,在全球范围广泛引起食源性疾病、对公共卫生安全构成了重大威胁。
质粒是梭状芽孢杆菌发挥致病性的关键因素。根据共轭位点,产气荚膜梭菌中的质粒可分为三个大类:共轭的 pCW3样质粒(Tcp 位点)和pCP3样质粒(Pcp 位点)以及非共轭的 pIP404样质粒。除了plc 外,所有的分型毒素基因均可由pCW3样质粒携带;并且一些耐药基因如 tetA/B(P)和erm(Q)也与 pCW3 样质粒相关。携带cpe的 F 型产气荚膜梭菌被认为是食源性感染的主要原因;F 型菌株通过在宿主的肠道中形成芽孢并产生CPE,进而导致食物中毒。最近的研究报告显示,由 pCP13 类质粒上的becA/B编码的二元肠毒素 BEC也与产气荚膜梭菌食源性感染相关。高通量测序技术已广泛应用于流行病学研究,以促进对病原体潜在传播的分析。作为一种人畜共患的食源性病原体,理解产气荚膜梭菌的基因组特征对公共健康至关重要。尽管一些研究已调查中国来自羊和猪的产气荚膜梭菌基因组特征,但尚无针对中国人源产气荚膜梭菌种群系统的基因组学研究。在本研究中,我们对来自中国人类肠道的产气荚膜梭菌分离株进行了全面的基因组分析;鉴定了菌株中包括耐药基因、毒素基因和质粒等遗传标记,并追踪了菌株在个体之间的潜在传播。此外,本研究还对全球人类和动物分离株基因组进行了比较研究以深入了解产气荚膜梭菌的种群结构和宿主相关性。
结 果
产气荚膜梭菌菌株的遗传标记
本研究对195个通过基因组质量检测的产气荚膜梭菌基因组进行了分析。这些菌株间的平均核苷酸一致性范围为95%到100%之间(图 1A)。大多数的分离株被鉴定为A型产气荚膜梭菌,有四株产气荚膜梭菌(CQ145、CQ147、CQ165 和 CQ169)被鉴定为 F 型产气荚膜梭菌。
在这些分离株中,鉴定出了四类抗生素耐药基因(图 1B),包括四环素耐药基因(tetA(P)、tetB(P) 和 tet(44))、大环内酯耐药基因(erm(B)、erm(G)、erm(Q) 和 erm(T))、氨基糖苷耐药基因(ant(6)-Ia、ant(6)-Ib 和 aac(6')-aph(2'')以及林可霉素耐药基因lnu(P)。此外,菌株中还鉴定出了包括 cpe 和becA/B 在内的16个毒素编码基因(图 1C)。Tcp 和 Pcp 基因位点的检出情况见表 S2。质粒 pCW3、pCP13 和本研究产气荚膜梭菌分离株之间的全基因组对比可见表 S3。结果显示,pCP13样质粒在20%(39/195)的分离株中发现,而pCW3 样质粒在分离株中更为普遍(141/195,72.3%,p < 0.01)(图 1D)。主要的耐药基因 [tetA(P)、tetB(P)、erm(Q) 和 lnu(P)] 在携带不同质粒组合的产气荚膜梭菌分离株中均被检测到(图 1E)。此外本研究分离株不携带新型的 pCPCP 样质粒(图 1F)。
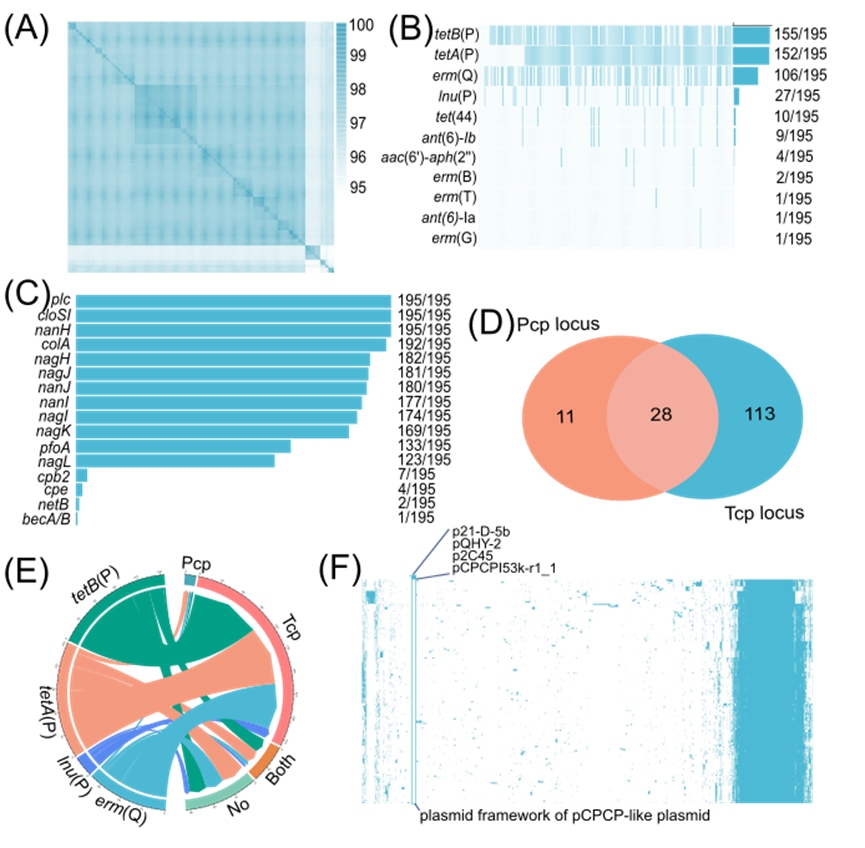
图1. 本研究195株产气荚膜梭菌分离株基因组信息
(A)分离株之间的平均核苷酸一致性;(B)产气荚膜梭菌分离株的耐药基因;(C)产气荚膜梭菌分离株中毒素基因的流行情况;(D)分离株中 Pcp 和 Tcp 基因位点的鉴定;(E)耐药基因与 Tcp/Pcp 基因位点之间的联系;(F)在分离株中检测新型 pCPCP 类似质粒的质粒框架,pCPCP 类似质粒的质粒框架用方框标出。
产气荚膜梭菌的传播
致病菌在生产链和人群中的传播是导致食源性疾病爆发的重要原因。平均核苷酸一致性分析显示八个分离株(CQ161&CQ162、CQ202&CQ231、CQ147&CQ165、CQ166&CQ167、CQ145&CQ169、CQ13&CQ217、CQ163&CQ242、CQ44&CQ56、CQ181&CQ258)之间的平均核苷酸一致性超过99.99%。单核苷酸多态性和全基因组分析表明这些分离株之间的SNP差异范围为2到89之间,并且具有相似的基因组组成(图2A),这表明这些菌株之间可能存在传播事件。对SNP差异很小的产气荚膜梭菌菌株CQ145和CQ169的比较基因组分析发现,CQ145的基因组中存在数个额外的基因,这些基因位于两个基因簇中,分别称为片段1和片段2。
基因注释表明了片段1上的基因可编码多个功能蛋白,包括TnpY和MocB。TnpY和MocB分别是转座子Tn4451/Tn4453和Tn4399的组成部分。Tn4451和Tn4453与产气荚膜梭菌和艰难梭菌中的耐药基因转移相关联。最近的研究表明,Tn4451/Tn4453和TnAs3在来自人类和食品动物源的细菌之间的耐药基因转移中发挥作用。此外,BLAST分析显示在其他肠球菌等多种细菌物种中也有类似片段,这表明片段1可能在不同细菌属之间进行水平转移。与片段1相比,片段2包含若干编码与温和噬菌体相关的噬菌体蛋白的基因(图2B)。这些发现表明,产气荚膜梭菌菌株在传播过程中可以获得外源基因簇,这可能增强它们在随后的传播过程中整合和传播新基因的能力(图2C)。
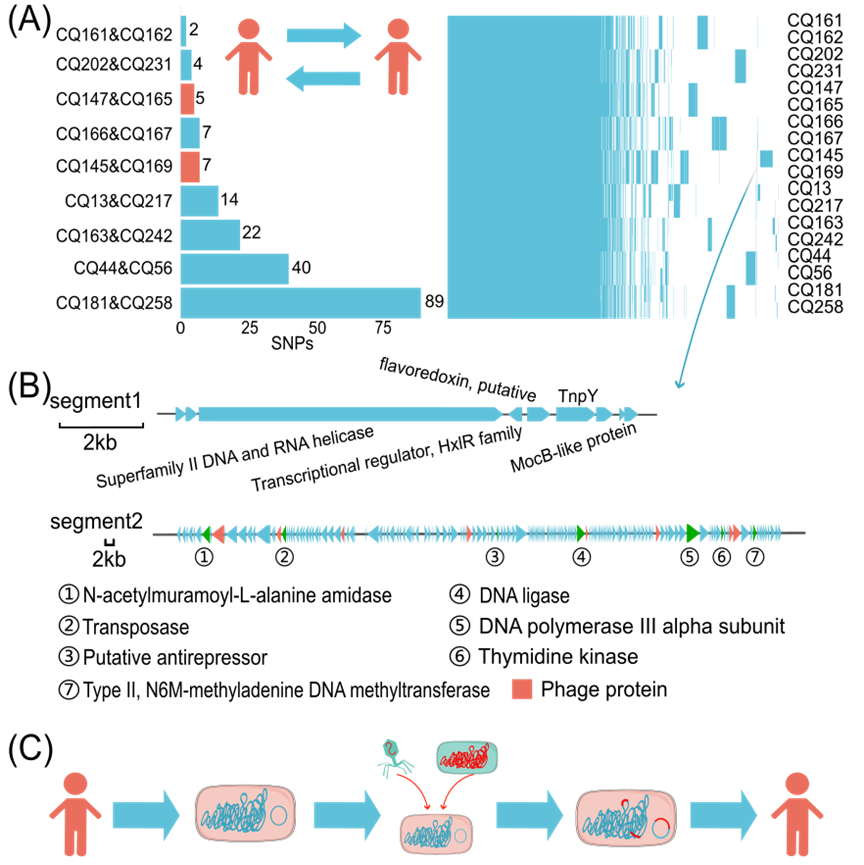
图2. 产气荚膜梭菌在人群中的潜在传播
(A) 分离菌株的单核苷酸多态性和泛基因组对比;(B)产气荚膜梭菌分离株CQ145中的额外基因片段;(C) 产气荚膜梭菌菌株潜在传播的示意图。
基因cpe和becA/B的基因环境
了解与临床疾病相关毒力因子的遗传特征对控制食源性疾病至关重要。在分离株CQ147和CQ167中发现的cpe基因位于包含tcp位点的一个contig上,并与pCW3-like质粒pZ1323HCP0095-2(NZ_CP148594)具有高序列同源性(图3A)。相比之下,在菌株CQ145或CQ169的cpe阳性contig的中未发现tcp位点。序列注释显示,cpe基因在CQ169中与dcm基因相邻,被插入序列△IS1469和IS1151环绕。有趣的是,在菌株CQ145中的cpe阳性contig中发现了Pcp位点(图3B)。序列分析表明,该contig由pCP13-like质粒unnamed3(CP134265.1)的一部分和来自CQ169的cpe阳性contig的部分序列(以△IS1469为起始)组成(图3C)。
为排除基因组组装过程中可能发生的错误,我们使用针对CQ145 cpe阳性contig上交接处的引物进行了PCR验证。结果与Illumina测序的WGS数据一致(图S1)。然而,Illumina测序的WGS分析并未组装到CQ145中cpe阳性质粒的完整序列。为进一步研究,我们使用PacBio平台对CQ145进行了WGS测序。结果(BioProject: PRJNA1118504)显示,Pcp和Tcp位点与cpe基因一起存在于质粒pcq145-tcppcp上,提示CQ145中pCW3样质粒和pCP13样质粒的融合。随后在NCBI上对pcq145-tcppcp的BLAST分析显示其与pCW3样质粒pZ1323HCP0095-2(CP148594.1)和pCP13样质粒pZ1323HCP0095-3(CP148595.1)具有高序列同源性。序列比较表明,pcq145-tcppcp是由pCW3样质粒插入到pCP13样质粒上胶原粘附素编码基因cna中的结果。并且插入序列△IS1469和△ISCpe2位于pCW3样质粒和pCP13样质粒之间的交界处(图3D和图S2),这表明这些元件在pcq145-tcppcp的形成中起了重要作用。在CQ92菌株中发现的becA/B阳性contig也包含Pcp位点,并与之前鉴定的pCP13-like质粒pCP-TS1(AP024974.1)具有高度序列相似性(图S3)。
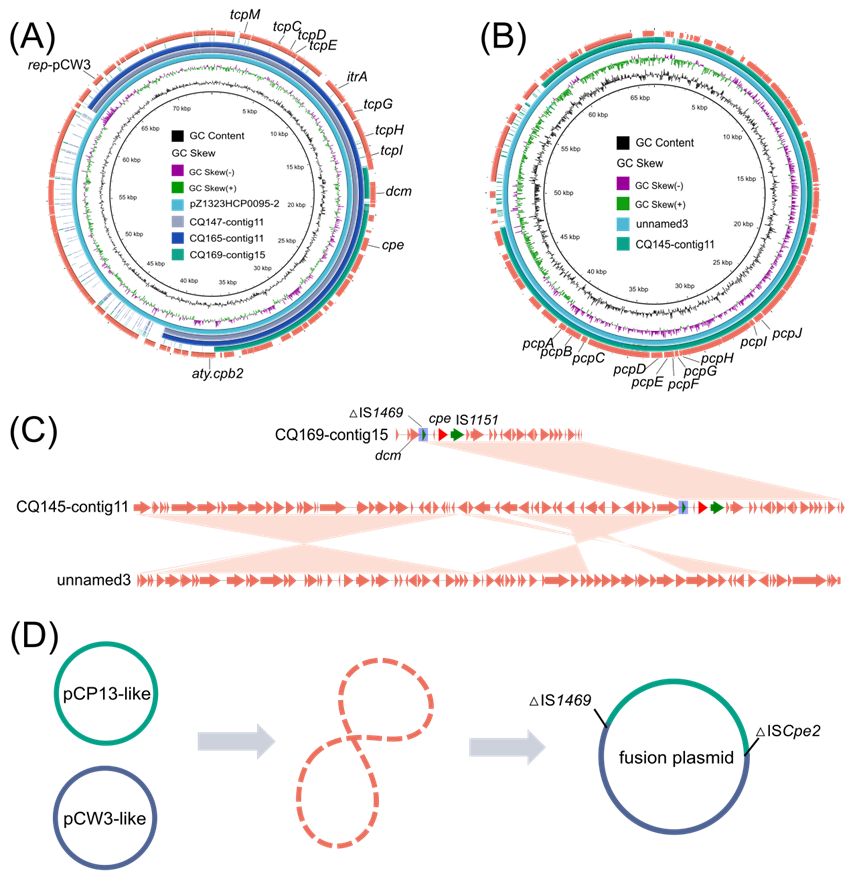
图3. F型菌株CQ145、CQ147、CQ165和CQ169中cpe基因的遗传环境
(A)pCW3-like质粒pZ1323HCP0095-2与CQ147、CQ165和CQ169中cpe阳性contig的序列对比;(B)pCP13-like质粒unnamed3与CQ145 cpe阳性contig的圈形对比;(C)CQ145中cpe阳性contig组成;(D)融合质粒形成示意图。
中国产气荚膜梭菌群体结构
为了调查中国人类和动物源性产气荚膜梭菌基因组之间的关系,我们基于本研究中195株人类来源的产气荚膜梭菌和NCBI中中国167株产气荚膜梭菌的162,222个核心基因组SNP进行了进化分析(表S5)。最大似然树中产气荚膜梭菌被分为五个进化分支,分别标记为Cluster 1到Cluster 5(图4A)。人类来源的菌株聚集在Cluster 2和Cluster 3,而动物来源的菌株则聚集在Cluster 1和Cluster 2。在Cluster 1和Cluster 4中出现较少的人类来源菌株。在Cluster 5中观察到11个人类来源的菌株与一些来自鸡和牛的菌株聚集在一起。
一些重要的临床耐药基因如optrA、cfr(B)和cfr(C)仅在动物源菌株中检测到。对菌株耐药基因总数的比较显示,动物尤其是猪是中国产气荚膜梭菌耐药基因的主要储存宿主(图4B)。此外,从绵羊、山羊和牛分离到的菌株中携带的毒素基因比人源菌株更多(图4C),这提示了不同动物来源的菌株在毒力上可能存在潜在差异。
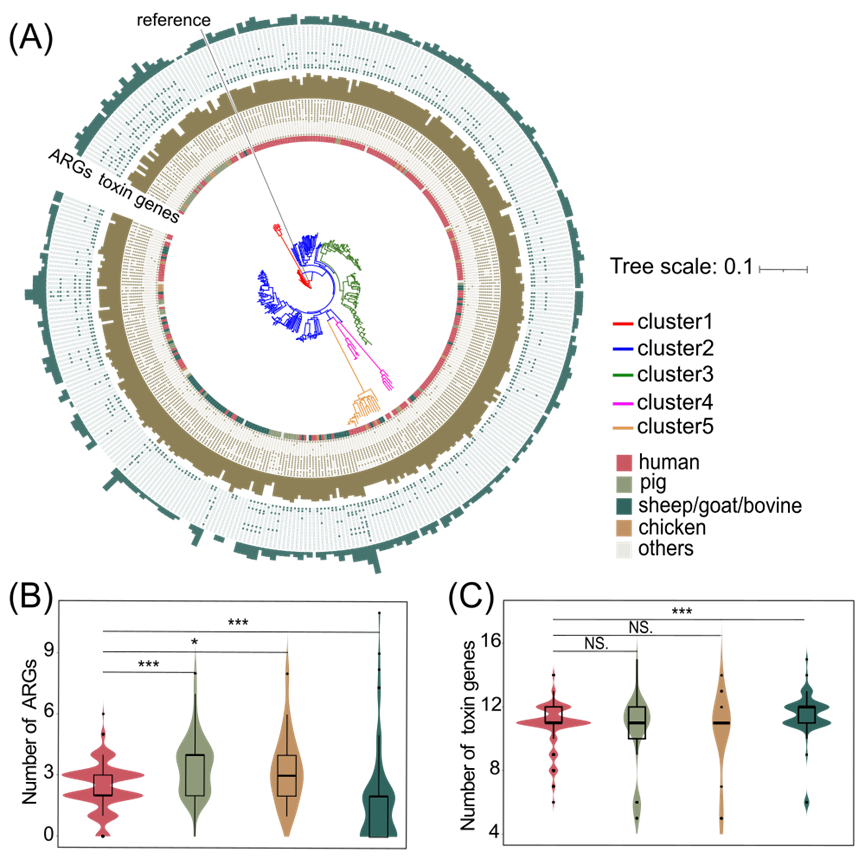
图4. 中国产气荚膜梭菌菌株的系统发育分析
(A)本研究中195株产气荚膜梭菌分离株与中国其他167株产气荚膜梭菌的最大似然树;(B)人类和动物分离株的耐药基因总数对比;(C)人类和动物分离株的毒素基因总数对比。
世界人源产气荚膜梭菌进化关系
在这项研究中,我们调查了来自全球人类的423株产气荚膜梭菌菌株的基因组特征和系统发育关系,菌株详细的数据在表S6中提供。在来自全球人类产气荚膜梭菌菌株的118,750个核心基因组SNP构建的最大似然树上,来自不同国家的大多数分离株聚集在一起(图5A),并且表现出高度相似的平均核苷酸相似性(图S4A)。此外,从不同国家分离到的产气荚膜梭菌附属基因未观察到显著差异(图S4B),这表明区域来源对产气荚膜梭菌的进化关系或基因组特征没有显著影响。
在这些产气荚膜梭菌中共鉴定出19个耐药基因,其中tetA(P)(326/423, 77.1%)、tetB(P)(252/423, 57.9%)和 erm(Q)(121/423, 28.6%)是比较常见的耐药基因。然而,lnu(P)仅在8.0%(34/423)的分离株中被鉴定出来,其他耐药基因如 aac(6')-aph(2'')和blaZ仅在不到10个分离株中发现(图S5)。鉴于大多数分离株来自中国(n = 208)和美国(n = 139),我们调查了这两个地区耐药基因的分布。结果表明,来自中国的产气荚膜梭菌携带较多的耐药基因,这主要反映在tetB(P) 和erm(Q)的较高检测率上(图5B)。
基于CQ145中的附加基因簇及其与可移动遗传元件(MGEs)和噬菌体的关联,我们选择了另外55株具有超过99.99%序列同一性的人源产气荚膜梭菌菌株来进一步鉴定可移动基因簇。值得注意的是,这些相似菌株大多来自同一国家,表明产气荚膜梭菌在人类中可能的区域传播。并且我们观察到菌株CQ93与MC066_D7(澳大利亚,GCF_951337105.1)以及CQ176与4928STDY7387889(英国,GCF_902166025.1)之间存在超过99.99%的核苷酸相似性,表明这些菌株潜在的跨区域传播。此外,在具有超过99.99%核苷酸相似性的菌株之间常常鉴定出小的基因片段差异(图5C),这突显了基因簇在产气荚膜梭菌菌株传播过程中转移的普遍性。
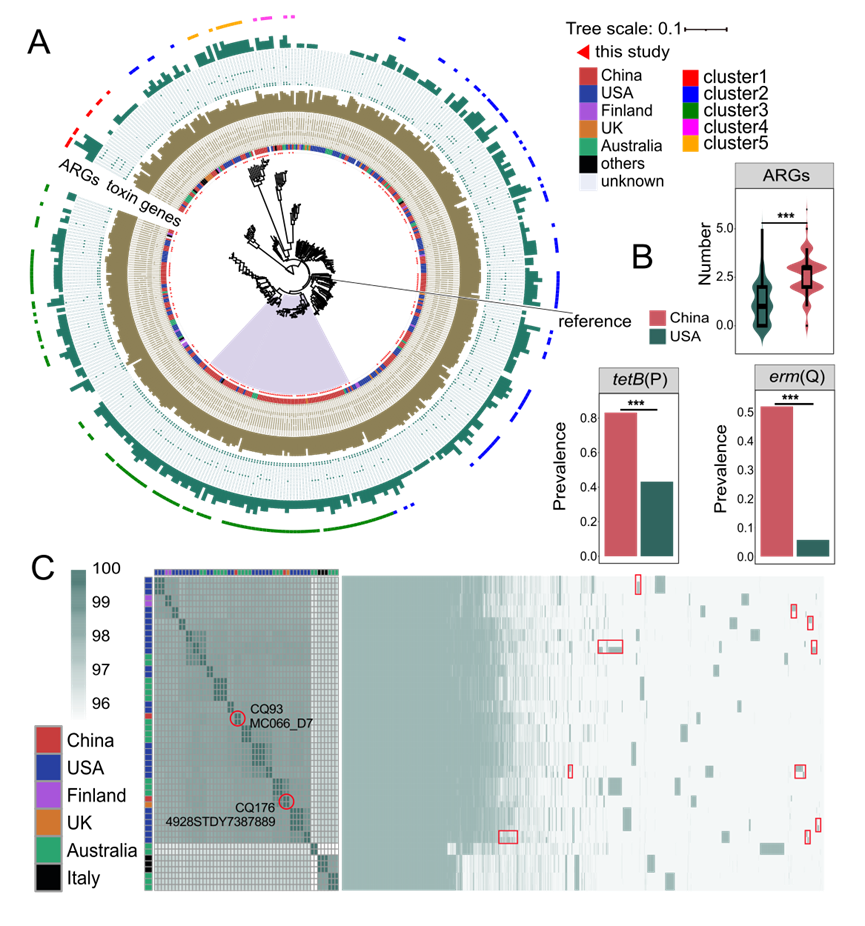
图5. 423株人源产气荚膜梭菌进化分析
(A)菌株之间最大似然树;(B)中国和美国菌株中耐药基因总数以及耐药基因erm(Q) 和tetB(P)检出率的对比;(C)具有高平均核苷酸一致性的产气荚膜梭菌菌株基因组组成,红色方框标出了微小的基因差异。
人源和动物源产气荚膜梭菌的泛基因组分析
通过对全球人源产气荚膜梭菌分离株的系统发育分析,我们发现区域来源并不影响产气荚膜梭菌的进化关系(图5A)。基于“一个健康”,我们进一步研究了涵盖来自全球人类(n = 423)、猪(n = 107)、鸡(n = 108)和羊/山羊/牛(n = 137)的产气荚膜梭菌分离株(表S7)的进化关系及附属基因相似性。如上述分析,猪是产气荚膜梭菌耐药基因的主要储存宿主(图S6)。在基于108,036个核心基因组SNP的最大似然树上,产气荚膜梭菌菌株被分为六个进化簇,标记为Ⅰ到Ⅵ(图6A)。来自中国及其他国家的人源菌株中的高比例聚集在簇Ⅲ中。不同进化簇中附属基因的非度量多维尺度和聚类分析表明,同一进化簇内的菌株共享相似的附属基因谱(图6B和图S7A)。
与来自中国的产气荚膜梭菌相比,世界来源产气荚膜梭菌中识别出了额外的一个进化簇。然而,区域来源的扩展并未改变最大似然树的基本结构,在Cluster 1到5中的中国来源菌株被发现相应分布在簇Ⅰ到Ⅵ中。这表明区域来源不是决定产气荚膜梭菌基因组特征的主要因素。类似于在中国分离的产气荚膜梭菌,全球一些人和动物来源的菌株被发现聚集在簇Ⅰ中。然而,其他相同动物来源的产气荚膜梭菌在系统发育树中聚集在一起,并且来自人类、猪和鸡的大部分菌株分别具有相似的附属基因谱(图S7B)。此外,还发现来自相同动物来源的菌株尤其是猪源菌株之间具有较高的平均核苷酸相似性(图S8)。这些结果共同表明宿主物种与产气荚膜梭菌基因组特征之间存在联系。
产气荚膜梭菌的毒力主要源自其产生的蛋白毒素和酶;为了初步了解不同动物产气荚膜梭菌之间的遗传差异,我们调查了染色体携带的毒素基因(除去在所有产气荚膜梭菌菌株中均存在的plc)的流行情况。结果表明,不同动物宿主菌株之间的cloSI、nagH和nanH的检测率没有显著差异;然而其他毒素基因的检测率因宿主种类而异(图6C)。具体而言,人源菌株中pfoA和nagI比例较低,而在猪源菌株中colA、nagK和nanI的检出率较低。此外,nagL只能在约50%的产气荚膜梭菌菌株中检测到。
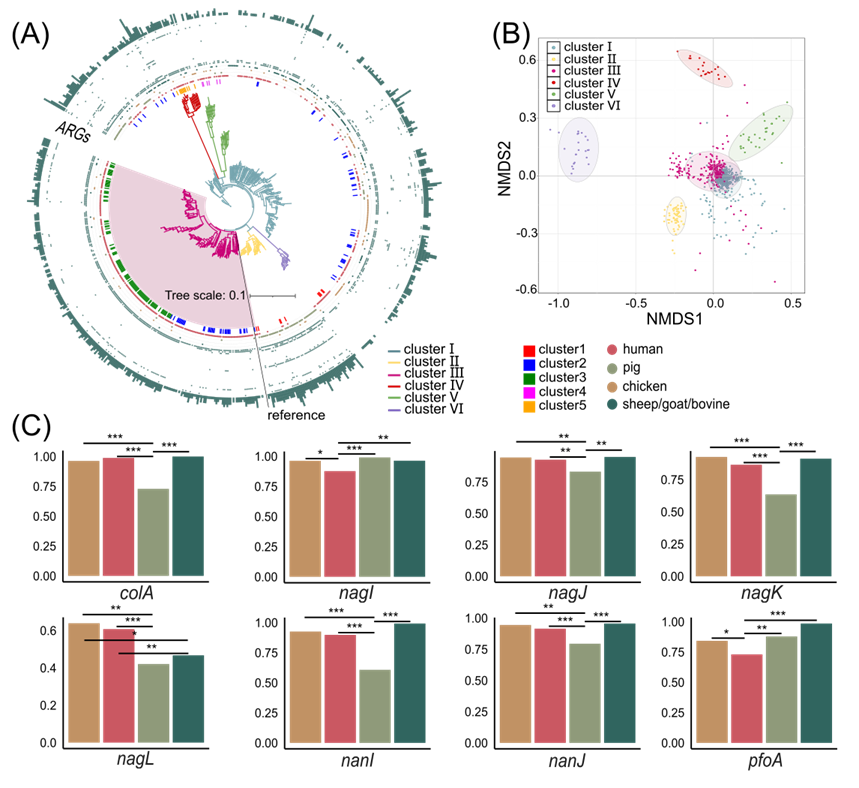
图6. 人源和动物源产气荚膜梭菌菌株的群体结构和泛基因组分析
(A) 人和动物来源的产气荚膜梭菌的进化关系;(B) 不同簇中菌株附属基因谱的非度量多维尺度分析;(C) 不同来源的产气荚膜梭菌菌株中毒素基因colA、nagI、nagJ、nagK、nagL、nanI、nanJ和pfoA的检出率。
产气荚膜梭菌是重要的食源性病原体,对公共健康构成重大威胁;包括抗生素相关性腹泻在内的多种肠道疾病均与产气荚膜梭菌感染有关。在本研究中,我们调查了来自中国的人源产气荚膜梭菌的基因组特征,并结合全球其他可用菌株进行了系统发育和泛基因组分析。研究结果显示,产气荚膜梭菌分离株的主要耐药基因包括tetA(P)、tetB(P)、erm(Q)和lnu(P),与红霉素、克林霉素和四环素的较高耐药性对应(表S1)。先前的研究已证明了产气荚膜梭菌耐药基因如erm(Q)、tetA(P)和optrA与pCW3样和pCPCP样质粒之间的关联。然而在此次研究中,携带各种质粒组合的产气荚膜梭菌中均发现了这些耐药基因;耐药基因在产气荚膜梭菌中基因背景的多样性。作为一种重要的食源性病原体,产气荚膜梭菌的耐药性对公共卫生构成显著关注。此外,产气荚膜梭菌中的耐药基因有可能转移至其他细菌,造成更广泛的抗生素抗性传播。特别是在中国人源菌株中检出率较高的erm(Q),具有持续监测的必要性。
毒素CPE和BEC被认为是导致产气荚膜梭菌食物中毒的主要致病因子。在本研究中,分别在4株和1株产气荚膜梭菌中检测到cpe和becA/B。以往认为cpe位于染色体和pCW3类质粒上。我们的分析揭示了cpe与△IS1469、IS1151和dcm在分离株CQ147、CQ165和CQ169中的共定位。这一观察支持了假设:产气荚膜梭菌中的cpe质粒可能起源于cpe元件在产气荚膜梭菌质粒的dcm区域附近的整合。不同的是,在分离株CQ145中,cpe位于一个融合质粒上,与△IS1469和IS1151共存,但不与dcm共存。这表明质粒融合造成了产气荚膜梭菌中cpe的新遗传背景。
病原微生物的传播是食源性疾病爆发的主要原因。本研究中的一些产气荚膜梭菌分离株以及来自其他国家的人源菌株共享>99.99%的ANI,提示了在人类中产气荚膜梭菌的区域/跨区域传播的可能性。此外,我们发现这些菌株之间的小遗传差异很普遍,说明在产气荚膜梭菌菌株传播过程中频繁发生基因转移。CPB2由cpb2基因或非典型cpb2(aty.cpb2)基因编码。非典型基因与cpb2具有60~80%的DNA同一性,但与其他aty.cpb2基因有99%以上的同一性,然而多数aty.cpb2基因具有移码突变。先前的研究证明猪是cpb2的主要宿主,而aty.cpb2基因则主要在非猪源的产气荚膜梭菌中检测到,这提示了产气荚膜梭菌的潜在宿主特异性关联。然而,目前尚无研究比较探讨产气荚膜梭菌基因组特征与宿主种类之间是否存在相关性。在本研究中,我们对来自中国的人源产气荚膜梭菌分离株进行了系统发育和泛基因组分析。结果表明,虽然只有某些来自人类和动物的菌株在进化树中聚集在一起,但来自同一宿主来源的菌株大部分在系统发育树中聚集在一起,并共享相似的附属基因谱。这些发现表明产气荚膜梭菌菌株在相同宿主种类中具有较强关联性。
群体中的遗传标记通常反映了生物体与其外部环境之间的相互作用。在本研究中,我们观察到某些毒素基因如pfoA、nanI和colA流行情况随宿主种类而改变。最近的一项研究表明,人源低致病性产气荚膜梭菌中缺乏pfoA,因此我们推测:(i)产气荚膜梭菌菌株通过有选择地丢弃特定毒素基因以降低毒力,从而适应与特定宿主动物的共生;(ii)不同动物的肠道生理环境的差异可能有助于具有独特生物特征的菌株的定殖。猪和人类中colA、nanI、nanJ和nagI的低流行率表明这些基因可能在产气荚膜梭菌的定植和发病机制中起重要作用。尚需进一步研究以阐明这些毒素基因的生物功能。
结 论
总之,本研究全面调查了来自中国人群的产气荚膜梭菌分离株的基因组特征,以及全球范围内产气荚膜梭菌的群体结构和泛基因组特征。研究结果提供了关于产气荚膜梭菌菌株传播动力学、关键毒力标记的定位及产气荚膜梭菌与宿主种类之间关系的新见解。此外,我们的结果表明,毒素基因可能在决定产气荚膜梭菌的宿主选择性方面起关键作用,强调了进一步研究此病原体毒力的分子机制的重要性。
方 法
产气荚膜梭菌收集
从426名健康个体和273名重症监护室的患者中获得了共计220株产气荚膜梭菌菌株。研究对象包括在例行体检中被认为总体健康的成年人,他们在知情同意后参加了研究。该组的排除标准包括胃肠炎、妊娠期以及在前30天内接受抗生素治疗的个体。同时,ICU患者需要已住院至少七天,并在其监护人提供知情同意后纳入研究。产气荚膜梭菌菌株的分离和鉴定方法在以前的一项研究中已有描述。每个样本接种在胰蛋白胨-亚硫酸盐-环丝氨酸琼脂上,并在37℃下厌氧培养20-24小时。随后,对可疑菌落在5%脱纤维羊血琼脂上进行纯化并鉴定为产气荚膜梭菌。
基因组测序
使用PureLink Genomic DNA mini kit (Invitrogen, Carlsbad, CA, USA)提取产气荚膜梭菌分离株的基因组DNA。使用TruSeq DNA PCR-free sample preparation kit (Illumina, Inc., San Diego, CA)制备索引化的DNA文库,并使用Illumina HiSeq X Ten系统进行测序后,获得每个分离株覆盖深度至少150倍的300 bp末端双向读取序列。原始读取序列通过SPAdes 3.11.1版本进行修剪并组装成重叠群。使用checkM v.1.0.12评估基因组质量,> 95%完整性和< 5%污染的基因组被纳入后续基因组分析。
遗传标记鉴定
鉴于产气荚膜梭菌耐药性、毒力和质粒的临床重要性,我们检测了分离株毒素基因、耐药基因、pCW3和pCP13样质粒,以及新型pCPCPI53k-r1_1样(简称pCPCP样)质粒。使用ABRicate基于ResFinder和VFDB数据库(--minid 90 --mincov 90)筛查毒素基因和耐药基因。利用ABRicate基于自建的基因数据库鉴定Tcp和Pcp位点。Tcp(http://www.ncbi.nlm.nih.gov/nuccore/NC_010937.1)和Pcp(http://www.ncbi.nlm.nih.gov/nuccore/AP003515.1)位点编码基因被用作BLAST数据库。携带超过三个Tcp或Pcp位点编码基因的菌株被认为含有相应的质粒。泛基因组分析被用作确定产气荚膜梭菌基因组中类似pCW3和pCP13质粒存在的验证方法。分析中包括质粒pCW3和pCP13以及本研究中测序的195个产气荚膜梭菌分离株。使用Roary v3.13.0和FriPan鉴定pCPCP-like质粒;p21-D-5b、pQHY-2、p2C45和pCPCPI53k-r1_1被用作参考。在所有泛基因组分析中,BLASTP的最小序列一致性阈值设置为95%。
cpe和becA/B基因环境研究
cpe和becA/B基因是与人类食源性感染相关的重要毒力因子。为了加深对这些具有临床重要性的遗传标记的理解,我们调查了产气荚膜梭菌分离株中的cpe和becA/B的遗传环境。使用在线工具RAST对cpe和becA/B阳性片段进行序列注释,利用ISfinder(https://isfinder.biotoul.fr/about.php)和DANMEL筛查插入序列。序列比对使用EasyFig 2.2.2和BRIG 0.95进行。通过PCR检测验证分离株CQ145中cpe的遗传环境,并使用Sanger测序对PCR产物进行测序。
进化关系分析
为了揭示产气荚膜梭菌菌株的潜在传播途径和群体结构,本研究基于核心基因组SNPs构建了最大似然树。研究对象包括中国及全球的产气荚膜梭菌菌株。使用Snippy v4.6.0(https://github.com/tseemann/snippy)从这些菌株中提取核心基因组SNPs。系统发育树使用IQ-TREE 2 构建,参数设定为“-m MF”。所得树状图通过rhierBAPS进行分群,并使用在线工具iTOL(https://itol.embl.de/)进行注释。
泛基因组分析
大多数主要毒素基因和抗药基因属于附属基因,表明泛基因组分析的重要性和必要性。使用Prokka v1.14.5对产气荚膜梭菌菌株进行基因组注释。随后,使用Roary v3.13.0和IPGA进行泛基因组分析。选择在5%到95%的基因组中存在的附属基因进行比较,以评估各组间附属基因的差异。
数据统计分析
所有统计分析均使用R版本4.3.2进行。附属基因的NMDS分析使用vegan进行,并使用ggplot2绘制。使用Wilcoxon检验评估不同组别产气荚膜梭菌菌株中耐药基因和毒力基因的总数差异。Pearson卡方检验用于测试基因在编码毒力和耐药表型的频率差异是否显著。*: p < 0.05, **: p < 0.01, ***: p < 0.001。
代码和数据可用性
所有测序数据已提交至NCBI,BioProject登录号为PRJNA983292。数据和脚本保存于https://github.com/s-ckk/data-and-scripts-for-IMO-2024-0069。补充材料(图形、表格、图文摘要、幻灯片、视频、中文版翻译及更新材料)可见在线DOI或iMetaOmics网站(http://www.imeta.science/imetaomics/)。
引文格式:
Ke Wu, Juan Wang, Zelin Yan, Yanyan Zhu, Shaolin Wang, Bo Fu, Chengtao Sun, et al. 2024. “Genomic adaptation of Clostridium perfringens to human intestine.” iMetaOmics 2: e38. https://doi.org/10.1002/imo2.38
作者简介

吴克(第一作者)
● 四川大学生命科学学院在读博士研究生。
● 研究方向为细菌传播溯源和适应性,以第一作者(含共同)在iMetaOmics、mSystems、Journal of Antimicrobial Chemotherapy、Veterinary Microbiology、One Health Advances、Anaerobe发表SCI论文7篇。

王娟(第一作者)
● 西北农林科技大学动物医学院副教授,博士生导师。
● 主要致力于细菌致病及耐药机制研究,先后主持国家自然科学基金面上项目、青年项目、中国博士后科学基金特别资助、陕西省重点研发计划项目(国际合作项目)等国家级、省部级科研项目10余项。已发表科研论文 80 余篇,以第一作者或通信作者发表SCI收录论文20余篇,参编英文专著1部,农业行业标准1项,参与《兽医传染病学》省级一流课程建设项目。

王红宁(通讯作者)
● 四川大学生命科学学院教授,博士生导师。
● 长期坚持在生产一线从事动物疫病防控与动物产品安全的基础和应用研究,先后主持十四五国家重点研发计划、国家科技支撑计划、“863”、“973”、国家自然科学基金重点、区域创新基金重点及面上项目、国家现代农业产业技术体系岗位科学家、国家农业科技成果转化基金、国家公益性行业专项等项目。聚焦猪、鸡疫病防控,解决依赖抗生素防控细菌病,耐药性和药物残留严重,产品不安全难题。成果已在我国24个省市猪、鸡主产区大规模应用,经济社会生态效益显著。制定国家和地方标准8项,获国家发明专利授权12件,与企业合作研发获国家二类新兽药证书6项,主编出版专著6部,以第一或通讯作者在本领域主要刊物发表SCI论文百余篇。

张嵘(通讯作者)
● 浙江大学医学院附属第二医院检验科主任技师,博士生导师。
● 长期从事临床微生物检验,临床细菌耐药及毒力检测等研究工作。 主持国家自然科学基金、国自然海外合作中英项目课题3项,浙江省自然科学基金等项目若干。发表数百篇SCI论文,主编《微生物质谱应用问与答》等书籍。现担任中华微免临床微生物学组委员、中华医学会浙江省微免分会副主任委员等。

殷喆(通讯作者)
● 军事科学院军事医学研究院微生物流行病研究所副研究员。
● 主要研究方向为细菌遗传多态性、耐药机制与进化规律、生物气溶胶感染防控。发表论文50余篇,其中第一或通讯作者发表SCI论文28篇。累计影响因子135.16,他引136次。作为负责人承担国家重点研发计划“生物安全技术专项”子课题2项,后勤科研项目课题1项,国家重点实验室自主课题、863课题子课题各1项,作为骨干参与国家科技重大专项、后勤科研重大项目、国家自然科学基金等课题5项。
iMetaOmics
更多资讯
● iMeta姊妹刊iMetaOmics(定位IF>10)欢迎投稿!(2024.2.27)
● iMeta姊妹刊iMetaOmics编委招募 (定位IF>10) (2024.3.2)
● iMeta姊妹刊iMetaOmics电子版和印刷版ISSN申请获批(2024.4.1)
● iMeta姊妹刊iMetaOmics投稿系统正式上线(2024.4.17)
● iMeta姊妹刊iMetaOmics主编正式官宣(2024.4.22)
● 出版社iMetaOmics主页正式上线!(2024.4.28)
● iMetaOmics | 浙江大学宗鑫组揭示两猪种宿主-肠道菌群互作差异
● iMetaOmics | 罗鹏/袁硕峰/苗凯/程全发表STAGER: 生成式人工智能可靠性的标准化测试和评估推荐
● iMetaOmics | 徐州医科大杨欢组揭秘沙门氏菌-宿主-微生物群在免疫与代谢中的相互作
● iMetaOmics | 中科院动物所金坚石组综述16S rRNA基因扩增子测序技术的“前世今生”
● iMetaOmics | 浙大张天真组完成二倍体棉种泛基因组构建
● iMetaOmics | 张勇/李福平-先进糖蛋白组学在男性生殖研究中的潜在应用
● iMetaOmics | 暨南大学潘永勤/杨华组-炎症蛋白联合检测利于诊断甲状腺乳头状癌和结节性甲状腺肿
● iMetaOmics | 张开春组利用多组学方法揭示甜樱桃加倍后果色变化的候选基因
● iMetaOmics | 杜娟/林婷婷-慢性泪囊炎患者眼部菌群类型和纵向菌群变化
● iMetaOmics | 陈汉清/陈俊综述有关肝细胞癌治疗的新兴纳米医学策略
● iMetaOmics | 基因组所刘永鑫/卢洪评述微生物在提高杂种优势中的作用
● iMetaOmics | 上科大刘雪松组开发基于通路的肿瘤细胞鉴别工具TCfinder
● iMetaOmics | 中山大学刘鹏/邹宇田-整合人工智能实现HER2阳性乳腺癌精准管理
● iMetaOmics | 安徽农大李晓玉组-丛枝菌根真菌对玉米内生菌群的影响
● iMetaOmics | 徐涛/黄蓉/苏国海-急性冠脉综合征纵向多组学队列建设
● iMetaOmics | 通过整合宏组学促进人类与环境健康发展
● iMetaOmics | 苏州大学林俊组-揭示活性微生物及益生元/益生菌与关节炎联系
● iMetaOmics | 中国药科大学徐文波开发叶绿体基因组数据分析软件
● iMetaOmics | 清华刘晓组和复旦王久存组揭示特定细菌在皮肤老化中的作用
● iMetaOmics | 中南大学夏晓波团队揭示青光眼和SLE发病机制新关联
● iMetaOmics | 庐山植物园刘芬组揭示了自噬在植物-根微生物互作机制中的调控作用
● iMetaOmics | 杨瑞馥/袁静综述微生物组与“同一健康”的联系
● iMetaOmics | 同济/上海交大-开发支持群体分组分析的宏基因组测序综合分析软件
● iMetaOmics | 陈绍鸣-关于靶向NF-κB的潜伏逆转剂及其在HIV潜伏期的表观遗传和突变影响的评论
● iMetaOmics | 甘肃农大刘自刚组-强抗寒甘蓝型冬油菜的基因组组装和基因组特征解析
● iMetaOmics | 南京农大朱伟云组-外周血清素在结肠稳态中的作用
● iMetaOmics | 魏来/贾慧珏/何明光-多组学助力揭示塑造转录组的基因型-微生物组相互作用
● iMetaOmics | 徐州医科大学朱作斌组-微生物对寿命的调节:机制和治疗策略
● iMetaOmics | 白立景/邢凯组-解析脊椎动物肠道微生物多样性的影响因素
● iMetaOmics | 刘永鑫/陈同-用于食物微生物组成和时间序列研究的微生物组数据库FoodMicroDB
● iMetaOmics | 重庆大学王贵学组-肠道微生物细胞外囊泡在神经退行性疾病中的新作用及其治疗策略
更多推荐
(▼ 点击跳转)
iMeta | 引用14000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
3卷1期
2卷2期封底
2卷4期封底
3卷2期
3卷3期
3卷3期封底
3卷4期
3卷4期封底
1卷1期
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百千华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表所有领域高影响力的研究、方法和综述,重点关注微生物组、生物信息、大数据和多组学等。目标是发表前10%(IF > 20)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、前3年免出版费、50万用户的社交媒体宣传等。2022年2月正式创刊发行!发行后相继被Google Scholar、SCIE、ESI、PubMed、DOAJ、Scopus等数据库收录!2024年6月获得首个影响因子23.8,位列全球SCI期刊前千分之五(107/21848),微生物学科2/161,仅低于Nature Reviews,同学科研究类期刊全球第一,中国大陆11/514!
“iMetaOmics” 是“iMeta” 子刊,主编由中国科学院北京生命科学研究院赵方庆研究员和香港中文大学于君教授担任,是定位IF>10的高水平综合期刊,欢迎投稿!
iMeta主页:
http://www.imeta.science
姊妹刊iMetaOmics主页:
http://www.imeta.science/imetaomics/
出版社iMeta主页:
https://onlinelibrary.wiley.com/journal/2770596x
出版社iMetaOmics主页:
https://onlinelibrary.wiley.com/journal/29969514
iMeta投稿:
https://wiley.atyponrex.com/journal/IMT2
iMetaOmics投稿:
https://wiley.atyponrex.com/journal/IMO2
邮箱:
office@imeta.science








































 291
291

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








