PubMed是一个免费的搜寻引擎,提供生物医学方面的论文搜寻以及摘要。它的数据库来源为MEDLINE(生物医学数据库),其核心主题为医学,但亦包括其他与医学相关的领域,像是护理学或者其他健康学科。它同时也提供对于相关生物医学资讯上相当全面的支援,像是生化学与细胞生物学。
PubMed是因特网上使用最广泛的免费MEDLINE, 该搜寻引擎是由美国国立医学图书馆提供, 它是基于WEB的生物医学信息检索系统,它是NCBI Entrez整个数据库查询系统中的一个。PubMed界面提供与综合分子生物学数据库的链接,其内容包括:DNA与蛋白质序列,基因图数据,3D蛋白构象,人类孟德尔遗传在线,也包含着与提供期刊全文的出版商网址的链接等。
医学导航链接:http://www.meddir.cn/cate/736.htm
PubMed官网:http://pubmed.cn/
实现代码
实现的代码主要是Selenium通过分析网页DOM结点进行爬取。
爬取的地址是:http://www.medlive.cn/pubmed/
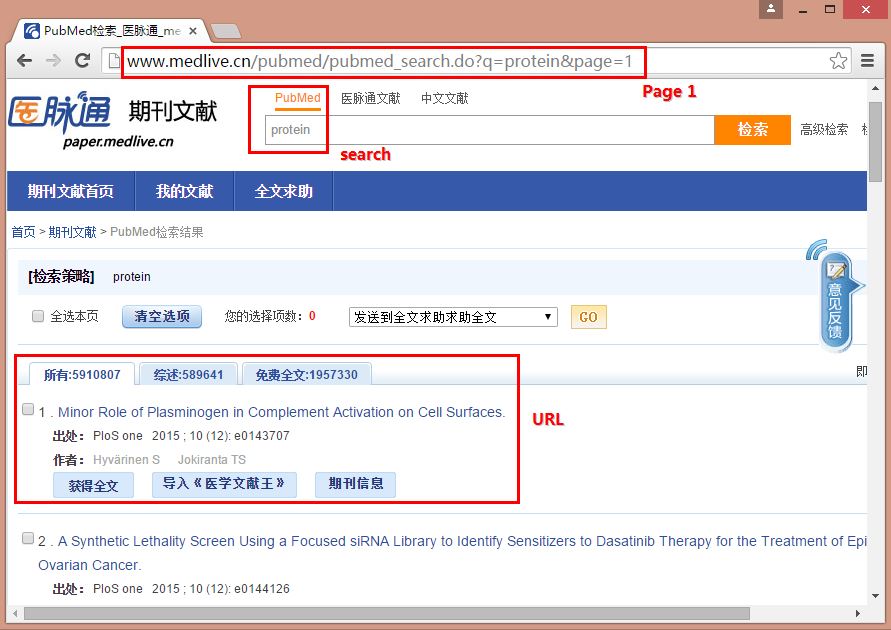
在网址中搜索Protein(蛋白质)后,分析网址可发现设置Page=1~20可爬取前1~20页的URL信息。链接如下:
http://www.medlive.cn/pubmed/pubmed_search.do?q=protein&page=1
# coding=utf-8
"""
Created on 2015-12-05 Ontology Spider
@author Eastmount CSDN
URL:
http://www.meddir.cn/cate/736.htm
http://www.medlive.cn/pubmed/
http://paper.medlive.cn/literature/1502224
"""
import time
import re
import os
import shutil
import sys
import codecs
from selenium import webdriver
from selenium.webdriver.common.keys import Keys
import selenium.webdriver.support.ui as ui
from selenium.webdriver.common.action_chains import ActionChains
#Open PhantomJS
driver = webdriver.Firefox()
driver2 = webdriver.PhantomJS(executable_path="G:\phantomjs-1.9.1-windows\phantomjs.exe")
wait = ui.WebDriverWait(driver,10)
'''
Load Ontoloty
去到每个生物本体页面下载摘要信息
http://paper.medlive.cn/literature/literature_view.php?pmid=26637181
http://paper.medlive.cn/literature/1526876
'''
def getAbstract(num,title,url):
try:
fileName = "E:\\PubMedSpider\\" + str(num) + ".txt"
#result = open(fileName,"w")
#Error: 'ascii' codec can't encode character u'\u223c'
result = codecs.open(fileName,'w','utf-8')
result.write("[Title]\r\n")
result.write(title+"\r\n\r\n")
result.write("[Astract]\r\n")
driver2.get(url)
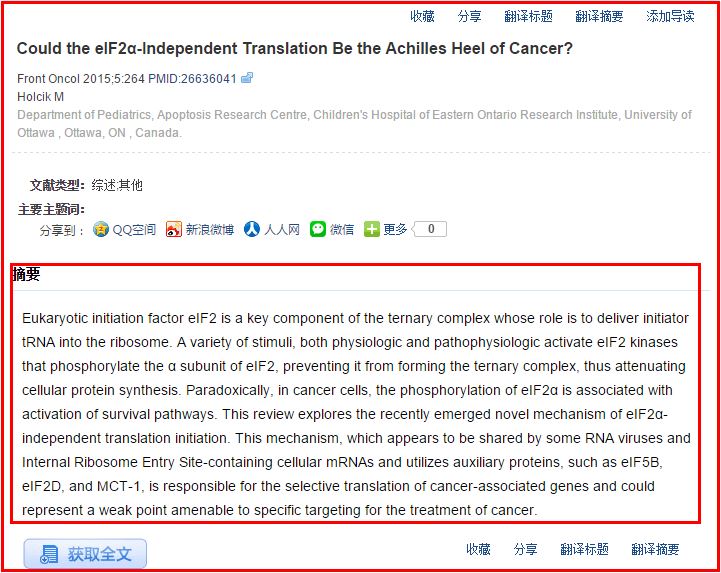
elem = driver2.find_element_by_xpath("//div[@class='txt']/p")
#print elem.text
result.write(elem.text+"\r\n")
except Exception,e:
print 'Error:',e
finally:
result.close()
print 'END\n'
'''
循环获取搜索页面的URL
规律 http://www.medlive.cn/pubmed/pubmed_search.do?q=protein&page=1
'''
def getURL():
page = 1 #跳转的页面总数
count = 1 #统计所有搜索的生物本体个数
while page<=20:
url_page = "http://www.medlive.cn/pubmed/pubmed_search.do?q=protein&page="+str(page)
print url_page
driver.get(url_page)
elem_url = driver.find_elements_by_xpath("//div[@id='div_data']/div/div/h3/a")
for url in elem_url:
num = "%05d" % count
title = url.text
url_content = url.get_attribute("href")
print num
print title
print url_content
#自定义函数获取内容
getAbstract(num,title,url_content)
count = count + 1
else:
print "Over Page " + str(page) + "\n\n"
page = page + 1
else:
"Over getUrl()\n"
time.sleep(5)
'''
主函数预先运行
'''
if __name__ == '__main__':
'''
path = "F:\\MedSpider\\"
if os.path.isfile(path): #Delete file
os.remove(path)
elif os.path.isdir(path): #Delete dir
shutil.rmtree(path, True)
os.makedirs(path) #Create the file directory
'''
getURL()
print "Download has finished."
分析HTML
1.获取每页Page中的20个关于Protein(蛋白质)的URL链接和标题。其中getURL()函数中的核心代码获取URL如下:
url = driver.find_elements_by_xpath("//div[@id='div_data']/div/div/h3/a")
url_content = url.get_attribute("href")
getAbstract(num,title,url_content)
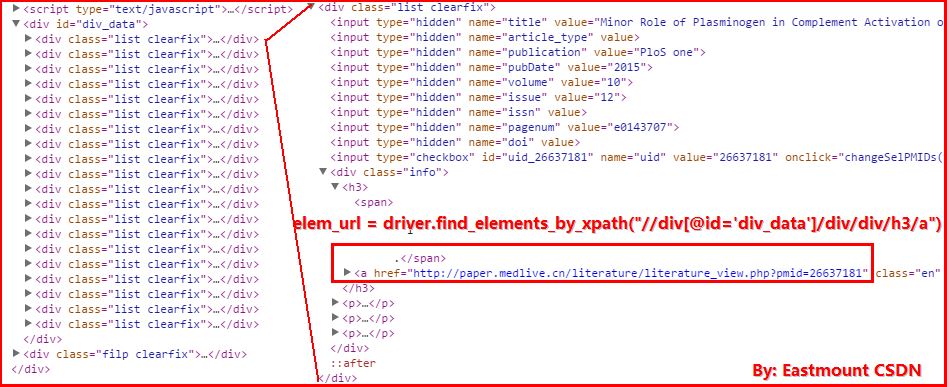
2.再去到具体的生物文章页面获取摘要信息

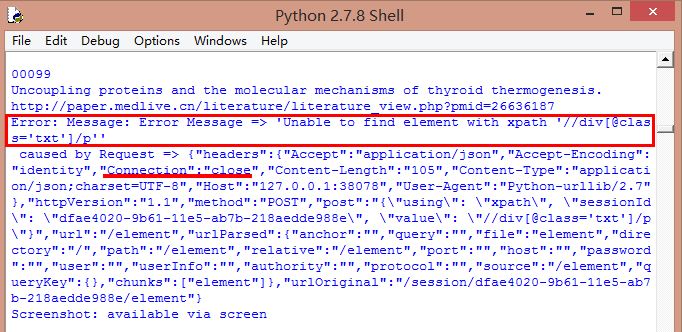
其中你可能遇到的错误包括:
1.Error: 'ascii' codec can't encode character u'\u223c'
它是文件读写编码错误,我通常会将open(fileName,"w")改为codecs.open(fileName,'w','utf-8') 即可。
2.第二个错误如下图所示或如下,可能是因为网页加载或Connection返回Close导致
WebDriverException: Message: Error Message => 'URL ' didn't load. Error: 'TypeError: 'null' is not an object
运行结果
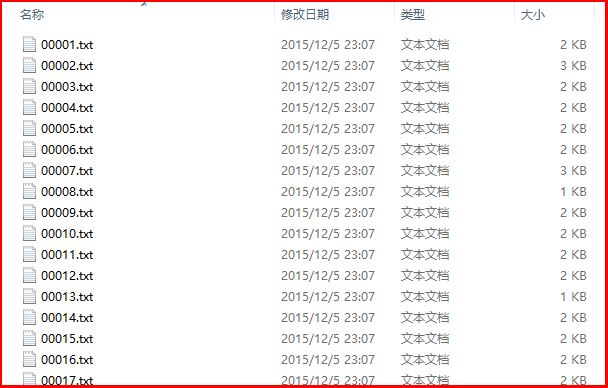
得到的运行结果如下所示:00001.txt~00400.txt共400个txt文件,每个文件包含标题和摘要,该数据集可简单用于生物医学的本体学习、命名实体识别、本体对齐构建等。
(By:Eastmount 2015-12-06 深夜3点半 http://blog.csdn.net/eastmount/)








 本文介绍使用Selenium爬取PubMed数据库中关于蛋白质的文章标题及摘要的方法。PubMed是一个免费的生物医学文献搜索引擎,涵盖广泛的生命科学领域。通过分析网页结构并利用Selenium,实现了对指定关键词搜索结果的自动化爬取。
本文介绍使用Selenium爬取PubMed数据库中关于蛋白质的文章标题及摘要的方法。PubMed是一个免费的生物医学文献搜索引擎,涵盖广泛的生命科学领域。通过分析网页结构并利用Selenium,实现了对指定关键词搜索结果的自动化爬取。


















 215
215

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










