本文主要介绍生物信息学软件 Mothur及其分析准备,接下来我们将持续更新Mothur中文教程。
Mothur是由密歇根大学微生物学和免疫学系的Patrick Schloss教授和其研究小组开发的一款开源、可扩展的软件,可用于生物信息学中的微生物生态群落分析。2009年2月发布了Mothur第一个版本,现已更新至V1.45.3。Mothur整合了dotur、sons、treeclimber、s-libshuff、unifrac等功能,可用于形成OTU/ASV,计算生物多样性指数。各位读者可以在Mothur官网下载其Windows版,并利用官网免费提供的标准化操作流程及命令说明,输入相关命令运行该软件,处理扩增子序列数据。目前,Mothur能够处理各种测序平台产生的序列数据,具体包括:454个焦磷酸测序,llumina公司的HiSeq和MiSeq, Sanger测序法,以及PacBio和IonTorrent等代表的三代测序技术。
Miseq SOP大致流程为:测序得到的PE reads首先根据overlap关系进行拼接,同时对序列质量进行质控和过滤,区分样本后进行OTU聚类分析和物种分类学分析。基于OTU可以进行多种多样性指数分析,以及对测序深度的检测;基于分类学信息,可以在各个分类水平上进行群落结构的统计分析。在上述分析的基础上,可以对多样本的群落组成和系统发育信息进行多元分析和差异显著性检验等一系列深入的统计学和可视化分析。
01 运行模式介绍
交互模式:从Mothur中输入用于分析的命令为交互模式。如果进行大量此类分析,或者在自己的数据上使用此SOP不必考虑太多,可以使用其他两个选项。
批处理模式:从wiki下载的文件夹中有一个名为stability.batch的文件。打开它可以看到运行的所有命令,但它不列出文件名,而是始终使用当前选项。可以从这个文件中复制和粘贴,并获得与上面相同的输出。批处理模式的优点在于:可以从命令行运行Mothur,无需输入太多内容。例如,可以运行以下命令:
$ ./mothurstability.batch不要输入代表提示的“$”。坐下来等待,让它继续。这就是所谓的批处理模式。这样做时,运行大约需要2.25分钟。这种方法的另一个好处是,可以使用此文件更改在make.contigs中列出的文件的名称。还可以使用“#”字符将注释输入批处理文件。
命令行模式:运行Mothur的第三种方法是直接使用命令行模式输入Mothur命令。是这样做的:
$./mothur"#make.contigs(file=stability.files, processors=8)"此命令将启动Mothur,运行make.contigs,然后退出。这对于想要编写命令脚本并在不同程序之间来回切换的人很有用。这里的关键要素是命令周围的引号和告诉Mothur这不是批处理文件的“#”字符。可以使用“;”组合命令像这样的字符:
$./mothur"#make.contigs(file=stability.files, processors=8);screen.seqs(fasta=current, maxambig=0, maxlength=275);unique.seqs();……;classify.otu(list=current, count=current, taxonomy=current, label=1);"运行Mothur时的另一个重要资源是日志文件。如果转到运行Mothur的文件夹,会找到一个或多个类似于mothur.1364488920.logfile 的文件。打开它,可以看到输入的所有命令以及显示在屏幕上的输出。
02 MiSeq SOP准备工作
硬件准备:笔记本电脑联想小新Pro-13ARE 2020(处理器:AMD R7-4800U 8核16线程,内存:2*8GB,硬盘:512GB SSD,操作系统:Windows 10 64bit,基于x64的处理器)
软件与数据准备
需要下载下列文件:

其中,miseqsopdata.zip是实例数据;Mothur.win.zip是软件Mothur Version 1.45.3安装包;silva.bacteria.zip是对比数据库;trainset9_032012.pds.zip是训练器。资源下载于https://mothur.org/wiki/miseq_sop/。
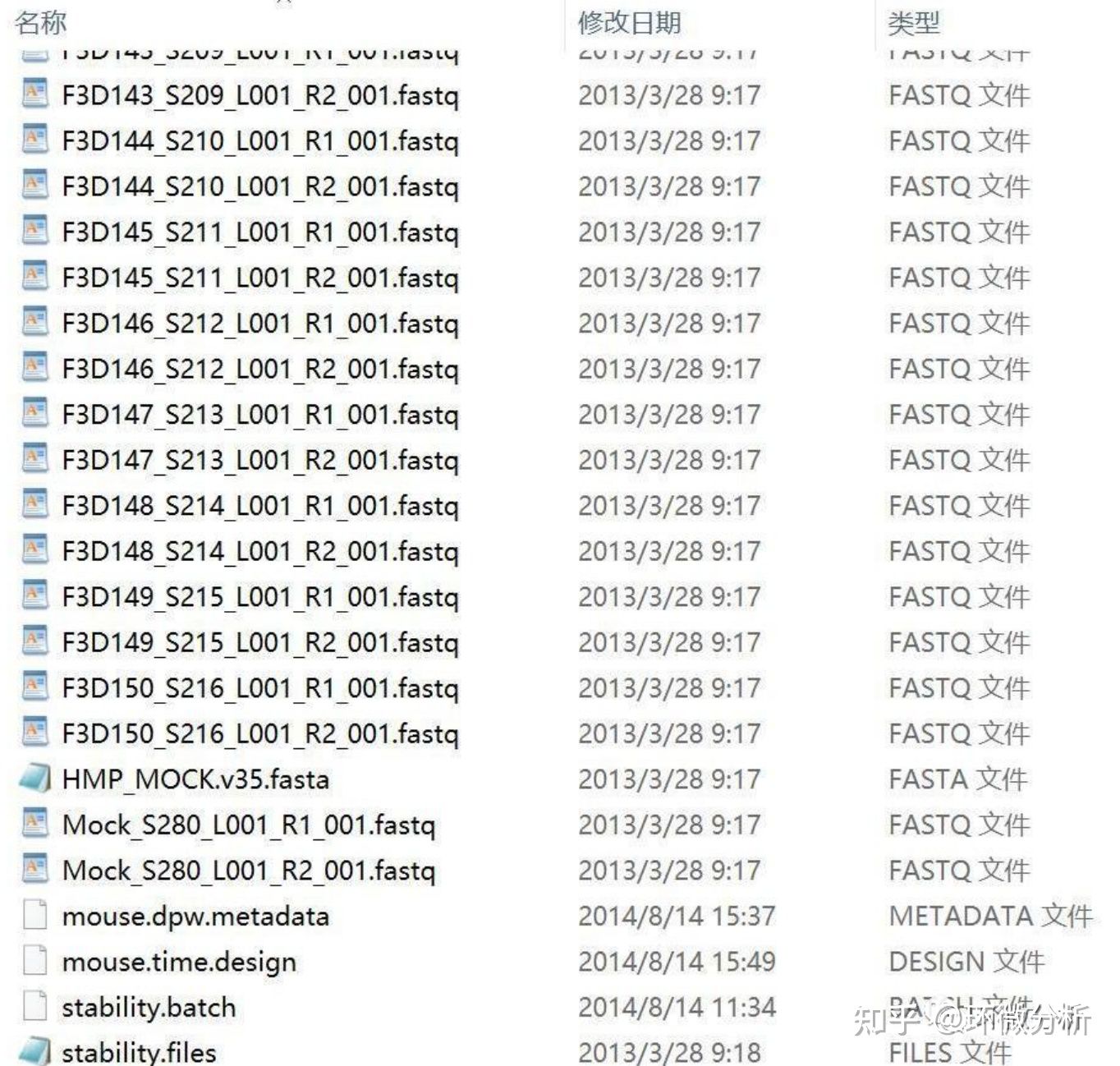
miseqsopdata文件夹内容包括:
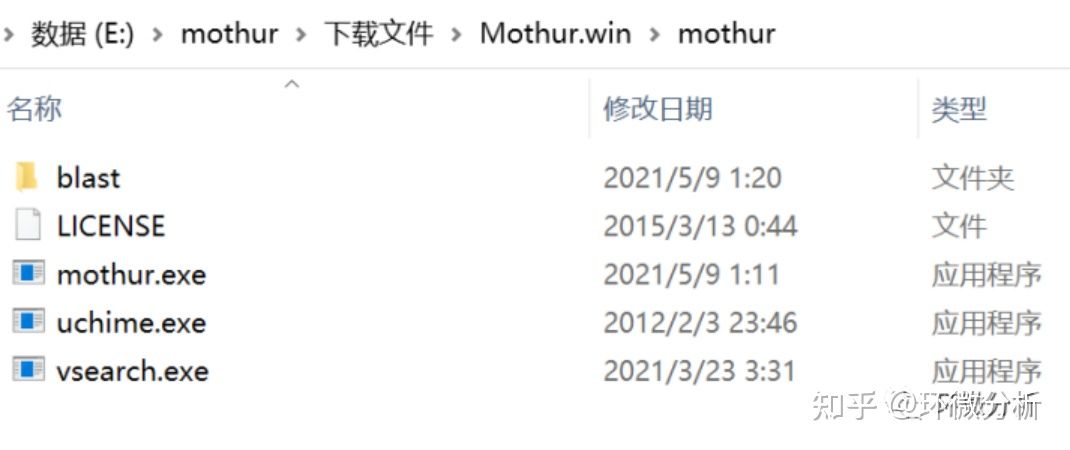
Mothur.win文件夹内容包括:
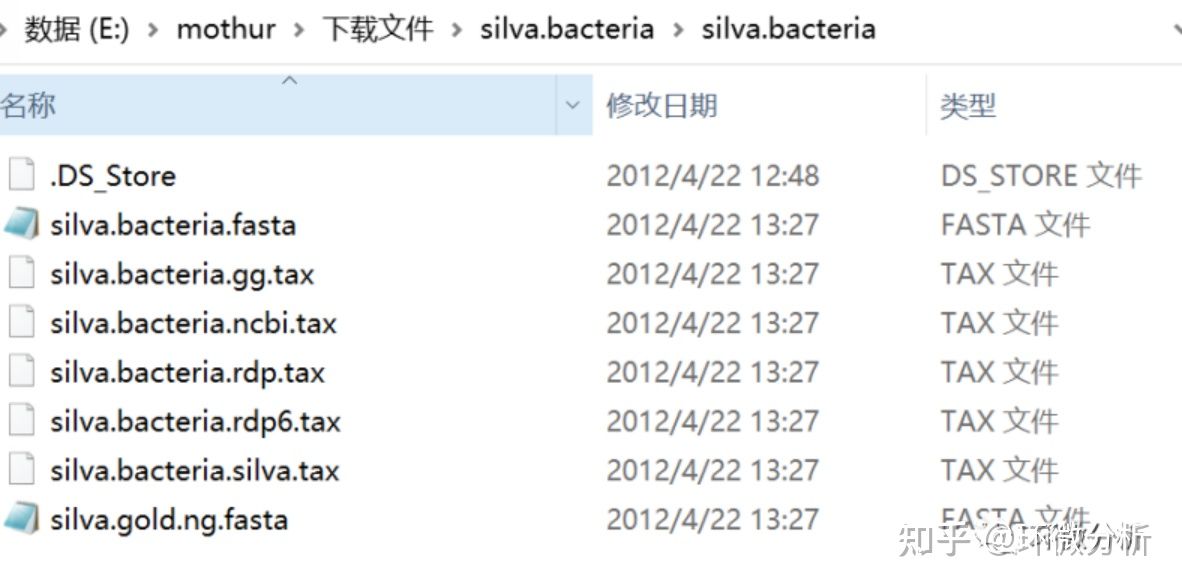
silva.bacteria文件夹内容包括:
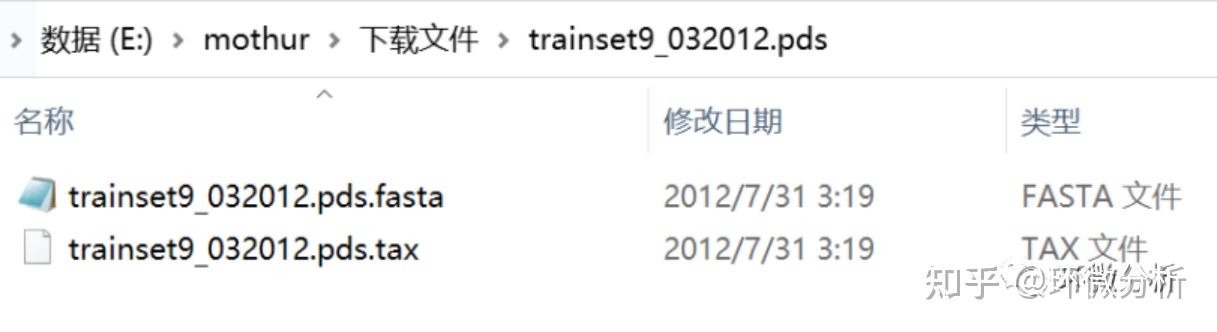
trainset9_032012.pds文件夹内容包括:
注意:虽然这是一个标准操作程序(Standard Operation Procedure, SOP),但它仍然是一个正在进行的工作。当对Mothur有了更加深入的了解之后会继续修订本SOP。如果在论文中使用了该方案,则须引用Schloss等人2013年于AEM上发表的论文以及您的访问日期。引文内容:Kozich JJ, Westcott SL, Baxter NT, Highlander SK, Schloss PD. (2013): Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform. Applied and Environmental Microbiology. 79(17):5112-20.
本教程旨在演示Schloss实验室用于处理由Illumina的MiSeq平台通过配对末端读取(PE reads)生成16S rRNA基因序列的SOP。该实验室采用的方法是使用索引序列(index reads)在单次运行中同时处理大量样本(即384个),将其应用于生成这些库最新的湿实验wet-lab SOP中。其他人也生成了类似的数据,但没有使用索引序列(index reads)。因此在每段序列的始端都能够找到索引(aka barcode)序列,本SOP将突出这两种方法在处理过程中的差异。本SOP是集成一系列已经出版的手稿产物,建议各位读者查阅这些手稿以获取更多详细信息和背景数据在一份正由Applied & Environmental Microbiology审阅的稿件中描述了MiSeq的具体步骤。
首先确认要研究的问题,Schloss实验室将研究肠道微生物组的正常变化对宿主健康的影响。为此,该实验室每天收集断奶后的小鼠的新鲜粪便,为期365天。断奶后的前150天(day post weaning,dpw),未对小鼠进行任何处理,小鼠们只负责健康快乐的长大。该实验想知道,与第140天-150天检测到的微生物组相比,小鼠断奶后的前10天体重的快速增长是否影响了其微生物组的稳定性。在本教程中将综合利用操作分类单元(OTU)、扩增子/精确序列变体(ASV/ESV)、phylotype和phylogenetic的组合来解决这个问题。为了使本教程更容易执行,本教程仅提供部分数据——单只小鼠10个时间点(5个早期和5个晚期)的数据文件(the flow files)。此外,为了对小鼠粪便样本进行测序,实验室对由21株细菌的基因组DNA组成的模拟群体进行了二次测序。因此将使用10个粪便样本来展示如何分析微生物群落和模拟群落,以检测错误率及其对其他分析的影响。
在提交给Applied & Environmental Microbiology的稿件中,描述了一组引物,可让仅使用80个引物(32+48)对1536个样本进行平行测序,获得质量同比454 SOP测序结果的测序序列。请参阅该稿件的附件以获取更多信息和wet-lab SOP。该研究的所有数据都可通过实验室的服务器获得。MiSeq的序列是成对的fastq文件,每对代表每个样本的两组读取。fastq文件包含序列数据和质量得分数据。如果没有从测序机器中获取这些文件,那么很可能是软件参数设置不正确。对于本教程,将需要几组文件。为了加快教程的速度,教程中提供了一些需要一段时间才能生成的下游文件(例如shhh.flows的输出):
来自schloss 实验室的示例数据(提取自完整数据集)将与本教程一起使用
基于SILVA的细菌参考比对
RDP训练集的Mothur格式版本(v.9)
可以使用最新的Silva参考文件,RDP参考文件和Greengenes格式的数据库将上述选项替换以作参考和分类法比对。使用上述文件是因为它们短小精悍、使用方便且效果很好。不同的分类参考在不同的样本类型中表现不同,因此分析过程可能会有所不同。通常最简单的方法是解压缩这些文件,然后将Trainset9_032012.pds和silva.bacteria文件夹的内容移动到MiSeq_SOP文件夹中。还需要将Mothur可执行文件夹的内容也移到那里。如果你是系统管理员向导(或新手),你可能想弄清楚如何将Mothur放在您的路径中,以下内容将为您解决这些问题。
此外,你可能还想了解以下内容:
atom/emacs/vi/或其他一些文本编辑器
R,Excel或者是其他用于绘制数据的程序
Adobe Illustrator,Safari或inkscape
figtree或其他程序以可视化树状图
由于文件名很长,对于许多命令使用“current”选项通常是最简单的。本教程旨在向人们展示如何具体使用Mothur,所以教程只会选择性地使用current选项来演示它是如何工作的。教程通常使用文件的全称。
这篇推文对你有帮助吗?喜欢这篇文章吗?喜欢就不要错过呀,关注本知乎号查看更多的环境微生物生信分析相关文章。亦可以用微信扫描下方二维码关注“环微分析”微信公众号,小编在里面载入了更加完善的学习资料供广大生信分析研究者爱好者参考学习,也希望读者们发现错误后予以指出,小编愿与诸君共同进步!!!

学习环境微生物分析,关注“环微分析”公众号,持续更新,开源免费,敬请关注!
转载自原创文章:
Mothur1_简介及运行准备_生信分析太难?Mothur学起来,免费生信分析课(文档,代码,数据)持续更新,值得系统学习
最后,再次感谢你阅读本篇文章,真心希望对你有所帮助。感谢!
























 2628
2628

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








