1.比对软件
作为初学者,在这里使用hisat2作为比对软件,关于常用的比对软件的特点引述一下hoptop的介绍:
最近的Nature Communication发表了一篇题为的Gaining comprehensive biological insight into the transcriptome by performing a broad-spectrum RNA-seq analysis的文章–被称之为史上最全RNA-Seq数据分析流程,也是我一直以来想做的事情,只不过他们做的超乎我的想象。文章中在基于参考基因组的转录本分析中所用的工具,是TopHat,HISAT2和STAR,结论就是HISAT2找到junction正确率最高,但是在总数上却比TopHat和STAR少。从这里可以看出HISAT2的二类错误(纳伪)比较少,但是一类错误(弃真)就高起来。
就唯一比对而言,STAR是三者最佳的,主要是因为它不会像TopHat和HISAT2一样在PE比对不上的情况还强行把SE也比对到基因组上。而且在处理较长的read和较短read的不同情况,STAR的稳定性也是最佳的。
就速度而言,HISAT2比STAR和TopHat2平均快上2.5~100倍。
文章链接:https://www.jianshu.com/p/681e02e7f9af
文献链接:
1:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-016-0881-8
2:https://www.nature.com/articles/s41467-017-00050-4
2 Hisat2的使用
HISAT2是TopHat2/Bowti2的继任者,使用改进的BWT算法,实现了更快的速度和更少的资源占用,因此作者推荐TopHat2/Bowti2和HISAT的用户转换到HISAT2,其官网地址是:http://ccb.jhu.edu/software/hisat2/index.shtml hisat2的使用主要有建立基因组Index,reads比对和sort排序。
我们可以从hisat2官网下载需要的人类和小鼠的Index,也可以自己在本地创建
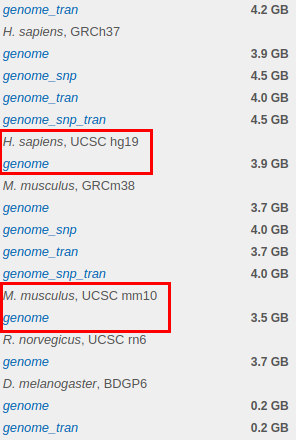
2.1 下载Index
cd ~/data/hg19
#下载链接
wget ftp://ftp.ccb.jhu.edu/pub/infphilo/hisat2/data/hg19.tar.gz
wget ftp://ftp.ccb.jhu.edu/pub/infphilo/hisat2/data/mm10.tar.gz








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章















 307
307

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








