动物食性分析是进行种群科学管理的前提。研究群落范围内的饮食-微生物群落相关性模式长期存在的问题在于:很难准确地确定自由放养的动物所吃的食物。而eDNA可以很好的表征饮食和微生物群落,为分辨饮食和微生物群落之间的细微变化创造了机会,是动物食性研究的利器!
那么对于食性分析中不同被捕食物种相互之间具有什么样的相互关联,复杂食物网络如何构建?这就要用到目前非常火的扩增子多营养级联合分析!(真菌、细菌、古菌也能联合分析啦!再来发一篇高分SCI吧!)
凌恩生物推出eDNA多营养级联合分析新策略,助您攻克生境中不同营养级物种群落关联机制研究难题。
接下来给大家分享eDNA宏条码技术分析动物食性的案例!
案例1 eDNA解析青藏高原多种食肉动物复杂食物网特征

-
发表期刊:Current Biology
-
影响因子:10.9
-
DOI:org/10.1016/j.cub.2022.12.049
研究设计
本研究采集了青藏高原食肉动物粪便603份,通过粪便DNA条形码技术(fecal DNA metabarcoding)来分析食肉动物的食物组成。
主要引物
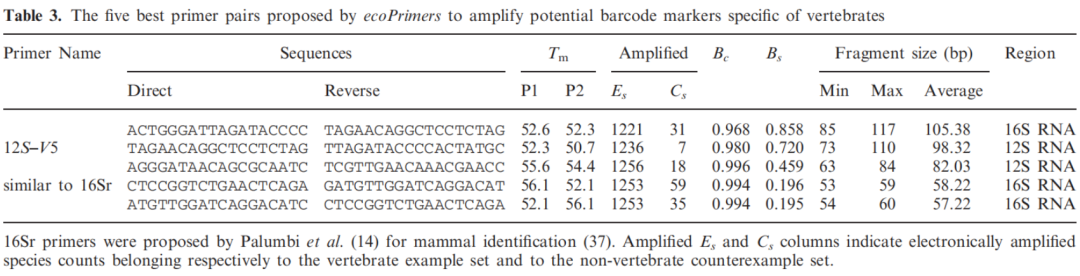
主要内容
全球范围内大规模的食肉动物灭绝导致了营养退化和生态系统服务受损。然而,多种食肉动物食物网的复杂性使其研究受阻。本研究利用eDNA技术深入解析了青藏高原地区食肉动物的精细食性组成。以青藏高原三江源地区的3个乡为研究区域,在野外系统收集889份食肉动物粪便样品,成功测序鉴定出食肉动物粪便603份,确定来源于4科的12种食肉目哺乳动物;利用粪便eDNA 技术获得其中8种主要食肉动物509份样品的1.74亿条序列数据,检测到11目的44种脊椎动物食物。基于分子食性分析结果,针对3个地点的食肉动物群落开展了定量食物网关系和网络结构的分析。
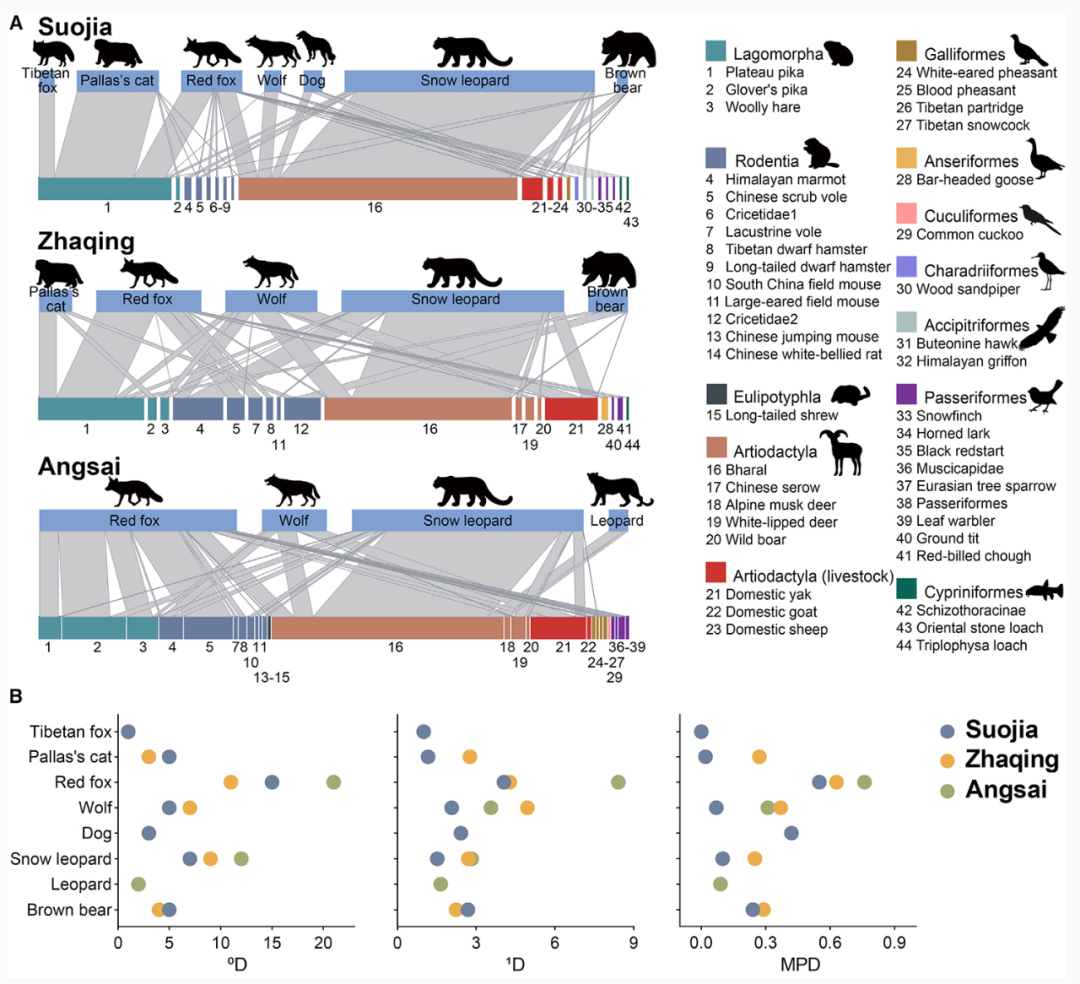
图1 3个研究区8种食肉动物的定量食物网和膳食多样性指标
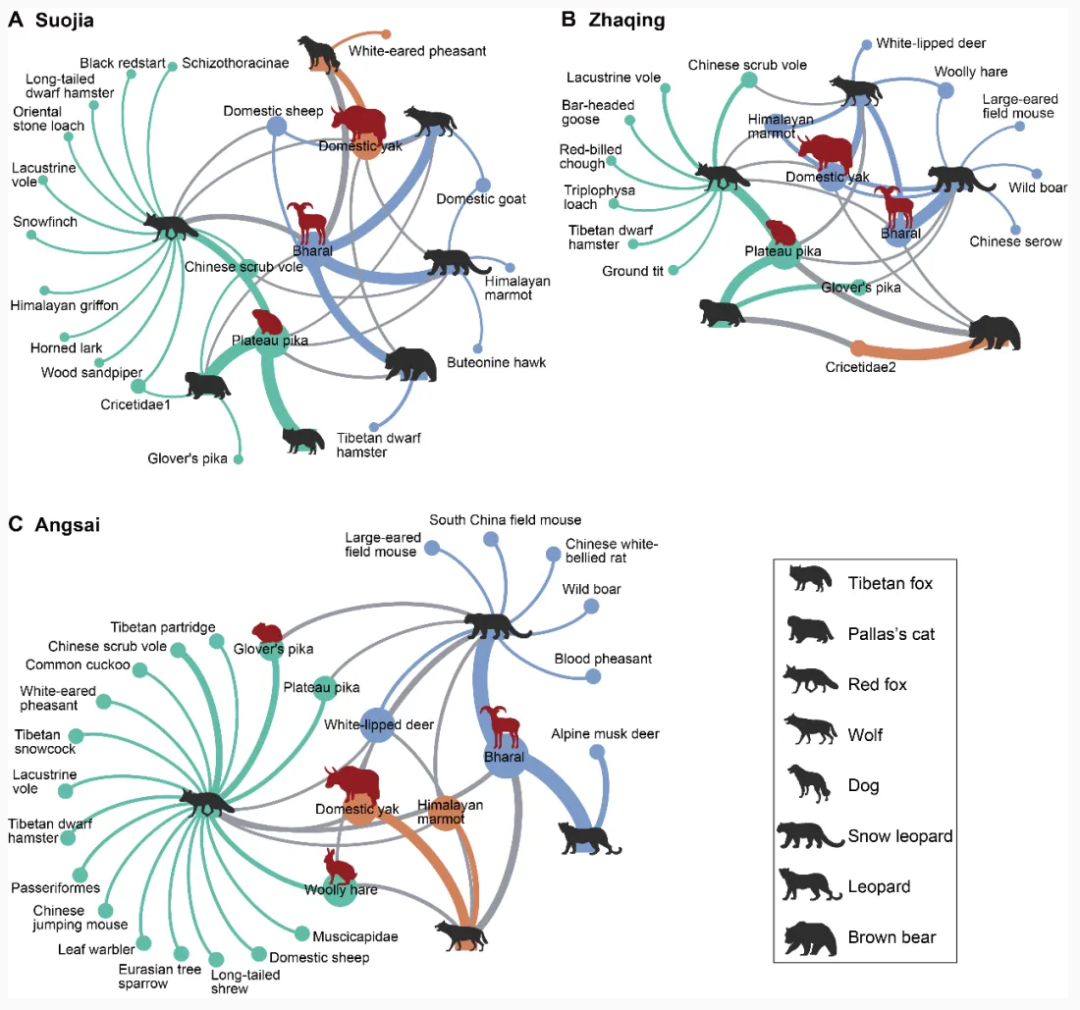
图2 青藏高原食肉动物食物网普遍存在模块化结构(绿、蓝、橙色显示不同模块;黑色图标显示食肉动物,深红图标显示关键猎物)
案例2 鲸鱼食性分析

-
发表期刊:Ecological Indicators
-
影响因子:7.1
-
DOI:org/10.1016/j.ecolind.2023.110125
研究设计
在2021年7月2 - 3日在大鹏湾发现鲸鱼“小布”的热点区域的6个地点,使用5L的水载体采集了水样。由于采样点深度<10m,所以从表层以下1m和底层以上1m采集了水样,然后混合在无菌塑料瓶中。1L水样使用醋酸纤维素微纤维膜(0.45μm)进行过滤。eDNA分析小布的食性。
主要引物
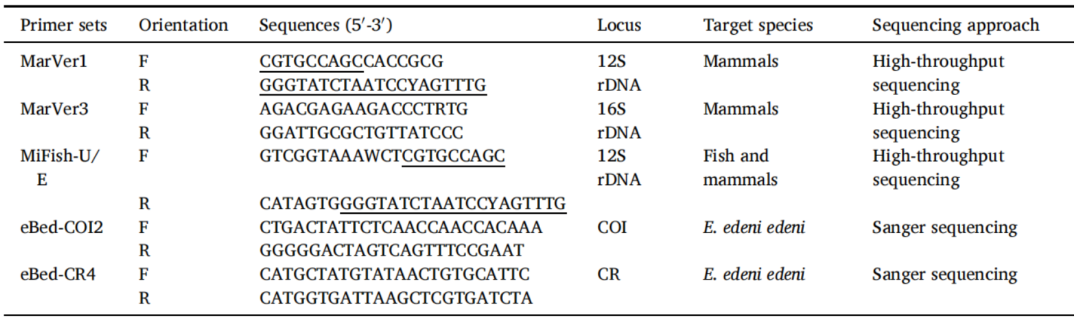
主要内容
由于鲸类本身数量稀少、且其行为难以捉摸,加之传统研究方法的限制,其监测工作面临多重挑战,这极大阻碍了保护行动的开展。本研究利用eDNA技术对深圳大鹏湾的一头须鲸(昵称“小布”)进行种类鉴定。通过获取的4个线粒体基因序列片段,证实其属于布氏鲸近岸亚种(Balaenoptera edeni edeni)。利用eDNA食性分析发现小布潜在的食物主要是沙丁鱼类和鳀鱼类。通过与邻近海域鱼类群落结构的对比,结合小布氏鲸的发现海域和时间,推断其很有可能是追踪这些洄游的鱼类进入大鹏湾。
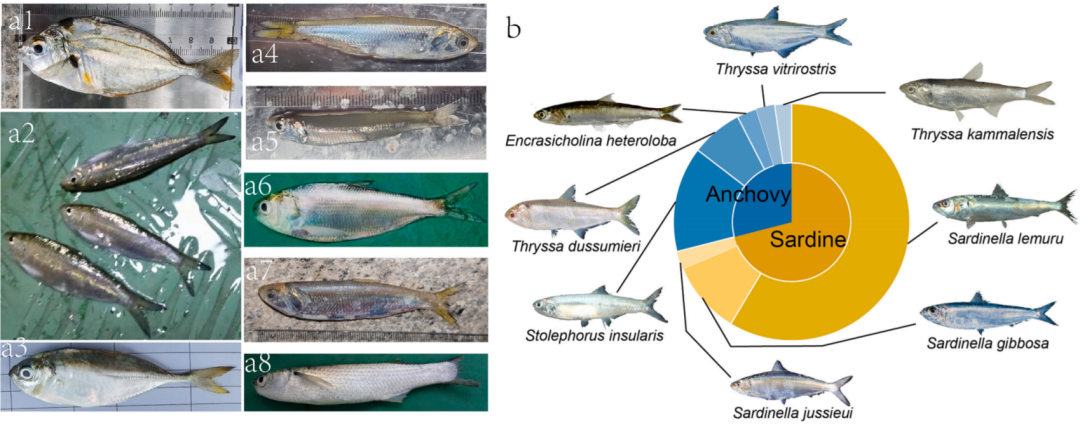
图3 小布的食物组成分析
案例3

-
发表期刊:Science Advances
-
影响因子:13.116
-
DOI:10.1126/sciadv.abf5908
研究设计
2018年7月在里索海豚(n = 4)和居维叶喙鲸(n = 2)的觅食生境中集中取样,确定了最大底部深度分别为922和1600 m。并按特定深度进行水样采集,每个采样点进行3次生物学重复,总共进行了144次收集。过滤后用于DNA提取和后续分析。应用18s和线粒体16s两对特异性引物检测eDNA生物多样性。
主要引物

主要内容
eDNA是一种在深海中建立头足类动物多样性和分布的有效技术,尤其是在使用垂直分层方法进行研究时效果更好。研究已经在亚速尔群岛周围的水域中发现了代表17个科的39个头足类群,另外还在该地区发现了两个新物种。这些为弄清捕食的特征和程度以及猎物的分布和种群组成,对于全面了解深海的食物网和碳预算至关重要。这种开创性的方法组合可以转移到其他捕食者——猎物系统,从而为增进我们对开放海洋和深海食物网的了解创造机会。


凌恩生物eDNA这个夏日最给力的优惠 来喽~
超级优~惠!
更有免费的云平台诚邀您使用!详情请后台联系我们~~~
好技术,好方案,高分paper只等你来!
凌恩生物竭诚服务您的科研
快来联系我们吧
参考文献
[1]Food webs reveal coexistence mechanisms and community organization in carnivores. Current Biology, 2023.
[2]Assessing the potential use of environmental DNA for multifaceted genetic monitoring of cetaceans: Example of a wandering whale in a highly disturbed bay area. Ecological Indicators, 2023
[3]Deep-sea predator niche segregation revealed by combined cetacean biologging and eDNA analysis of cephalopod prey.Science Advances,2021.
























 511
511

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










