Dayu Hu, Ke Liang, Sihang Zhou, Wenxuan Tu, Meng Liu, Xinwang Liu, scDFC: A deep fusion clustering method for single-cell RNA-seq data, Briefings in Bioinformatics, Volume 24, Issue 4, July 2023, bbad216, scDFC: A deep fusion clustering method for single-cell RNA-seq data | Briefings in Bioinformatics | Oxford Academic
代码:
摘要
聚类方法已被广泛应用于单细胞RNA测序数据以研究肿瘤的异质性。由于传统的聚类方法无法捕捉高维数据,近年来深度聚类方法因其在任务上的有希望的优势而越来越受到关注。然而,现有方法要么考虑每个细胞的属性信息,要么考虑不同细胞之间的结构信息。换句话说,它们无法同时充分利用所有这些信息。为此,本文提出了一种新颖的单细胞深度融合聚类模型,其中包含两个模块,即属性特征聚类模块和结构-注意特征聚类模块。更具体地说,构建了两个精心设计的自编码器来处理这两种特征,而不考虑它们的数据类型。实验证明了所提出方法的有效性,表明在单细胞RNA测序数据上融合属性、结构和注意信息是有效的。
动机:进而引出注意力
首先,目前尚不确定结构信息是否始终是首选的。细胞的状态与宿主代谢密切相关,仅依靠相关系数来映射细胞之间的关系可能不足以满足需求。虽然构建细胞间图已显示出提高聚类准确性的潜力,但生物环境的多样性和复杂性使得构建准确代表细胞之间关系的可信图形成了一项具有挑战性的任务。不准确的结构信息可能损害信息共享模式,最终削弱聚类性能。
另外,图卷积网络(GCNs)通常在不进行识别的情况下在相邻细胞之间传输信息。由于单细胞RNA数据本质上具有噪音,因此细胞间图中的许多边缘连接错误是很常见的。因此,在处理单细胞RNA数据时,注意机制是必要的,以确保获得准确的图结构信息。
模型架构
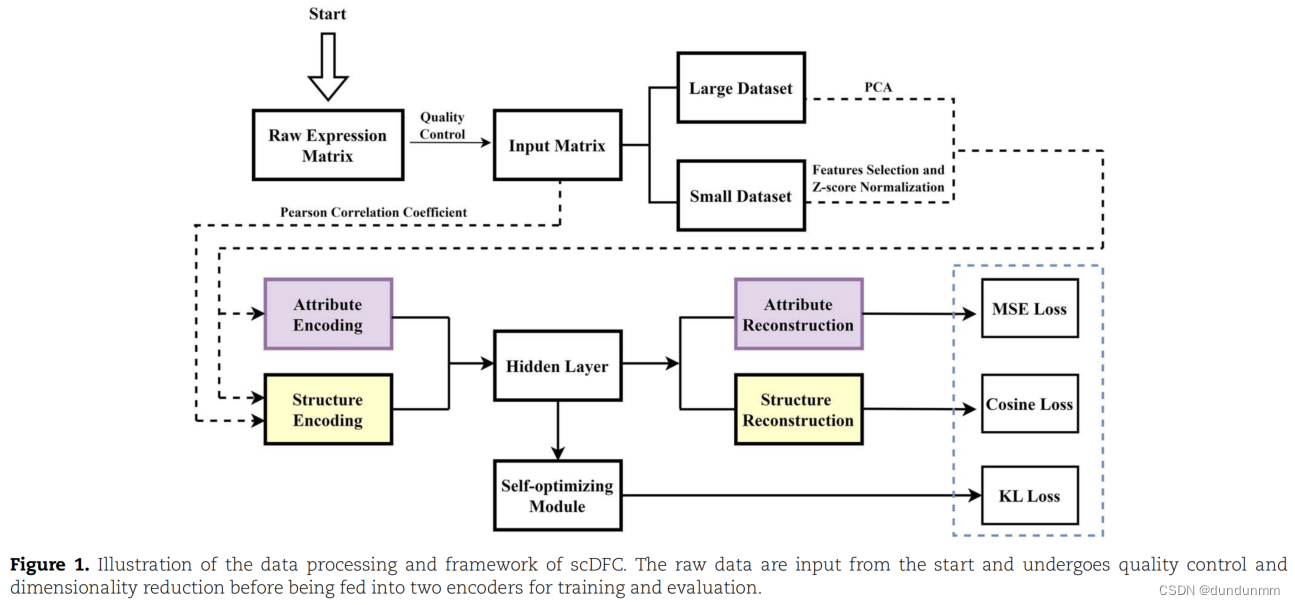
模型主要是AE和GAT的融合。
总体学习目标包括两个主要组成部分:自编码器(AE)和图注意力网络(GAT)的重构损失,以及聚类损失。自编码器模型中的重构损失确保网络学习到输入数据的有意义且压缩的表示。聚类损失衡量了数据的聚类分配与聚类算法生成的预测聚类分配之间的差异。联合损失被表述如下:
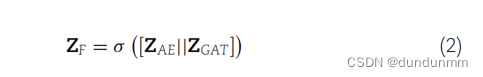
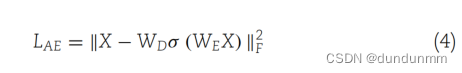

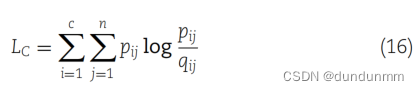
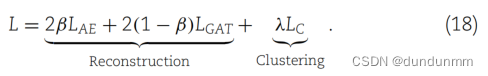
实验
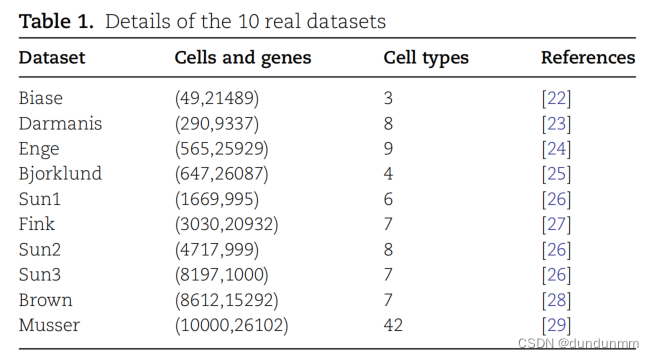
数据集
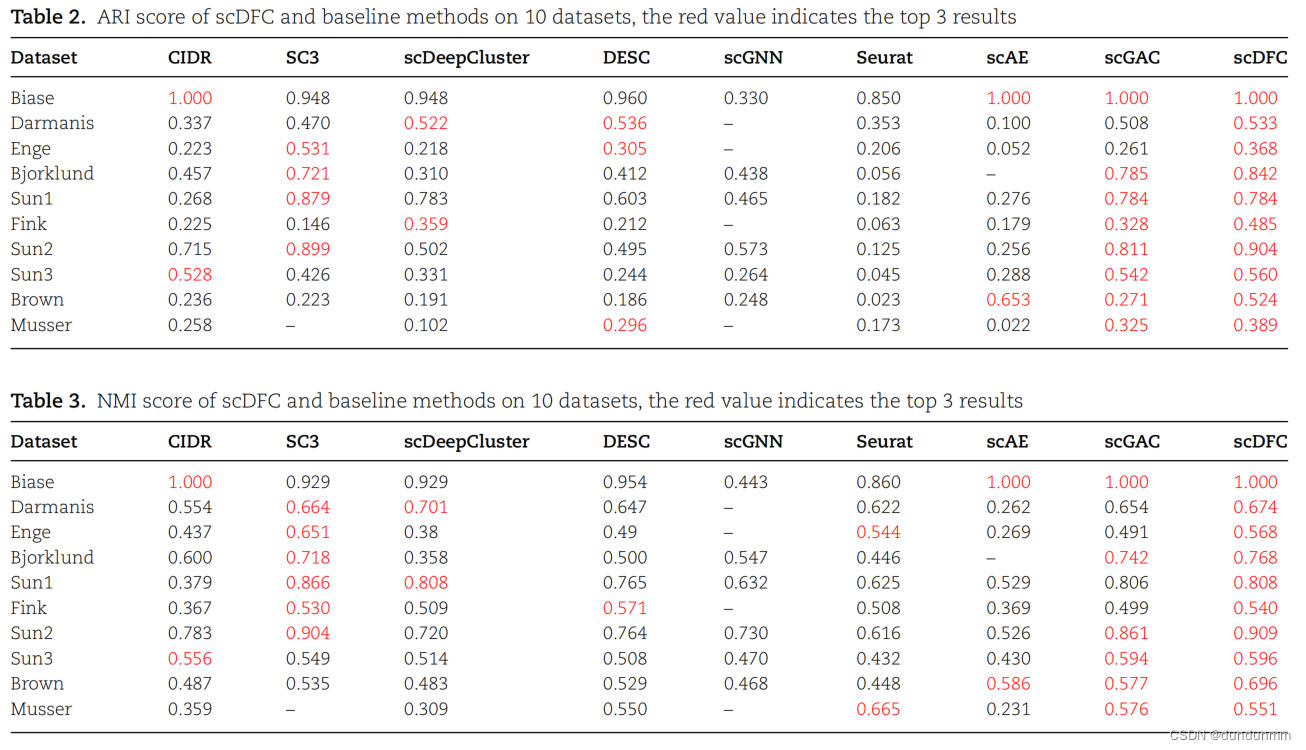
实验结果
类似,都选择了GAT.
但是,两篇文章的出发点是不同的,一篇文章的出发点和立意太重要了!























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








