目录
核心:气泡的信息主要体现在气泡大小和气泡颜色变化。
普通气泡图ggplot2
rm(list = ls())
library(ggrepel)
library(ggplot2)
dat <- mtcars数据格式查看:
head(dat)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258
p1 <- ggplot(data=mtcars, aes(x=wt,y=mpg))+
geom_point(aes(size=disp,fill=disp), ##展示点的数据
shape=21,colour="black",alpha=0.8)+
# 绘制气泡图,填充颜色和面积大小
scale_fill_gradient2(low="#377EB8",high="#E41A1C",
limits = c(0,max(mtcars$ disp)),
midpoint = mean(mtcars$disp))+ #设置填充颜色映射主题(colormap)
scale_size_area(max_size=12)+ # 设置显示的气泡图气泡最大面积
geom_text_repel(label = mtcars$disp)+ # 展示disp的具体数值
theme_bw()
p1
dev.off()更多关于标签相关的设置:R语言可视化学习笔记之ggrepel包 - 简书 (jianshu.com)
R语言-ggplot自定义点的形状、线条的类型_ggplot点的形状-CSDN博客

相关性气泡图
气泡大小展示相关系数和气泡颜色都只有一个图例:
数据处理1:生成两个dataframe
rm(list = ls())
library(ggplot2)
library(tidyverse)
library(ggrepel)
library(corrplot)
library(reshape2)
library(Hmisc)
#生成模拟数据①:表达矩阵
data <- sample(1:15, 225, replace = TRUE)
matrix_data <- matrix(data, nrow = 15, ncol = 15)
df <- data.frame(matrix_data)
colnames(df) <- paste0("gene",1:ncol(df))
rownames(df) <- paste0("sample",1:nrow(df))
#生成模拟数据②:某种评分矩阵
data1 <- sample(50:100, 225,replace = TRUE)
matrix_data1 <- matrix(data1, nrow = 15, ncol = 15)
df1 <- data.frame(matrix_data1)
colnames(df1) <- paste0("score",1:ncol(df))
rownames(df1) <- paste0("sample",1:nrow(df))head(df)
gene1 gene2 gene3 gene4 gene5 gene6 gene7 gene8 gene9 gene10 gene11
sample1 6 3 9 15 6 9 13 15 15 6 14
sample2 1 15 10 11 12 1 11 9 10 7 11
sample3 7 10 9 5 8 6 3 9 5 9 13
head(df1)
score1 score2 score3 score4 score5 score6 score7 score8 score9
sample1 81 95 70 75 83 88 87 61 54
sample2 61 63 85 69 59 83 55 82 60
sample3 81 56 85 59 67 74 56 74 59
数据处理2:计算相关性R和P
##进行相关性R和P值计算
dat <- cbind(df,df1)
dat1 <- as.matrix(dat)##需要为matrix格式
rescor <- rcorr(dat1,type="spearman")#计算相关性 ("pearson","spearman")
##R值数据处理
rescor_R <- rescor$r
rescor_R1 <- as.data.frame(rescor_R[colnames(df),colnames(df1)])##提取相关性R值
rescor_R1$GENE <- rownames(rescor_R1)##转换为长数据
rescor_long <- melt(rescor_R1, id.vars = c("GENE"))
##P值数据处理
rescor_P <- rescor$P
rescor_P1 <- as.data.frame(rescor_P[colnames(df),colnames(df1)])##提取相关性P值
rescor_P1$GENE <- rownames(rescor_P1)##转换为长数据
rescor_longp <- melt(rescor_P1, id.vars = c("GENE"))
colnames(rescor_longp) <- c("GENEP","variableP","valueP")
identical(rescor_long$GENE,rescor_longp$GENE)#[1] TRUE 前两列数据一致,进行合并
identical(rescor_long$variable,rescor_longp$variable)#[1] TRUE
res <- cbind(rescor_long,rescor_longp)
res1 <- res[,c(1:3,6)]
head(res1) GENE variable value valueP 1 gene1 score1 0.069495604 0.8056036 2 gene2 score1 0.084838789 0.7637076 3 gene3 score1 -0.277011560 0.3175398 4 gene4 score1 -0.108011195 0.7016025
数据处理3:添加细节
colnames(res1) <- c("GENE","SCORE","correlation","pvalue")
#绘图添加分列#
#添加一列,来判断pvalue值范围
res1$sign<-case_when(res1$pvalue >0.05~">0.05",
res1$pvalue <0.05 &res1$pvalue>0.01 ~"<0.05",
res1$pvalue <0.01 &res1$pvalue>0.001 ~"<0.01",
res1$pvalue <0.001 &res1$pvalue>0.0001~"<0.001",
res1$pvalue <0.0001 ~"<0.0001")
#添加一列来判断correlation的正负
res1$core<-case_when(res1$correlation >0 ~"positive",
res1$correlation<0 ~"negtive")head(res1) GENE SCORE correlation pvalue sign core 1 gene1 score1 0.069495604 0.8056036 >0.05 positive 2 gene2 score1 0.084838789 0.7637076 >0.05 positive 3 gene3 score1 -0.277011560 0.3175398 >0.05 negtive 4 gene4 score1 -0.108011195 0.7016025 >0.05 negtive
绘图
#主要是两种填充方式shape=21和shape=16
#主要是两种填充方式shape=21和shape=16
p <- ggplot(data=res1,aes(x=GENE,y=SCORE))+
#正相关的点(正负相关的点分开绘制)
geom_point(data=filter(res1,core=="positive"),
aes(x=GENE,y=SCORE,size=abs(correlation),fill=sign),shape=21)+
#负相关的点
geom_point(data=filter(res1,core=="negtive"),
aes(x=GENE,y=SCORE,size=abs(correlation),color=sign),shape=16)+
#自定义颜色fill为正相关颜色填充
scale_fill_manual(values=c("#FF6666","#FF9999","#FFCCCC","#FFCCCC","#CCCCCC"))+
#自定义颜色负相关颜色
scale_color_manual(values=c("#333366","#336699","#3399CC","#66CCFF","#CCCCCC"))+
#去除x轴和y轴
labs(x="",y="")+
#修改主题
theme_bw()+
theme(axis.text.x=element_text(angle=45,hjust=1),
axis.ticks.x=element_blank())+
#修改legend,title表示标题,order表示标题的顺序,override.aes来设置大小
guides(color=guide_legend(title="Negitive",order=1,override.aes=list(size=4)),
size=guide_legend(title="Spearman's p",order=2),#为相关性大小
fill=guide_legend(title="Positive",order=3,override.aes=list(size=4)))+#fill为正相关
labs(title = "Spearman's p Correlation")
p
dev.off()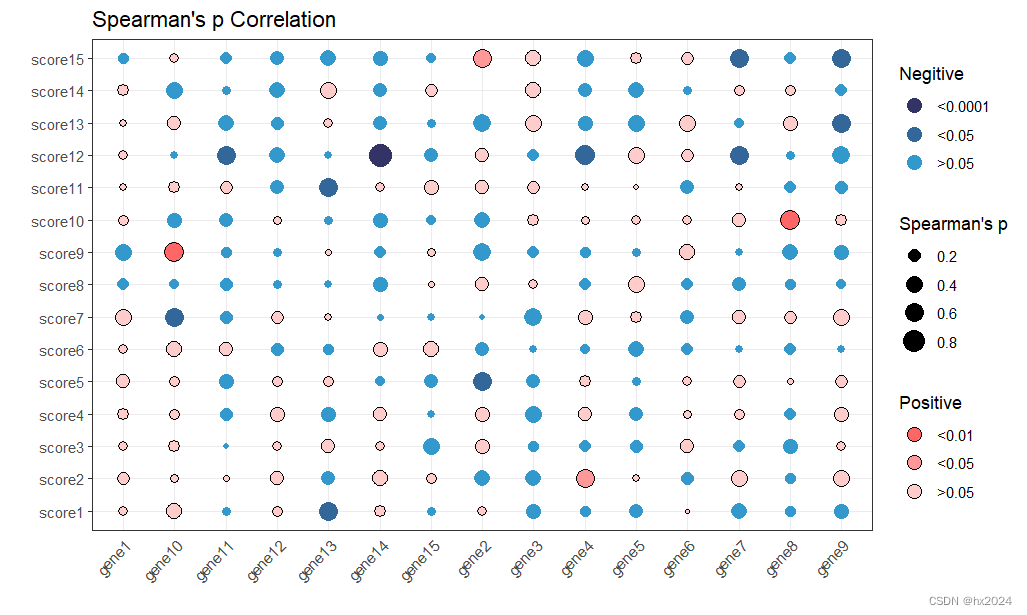







 本文介绍了如何使用R语言中的ggplot2和相关库(如ggrepel、corrplot等)创建相关性气泡图,展示了如何处理数据(生成dataframe、计算相关性R和P值),并用气泡大小和颜色编码展示相关系数和显著性。
本文介绍了如何使用R语言中的ggplot2和相关库(如ggrepel、corrplot等)创建相关性气泡图,展示了如何处理数据(生成dataframe、计算相关性R和P值),并用气泡大小和颜色编码展示相关系数和显著性。














 1649
1649











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








