0.介绍:
(Whole Slide Image, WSI)图像非常的大,处理起来比较麻烦,在深度学习中的病理切片图像大多数在 10万x10万分辨率,用平常的图像处理库没有办法读取,openslide 提供了一个很好的接口,这里介绍一个可用于处理大型病理切片图像的 python 库 (OpenSlide)。
1.Openslide安装:
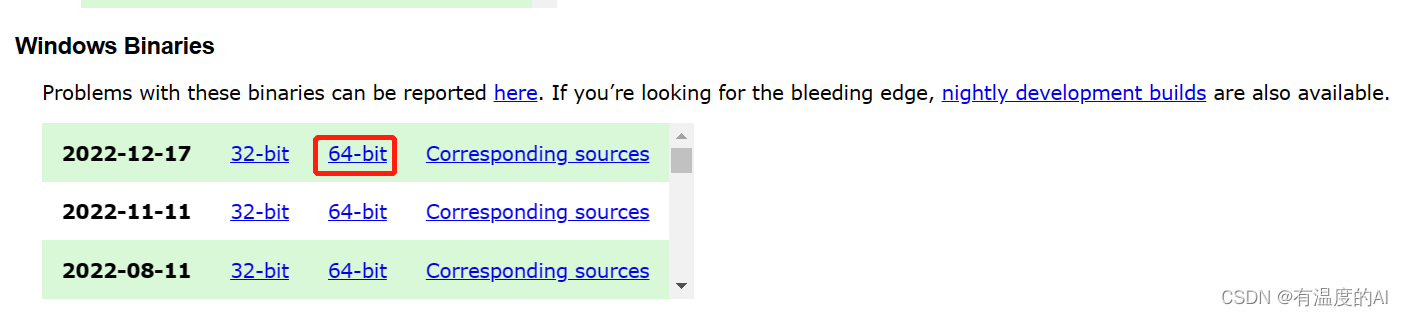
pip install openslide-python #终端输入OpenSlide官网下载
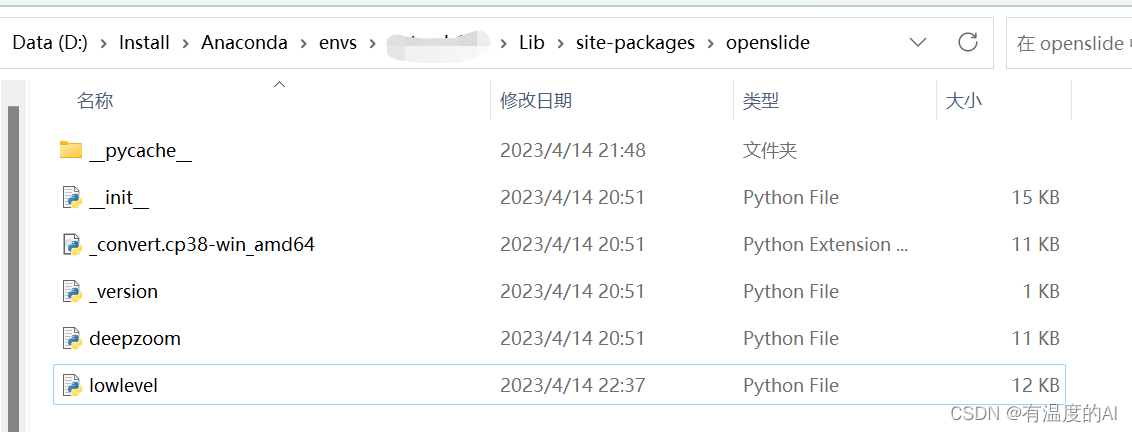
配置路径:打开Lib\site-packages下的lowlevel.py文件, 将解压的openslide-win64-20221217文件中的bin文件路径粘贴到如下
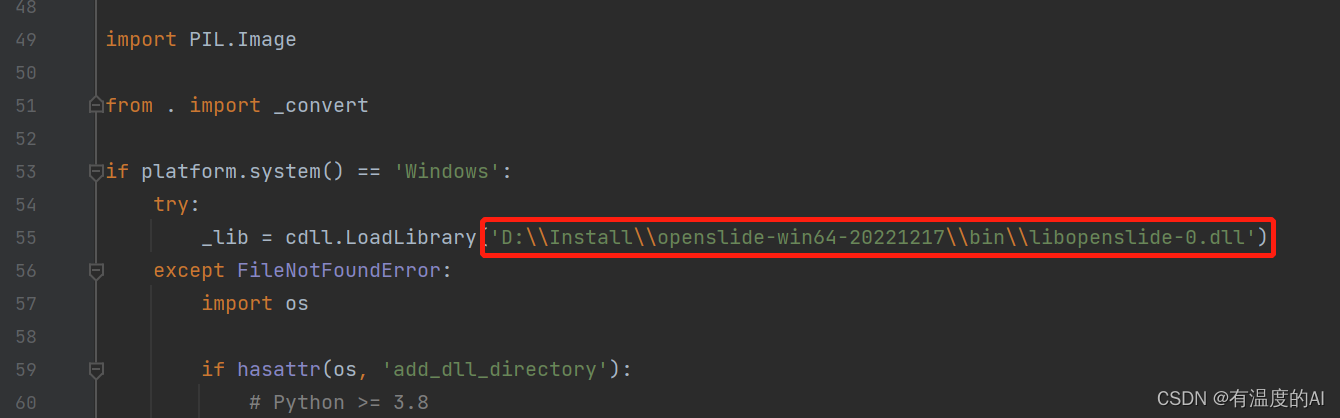
此时,进入python的控制台,输入import openslide,无任何异常提示表示opensldie安装好了!

如果这种方法不行,那就在导入openslide前需要导入相关路径!(简单粗暴)
路径为openslide-win64-20221217文件中的bin文件路径
import os
os.add_dll_directory('D:\\CNN\\openslide-win64-20221217\\openslide-win64-20221217\\bin')
import openslide2.切割图片
# 注意这里只能切割单张图片,不支持文件夹
import openslide
import numpy as np
import imageio #用于保存图片
slide = openslide.OpenSlide("D:\Visio_relatation\\111\\") #原始图片文件夹
dst_path = 'D:\Visio_relatation\\222\\' #保存图片文件夹
[m, n] = slide.dimensions # 得出高倍下的(宽,高)
print(m, n)
N = 1024
ml = N * m//N
nl = N * n//N
for i in range(0, ml, N): # 这里由于只是想实现剪切功能,暂时忽略边缘不足N*N的部分
for j in range(0, nl, N):
im = np.array(slide.read_region((i, j), 0, (N, N)))
imageio.imwrite(dst_path + '1' + 'a' + str(i) + 'a' + str(j) + '.png', im) # patch命名为‘x-y’
slide.close() # 关闭文件# 这里可以循环遍历文件夹中的所有图片
import os
import openslide
import numpy as np
import imageio # 用于保存图片
dst_path = 'D:\Visio_relatation\\222\\' #切割后保存地址
img_floder = r'D:\Visio_relatation\\111' #原tif图片文件夹
img_list = os.listdir(img_floder)
print(img_list)
for img_name in img_list:
name = img_name[:-4]
slide = openslide.OpenSlide(img_floder + "\\" + img_name)
[m, n] = slide.dimensions # 得出高倍级别下的(宽,高)
print(m, n)
N = 1024
ml = N * m//N
nl = N * n//N
for i in range(0, ml, N): # 这里由于只是想实现剪切功能,暂时忽略边缘不足N*N的部分
for j in range(0, nl, N):
im = np.array(slide.read_region((i, j), 0, (N, N)))
imageio.imwrite(dst_path + name + '_' + str(i) + '_' + str(j) + '.png', im) # patch命名为‘x-y’
slide.close() # 关闭文件注意:Openslide切出来的图片为4通道(RGBA),如果想得到三通道(RGB)图像请使用下面方法~~
import os
import numpy as np
import cv2 as cv
import PIL.Image as Image
from PIL import ImageFile
ImageFile.LOAD_TRUNCATED_IMAGES = True
Image.MAX_IMAGE_PIXELS = None
clip_img_path = 'D:/CNN/GHHE/zhimi/' # 切割后保存地址
img_floder = r'D:/CNN/GHHE/zhimi_tif/large' # 原tif图片文件夹
img_list = os.listdir(img_floder)
print(img_list)
def pixel_equal(image, x, y):
# 取图片像素点
piex = image.load()[x, y]
threshold1 = 250
threshold2 = 30
# 比较每个像素点的RGB值是否大于阈值
if piex[0] > threshold1 and piex[1] > threshold1 and piex[2] > threshold1:
return 1
elif piex[0] < threshold2 and piex[1] < threshold2 and piex[2] < threshold2:
return 2
else:
return 3
img_size = 1024
l = 0
right_num1 = 0 # 记录白色像素点个数
right_num2 = 0 # 记录黑色像素点个数
for img_name in img_list:
name = img_name[:-4]
# print(img_floder + '/' + img_name)
img0 = Image.open(img_floder + '/' + img_name) #PIL的形式可以显示size
img = np.array(Image.open(img_floder + '/' + img_name)) #array的形式才能显示shape
print(img0.size)
w, h = img0.size[0], img0.size[1]
# print(h, w)
for i in range(0, h, img_size):
for j in range(0, w, img_size):
end_i, end_j = i + img_size, j + img_size
cropped = img[i:end_i, j:end_j]
# img_orig = Image.fromarray(cv.cvtColor(cropped, cv.COLOR_BGR2RGB)) # bgr 转 rgb
img_orig = Image.fromarray(cropped) # 实现array到image的转换
to_image = Image.new('RGB', (img_size, img_size)) # 创建1024大小图片
to_image.paste(img_orig, (0, 0))
# print(to_image.size)
# to_image.save(clip_img_path + name + '_' + str(m) + '_' + str(n) + ".jpg")
for m in range(0, to_image.size[0]):
for n in range(0, to_image.size[1]):
if pixel_equal(to_image, m, n) == 1:
right_num1 += 1
elif pixel_equal(to_image, m, n) == 2:
right_num2 += 1
else:
continue
# print(right_num1, right_num2)
# 剔除不符合要求的图片
if right_num1 / (1024 * 1024) < 0.4 and right_num2 / (1024 * 1024) < 0.05:
to_image.save(clip_img_path + name + '_' + str(l) + ".jpg")
right_num1 = 0
right_num2 = 0
else:
right_num1 = 0
right_num2 = 0
l += 1
l = 0





















 4385
4385











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








