✅作者简介:热爱科研的Matlab仿真开发者,修心和技术同步精进,matlab项目合作可私信。
🍎个人主页:Matlab科研工作室
🍊个人信条:格物致知。
更多Matlab仿真内容点击👇
⛄ 内容介绍
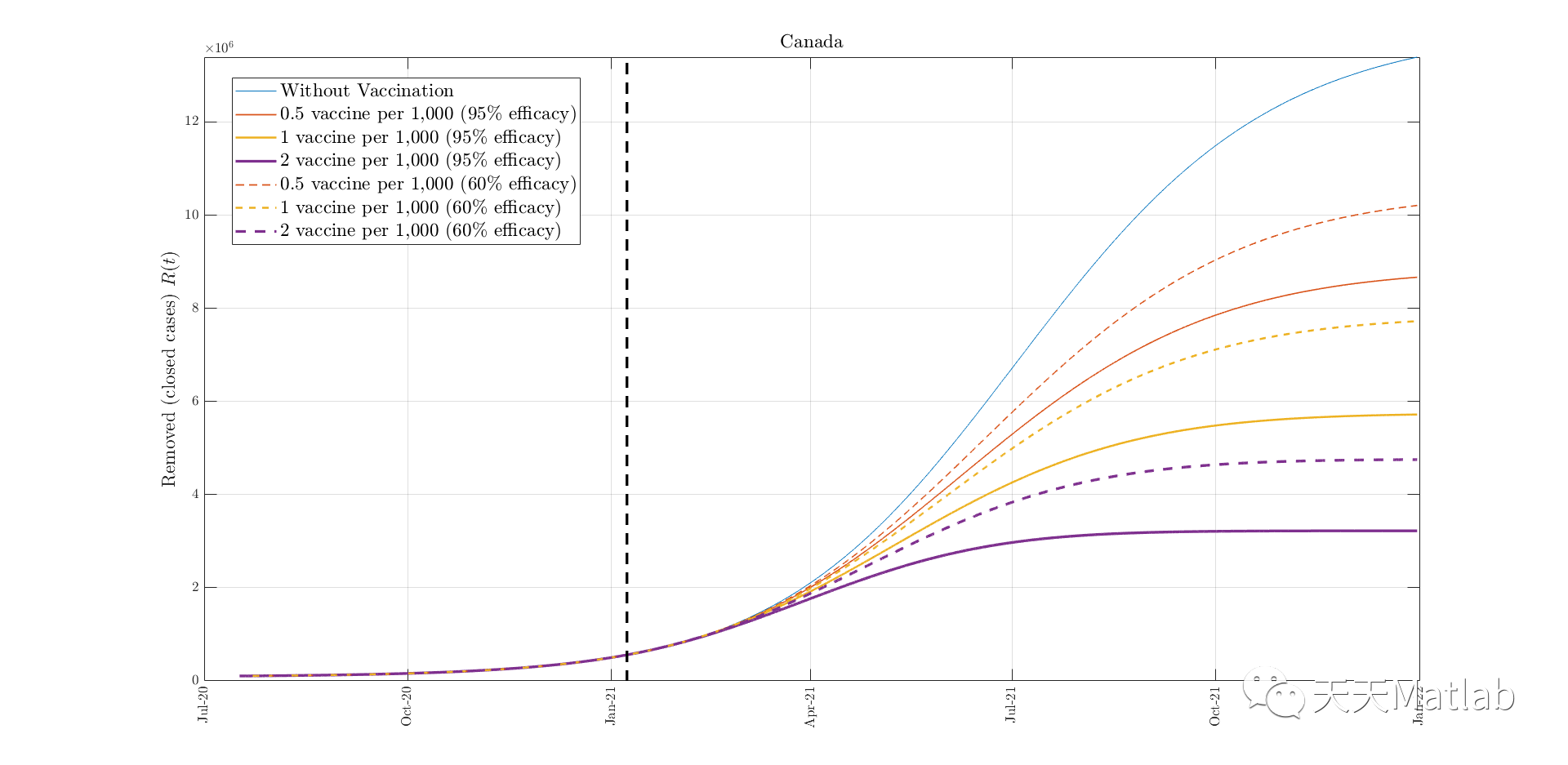
目的拟合并预测新型冠状病毒肺炎(COVID-19)疫情的发展趋势,为疫情防控提供科学依据。方法基于SIRV动力学模型,考虑COVID-19的传播机制、感染谱、隔离措施等,建立SIRV传播动力学模型。基于官方公布的每日确诊病例数进行建模,利用2020年1月20日至2月7日的报告疫情数据进行拟合。采用2月8-12日的数据评估预测效果,并进行疫情预测。结果该模型对加拿大的累计确诊病例数的过去10日拟合偏差<5%;
⛄ 部分代码
%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%
% MATLAB Code for the fitting of the SIRV model proposed
% in the following work:
%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%
%% Load data
clear all;clc;close all
population='Canada';
data = xlsread('COVID19-Data-Canada',population);
N=data(1,1);
I=data(:,2);
R=data(:,3);
D=data(:,4);
S=N*ones(size(I))-R-I;
startDate= datenum('17-Jul-2020'); %start day of the data
endDate=datenum('08-Jan-2021'); %end day of the data
tmonths=startDate:30:(endDate);
%% Fitting R0;
clear x y
yS=log(S/S(1));
yS=yS(1:end);
xS=R(1:end)-R(1)*ones(size(R(1:end)));
R0=-(xS\yS)*N;
%% Fitting for gamma
idx=1;
for t=2:length(I)
xR(idx)=trapz(1:t,I(1:t));
yR(idx)=R(t)-R(1);
idx=idx+1;
end
gamma=xR'\yR';
beta=gamma*R0;
%% Fitting for mu
xD=R(1:end)-R(1);
yD=D(1:end)-D(1);
mu=xD\yD;
yDfit=mu*xD;
%% Simulate the identified model
dt=1/24;
T=length(I)-1+dt;
[par,rms]=fminsearch(@(par)RMS_SIRV(par,I(1:end)/N,R(1:end)/N,T,dt),[beta gamma]);
beta=par(1);
gamma=par(2);
R0=beta/gamma;
[~,x2,x3,~] = SIRV_model(0,beta,gamma,I(1)/N,R(1)/N,0,T,dt,zeros(1,T/dt));
Dfit=mu*(x3-x3(1))*N+D(1);
t=startDate:dt:endDate;
% Plot the figures
figure(4)
plot(t,x2*N,'LineWidth',1.5);grid on; hold on; box on;
stem(startDate:endDate,I(1:end),'o')
ylabel('Infected $I(t)$','Interpreter','latex','fontsize',15)
title(population,'Interpreter','latex','fontsize',15)
legend({'Fitted Model','Real Data'},'Interpreter','latex','fontsize',15)
ax=gca;
ax.XTick=tmonths;
xtickangle(90)
datetick('x','mmm-yy')
set(gca,'TickLabelInterpreter','latex','fontsize',15)
xlim([startDate endDate])
ax.YAxis.Exponent = 3;
ySfit = -R0*xS/N;
axes('position',[.25 .6 .25 .25])
hold on;grid on;box on
plot(xS+R(1),ySfit,'-.','LineWidth',2);
scatter(xS+R(1),yS)
xlim([R(1) R(end)])
ylabel('$\log(S(t)/S(t_0))$','Interpreter','latex','fontsize',14)
xlabel('$R(t)$','Interpreter','latex','fontsize',14)
figure(5)
plot(t,x3*N,'LineWidth',1.5);grid on; hold on; box on;
stem(startDate:endDate,R(1:end),'o')
ylabel('Removed $R(t)$','Interpreter','latex','fontsize',15)
title(population,'Interpreter','latex','fontsize',15)
legend({'Fitted Model','Real Data'},'Interpreter','latex','fontsize',15)
ax=gca;
ax.XTick=tmonths;
xtickangle(90)
datetick('x','mmm-yy')
set(gca,'TickLabelInterpreter','latex','fontsize',15)
xlim([startDate endDate])
axes('position',[.25 .6 .25 .25])
hold on;grid on;box on
yRfit = gamma*xR;
plot(xR,yRfit+R(1),'-.','LineWidth',2);
scatter(xR,yR+R(1))
ylabel('$R(t)$','Interpreter','latex','fontsize',14)
xlabel('$\int_{t_0}^tI(\tau)d\tau$','Interpreter','latex','fontsize',14)
figure(6)
plot(t,Dfit,'LineWidth',1.5);grid on; hold on; box on;
stem(startDate:endDate,D(1:end),'o')
ylabel('Deaths $D(t)$','Interpreter','latex','fontsize',15)
title(population,'Interpreter','latex','fontsize',15)
legend({'Fitted Model','Real Data'},'Interpreter','latex','fontsize',15)
ax=gca;
ax.YAxis.Exponent = 3;
ax.XTick=tmonths;
xtickangle(90)
datetick('x','mmm-yy')
set(gca,'TickLabelInterpreter','latex','fontsize',15)
xlim([startDate endDate])
axes('position',[.25 .6 .25 .25])
hold on;grid on;box on;
plot(xD+R(1),yDfit+D(1),'-.','LineWidth',2);
scatter(xD+R(1),yD+D(1))
xlim([R(1) R(end)])
ylabel('$D(t)$','Interpreter','latex','fontsize',14)
xlabel('$R(t)$','Interpreter','latex','fontsize',14)
⛄ 运行结果
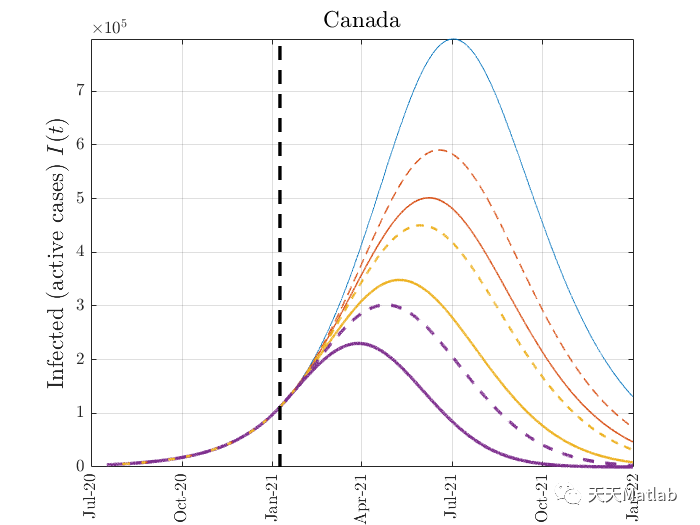
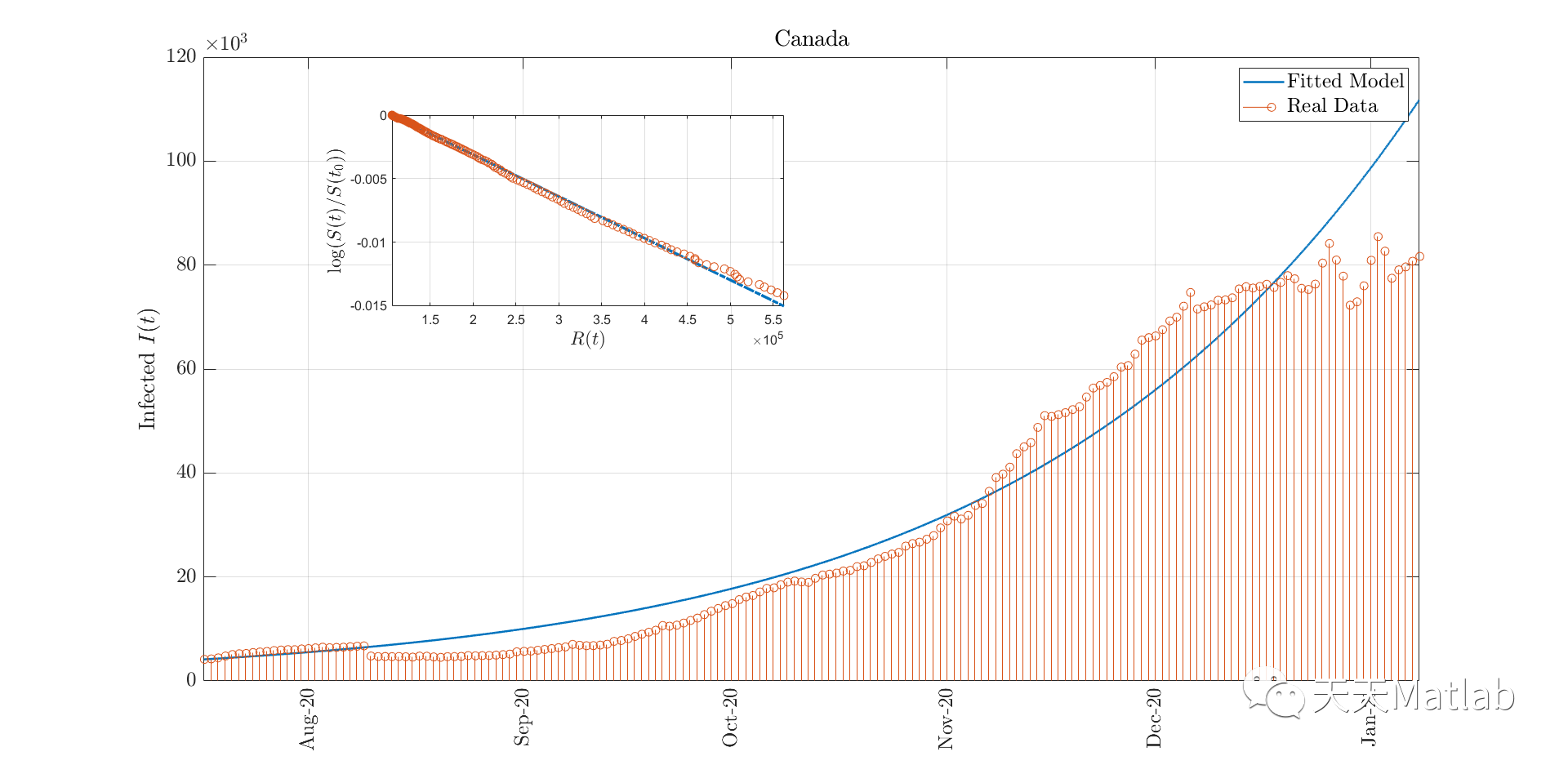
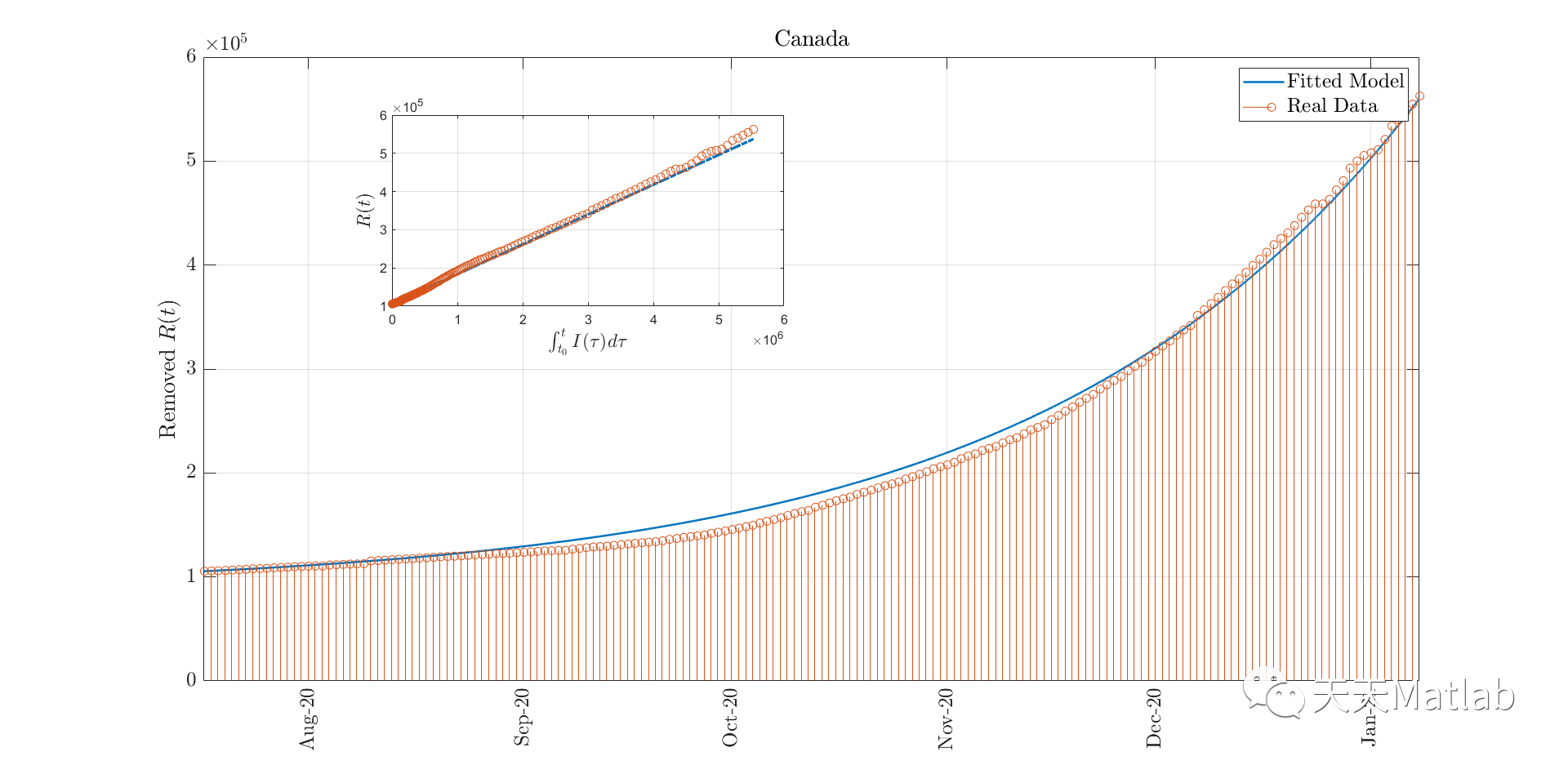
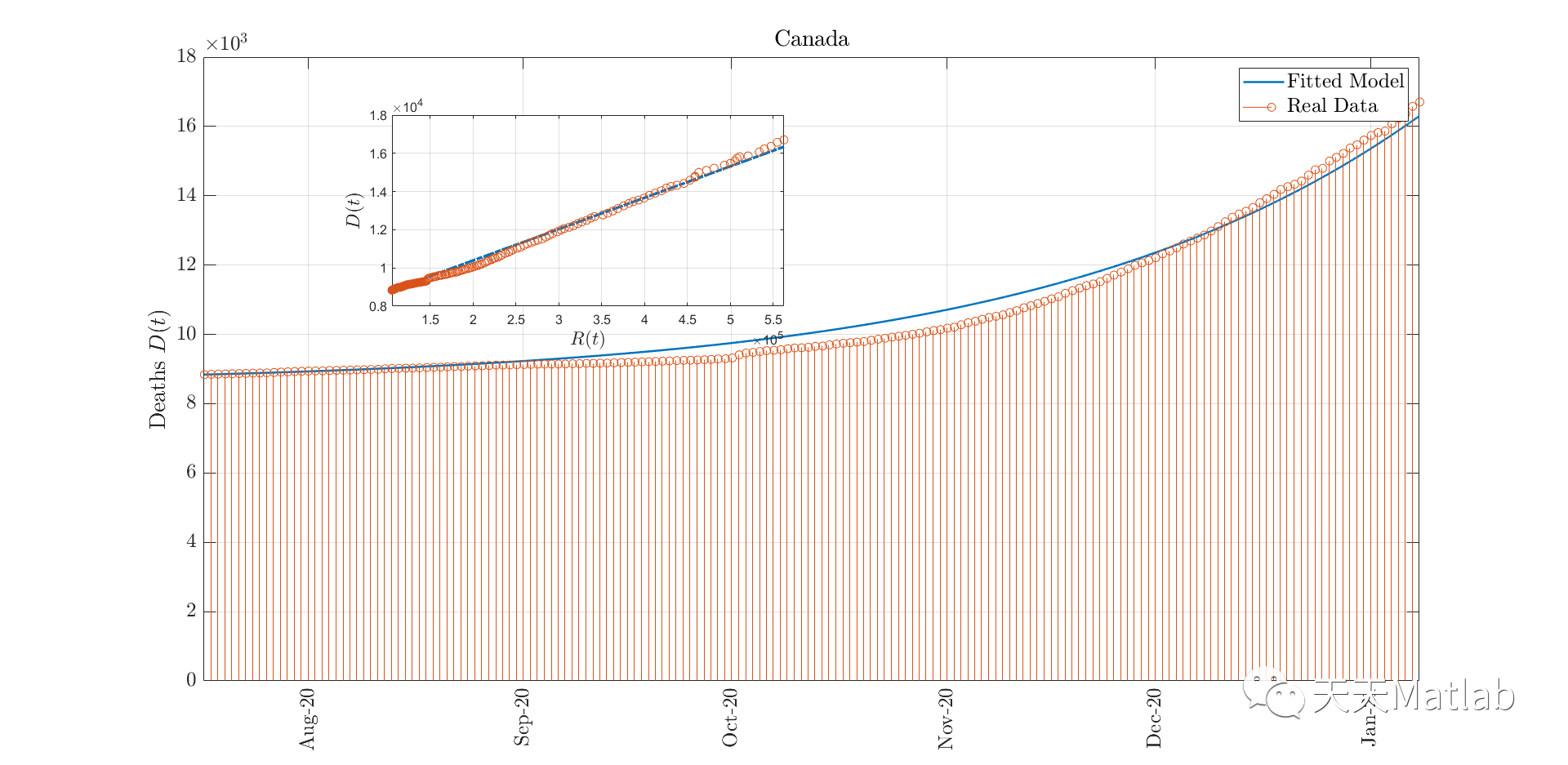
⛄ 参考文献
[1] 朱仁杰, 唐仕浩, 刘彤彤,等. 基于改进SIR模型的新型冠状病毒肺炎疫情预测及防控对疫情发展的影响[J]. 陕西师范大学学报:自然科学版, 2020, 48(3):6.
[2] 康观龙, 柳炳祥. 基于SIR模型的新型冠状病毒肺炎预测分析[J]. 中阿科技论坛(中英文), 2020, 000(006):P.151-153.
[3] 丁美丽, 彭润龙, 郭荣伟. 基于传染病模型的新冠肺炎传播问题研究[J]. 齐鲁工业大学学报, 2020, 34(6):9.
[4] 祁冬, 姜研, 姚传顺,等. 单能量技术结合ASIR-V算法在疑似新型冠状病毒肺炎患者行低剂量CT扫描中的应用价值[J]. 中国中西医结合影像学杂志, 2020, 018(005):453-457.
⛳️ 代码获取关注我
❤️部分理论引用网络文献,若有侵权联系博主删除
❤️ 关注我领取海量matlab电子书和数学建模资料






















 1358
1358











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








