噬菌体展示抗体库,第一步先是通过免疫相关模式动物,取得其PBMC或者脾器官;第二步反转录mRNA为cDNA,然后以cDNA作为模板进行pcr扩增,这里通常是做scfv较多,所以一般进行两步overlapping pcr或者设计同源臂直接同源重组。传统抗体库筛选是构建免疫细胞的杂交瘤细胞,然后进行体外扩增表达,优点是产生的抗体为天然抗体,缺点是构建的杂交瘤细胞库数量较少,一次需要多只免疫动物,而且需要培养细胞,换液等频繁操作,费时费力不说,还浪费钱。噬菌体展示优点就是快,库容量大,省事省力,节约资金,但抗体库构建的好不好跟很多原因相关,最直接的就是兼并引物设计不合理,不能覆盖所需要的抗体,扩增出其他类型的抗体等。
通过查阅已有噬菌体展示的兼并引物对,我们发现大部分引物遵从以下三个
规则(经验之谈,不作为噬菌体展示必须遵守的原则):
-
兼并核苷酸在一条引物中只出现一次(有些也有两个)
-
兼并核苷酸只包含两种核苷酸:M=A/C, Y=C/T, S=C/G, R=A/G, W=A/T,K=G/T
一、获取目的抗体序列
如可在NCBI中获取兔igg抗体,通常我们得到的igg的轻链和重链,同时我们需要注意的是重链通常有alpha、δ、e、gamma、μ等组成,而igg重链是gamma,所有抗体的轻链由λ或者k组成,但注意部分标记λ或者k的不一定指的是抗体序列。
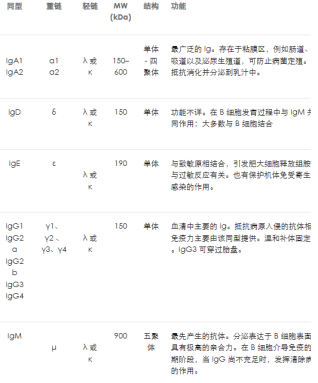
因此NCBI关键词搜索对于重链是rabbit IGG或者rabbit Ig Gamma,对于轻链则没有要求,因为λ或者k在所有抗体中都出现了,所以轻链关键词可为rabbit light或者rabbit KAPPA或者rabbit LAMBDA。所有的轻链和重链,可通过图左上角选择动物源的mRNA来减少序列输出量,简化程序计算时间。
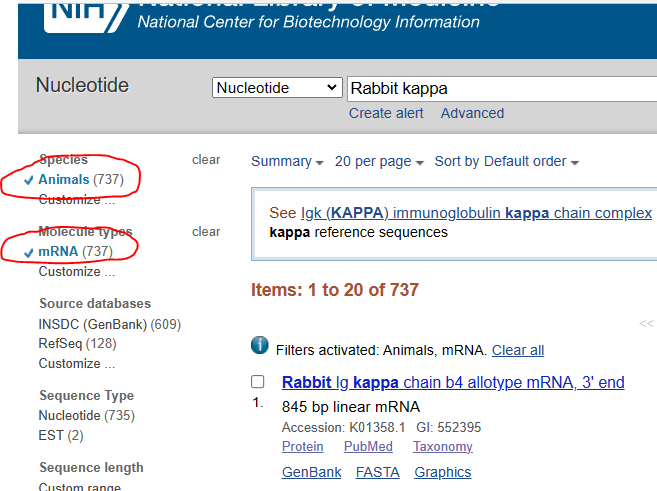
二、获取轻链和重链
heavy = []
heavyseq = []
i = 0
a = ""
name = []
f = open("E:\资料\课题\数据挖掘\兔scfv抗体引物设计\兔重链light and heavy DNA序列.txt","r")
f1 = open("E:\资料\课题\数据挖掘/兔scfv抗体引物设计/heavy DNA序列.txt","w")
for line in f.readlines():
if ">" in line:
i += 1
name.append(line.upper())
if i > 1:
if "RABBIT" in name[-2] or "ORYCTOLAGUS" in name[-2]:
if "HEAVY" in name[-2]:
if "IGG" in name[-2] or "GAMMA" in name[-2]:
if a not in heavyseq:
heavy.append([name[-2],a])
print(name[-2], a)
f1.write(name[-2] + a + "\n")
heavyseq.append(a)
a = ""
if ">" not in line:
a += line.replace("\n", "")
f.close()
f1.close()
light = []
lightseq = []
i = 0
a = ""
name = []
f = open("E:\资料\课题\数据挖掘/兔scfv抗体引物设计/兔重链light and heavy DNA序列.txt","r")
f1 = open("E:\资料\课题\数据挖掘/兔scfv抗体引物设计/light DNA序列.txt","w")
for line in f.readlines():
if ">" in line:
i += 1
name.append(line.upper())
if i > 1:
if "RABBIT" in name[-2] or "ORYCTOLAGUS" in name[-2]:
if "LIGHT" in name[-2] or "KAPPA" in name[-2] or "LAMBDA" in name[-2]:
if a not in lightseq:
light.append([name[-2],a])
print(name[-2], a)
f1.write(name[-2] + a + "\n")
lightseq.append(a)
a = ""
if ">" not in line:
a += line.replace("\n", "")
f.close()
f1.close()
三、通过bioedit等可视化软件观察引物截取片段附件的核苷酸保守性并截取相应的F端引物盒R端引物序列
heavyF = []
for a in heavy:
if "AGGAGTCCG" in a[1]:
m = a[1].index("AGGAGTCCG")
heavyF.append(a[1][m-10:m+11])
lightF = []
for a in light:
if "TGACCCAG" in a[1]:
m = a[1].index("TGACCCAG")
lightF.append(a[1][m-4:m+14])四、多条不同序列的兼并引物设计
from tqdm import tqdm
primer1 = heavyF
primer = []
for a in primer1:
if a not in primer:
primer.append(a)
M = ['A', 'C']
Y = ['C', 'T']
S = ['C', 'G']
R = ['A', 'G']
W = ['A', 'T']
K = ['G', 'T']
mutipre1 = []
mutipre2 = []
for a in tqdm(range(len(primer) - 1)):
for b in range(a + 1, len(primer)):
if (primer[a] != '') & (primer[b] != ''):
print(primer[a], primer[b])
for c in range(len(primer1[0])):
if (primer[a] != '') & (primer[b] != ''):
print(primer[a][c], primer[b][c])
if primer[a][0:c] + primer[a][c + 1:] == primer[b][0:c] + primer[b][c + 1:]:
print(primer[a][0:c] + primer[a][c + 1:])
if (primer[a][c] in S) & (primer[b][c] in S):
mutipre1.append(primer[a][0:c] + 'S' + primer[a][c + 1:])
print(primer[a][0:c] + "S" + primer[a][c + 1:])
primer[a] = ''
primer[b] = ''
elif (primer[a][c] in M) & (primer[b][c] in M):
mutipre1.append(primer[a][0:c] + 'M' + primer[a][c + 1:])
print(primer[a][0:c] + "M" + primer[a][c + 1:])
primer[a] = ''
primer[b] = ''
elif (primer[a][c] in Y) & (primer[b][c] in Y):
mutipre1.append(primer[a][0:c] + 'Y' + primer[a][c + 1:])
print(primer[a][0:c] + "Y" + primer[a][c + 1:])
primer[a] = ''
primer[b] = ''
elif (primer[a][c] in M) & (primer[b][c] in R):
mutipre1.append(primer[a][0:c] + 'R' + primer[a][c + 1:])
print(primer[a][0:c] + "R" + primer[a][c + 1:])
primer[a] = ''
primer[b] = ''
elif (primer[a][c] in W) & (primer[b][c] in W):
mutipre1.append(primer[a][0:c] + 'W' + primer[a][c + 1:])
print(primer[a][0:c] + "W" + primer[a][c + 1:])
primer[a] = ''
primer[b] = ''
elif (primer[a][c] in K) & (primer[b][c] in K):
mutipre1.append(primer[a][0:c] + 'K' + primer[a][c + 1:])
print(primer[a][0:c] + "K" + primer[a][c + 1:])
primer[a] = ''
primer[b] = ''
for a in primer:
if a != '':
mutipre2.append(a)
mutipre3 = mutipre1 + mutipre2
mutipre3






















 282
282











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








