生物数据库的定义
生物数据库是被组织起来的大量生物数据,这些数据通过计算机可以被方便的访问、管理及更新。
生物数据库的分类
核酸数据库
一级核酸数据库:存储的是通过各种科学手段得到的最直接的基础数据。如测序获得的核酸序列等。
二级核酸数据库:是通过对一级数据中数据的分析整理归纳注释构建的具有特殊生物学意义和专门用途的数据库。如从三大核酸数据库和基因组数据库中提取并加工出的果蝇和蠕虫数据库。
蛋白质数据库
一级蛋白质数据库:存储的是通过各种科学手段得到的最直接的基础数据。如X射线衍射法获得的蛋白质三级结构等。一级蛋白质数据库又分为一级蛋白质序列数据库和一级蛋白质结构数据库。
二级蛋白质数据库:是通过对一级数据中数据的分析整理归纳注释构建的具有特殊生物学意义和专门用途的数据库。如根据蛋白质三级结构数据库中的结构信息分析统计出的蛋白质结构分类数据库。
专用数据库
如文献数据库PubMed等。
文献数据库PubMed
PubMed拥有超过两百四十万的生物医学文献。它们来源于MEDLINE(生物医学文献数据库)、生命科学领域学术杂志以及在线的专业数据。这些文献部分提供全文链接。
一级核酸数据库
GenBank
GenBank,ENA与DDBJ共同构成国际核酸序列数据库合作联盟(INSDC)。通过INSDC,三大核酸数据库的信息每日相互交换、更新汇总,这使得他们几乎在任何时候都享有相同的数据。

DEFINITION:简短的定义,标题。
ACCESSION:检索号在数据库中是唯一且不变的,即使数据库提交者改变了数据内容。
VERSION:版本号的格式是“检索号.版本代号”。
KEYWORDS:能够大致描述该条目的几个关键词,可用于数据库检索。
SOURCE:基因序列所属物种的俗名。
基因组数据库:Ensemble
Ensemble由欧洲生物信息学研究所(EBI)和美国桑格研究院(Sanger Institute)合作开发。Ensemble收入了各种动物的基因组,特别是哪些离我们人类近的动物(脊椎动物)。这些基因组的注释都是通过配套开发的软件自动添加的。
微生物宏基因组数据库:JCVI
美国基因组研究所(TIGR)致力于微生物基因组的研究,也有部分动物基因组项目。它是克莱格·凡特研究所的一部分,自1995年成立之初的两个基因组,至今已拥有超过700个基因组,而且还将更多。TIGR是NCBI基因组资源的有力补充,因为它不仅拥有已完成测序的基因组,还有那些测序中的基因组信息。
二级核酸数据库
RefSeq数据库
参考序列数据库,是通过自动及人工精选出的非冗余数据库,包括基因组序列、转录序列和蛋白质序列。
dbEST数据库
表达序列标签数据库,包含来源于不同物种的表达序列标签(EST)。
Gene数据库
以基因为记录对象,为用户提供基因序列注释和检索服务,收录了来自5300多个物种的430万条基因记录。
ncRNAdb
非编码RNA数据库,提供非编码RNA的序列和功能信息。包含来源于99中细菌,古细菌和真核生物的3万多条序列。
miRbase
主要存放已发表的microRNA序列和注释。可以分析microRNA在基因组中的定位和挖掘microRNA序列间的关系。
一级蛋白质数据库
UniProt数据库(蛋白质序列数据库)简介
2002年,SIB和EMBL-EBI的swissprot及TrEMBL管理组与Georgetown大学医学中心(GUMC)及美国生物医学研究基金会(NBRF)的PIR管理组成立联合蛋白质数据库协作组,管理UniProt联合蛋白质序列数据库。
UniPort三个层次数据库:
- UniParc:收录所有UniProt数据库子库中的蛋白质序列,量大、粗糙。
- UniRef:归纳UniProt几个主要数据库并将重复序列去除后的数据库。
- UniProtKB:由详细注释并与其他数据库有链接的数据库,分为UniProtKB/Swiss-Prot和UniProtKB/TrEMBL。
UniProtKB注释解读

Function:提供与蛋白质功能相关的信息。
Names&Taxonomy:蛋白质的名字,所属物种的分类学信息等基本信息。
SubcellularLocation:提供蛋白质亚细胞定位的信息。
Pathology&Biotech:提供蛋白质突变或缺失导致的疾病及表型信息。
PTM/Processiing:提供蛋白质翻译后修饰(Post-translational modification,PTM)或翻译后加工的相关信息。
Expression:提供了基因在mRNA水平上的表达信息,或者在细胞中蛋白质水平上的表达信息,或者在不同器官组织中的表达信息。
Interaction:提供了蛋白质之间相互作用的信息。
Structure:提供蛋白质二级结构和三级结构信息。
Family&Domains:提供蛋白质家族及结构信息。
Sequence:提供蛋白质氨基酸序列信息。
Cross-references:列出所有通往其他含有该蛋白质信息的数据库的链接。
Publications:列出了有关这个蛋白质已发表的所有文献信息。
Entry information:提供有关这条数据库记录的录入信息。
Miscellaneous:杂项,包含任何无法归入前面几项的内容。
Similar Proteins:在UniRef数据库里找到与该蛋白质在序列水平上相似的其他蛋白质,并按一定相似度分组。
PDB数据库(蛋白质结构数据库简介)简介

二级蛋白质数据库
结构域家族数据库Pfam
Pfam数据库是一个蛋白质结构域家族的集合。
蛋白质一般是由一个或多个功能区域组成,这些功能区域通常称作结构域。在不同的蛋白质中结构域以不同的组合出现,形成了蛋白质的多样性。识别出蛋白质中的结构域对于了解蛋白质的功能由重要意义。
结构分类数据库CATH
CATH由伦敦大学于1993年开发和维护的。该数据库的名字CATH分别是数据库中四种结构分类层次的首字母,即:
- 蛋白质的种类(class ,C)
- 蛋白质二级结构的架构(architecture ,A)
- 蛋白质的拓扑结构(topology[fold] ,T)
- 蛋白质同源超家族(homologous superfamily ,H)
数据库的分类对象是PDB中存储的那些已测定结构的蛋白质结构域,分类时既使用计算机程序,也进行人工检查。
CATH-Gene3D还为超过500万条来自公共数据库的蛋白质序列进行了结构分类预测。Gene3D里的信息为绝大多数还未解析3D结构的蛋白质提供了重要的功能研究依据。
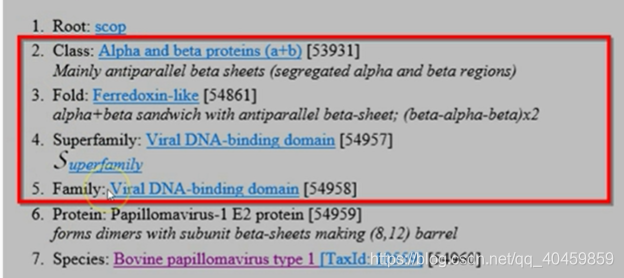
结构分类数据库SCOP2
英国医学研究委员会(MRC)的分子生物学实验室和蛋白质工程研究中心于2014年2月正式发布了蛋白质结构分类数据库SCOP的全面升级版SCOP2。该数据库在搜索、整理、分析PDB数据中已知的蛋白质三维结构的基础上,详细描述了已知结构的蛋白质在结构、进化事件与功能类型三个方面的关系。数据库的构建除了使用计算机程序外,主要依赖人工验证。SCOP2把SCOP中仅基于蛋白质结构的树状等级分类系统发展成为单向非循环网状分类系统。
SCOP2分类基于四个层次,从顶部到底部分别为:
- 类(Class)
- 家族(Fold)
- 超家族(Super family)
- 折叠(Family)
专项数据库
京都基因与基因组百科全书KEGG
京都基因与基因组百科全书(KEGG)是关于基因、蛋白质、生化反应以及通路的综合生物信息数据库,由多个子库构成。

图表 5 KEGG的子库
人类孟德尔遗传在线OMIM
人类孟德尔遗传(MIM)是一个将遗传病分类并链接到相关人类基因组中的数据库,它的在线版本是人类孟德尔遗传在线(OMIM)。OMIM为临床医生和科研人员提供了权威可信的关于遗传病及相关疾病基因位点的详细信息。
























 1949
1949

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








