使用大规模数据注释和深度学习对具有人类水平性能的组织图像进行全细胞分割
原文见:https://www.nature.com/articles/s41587-021-01094-0

摘要
组织成像数据分析的主要挑战是细胞分割,即识别图像中每个细胞的精确边界。为了解决这个问题,我们构建了TissueNet,这是一个用于训练分割模型的数据集,包含超过100万个手动标记的细胞,比之前发布的所有分割训练数据集都多一个数量级。我们使用TissueNet来训练Mesmer,这是一种能够进行深度学习的分割算法。我们证明了Mesmer比以前的方法更准确,可以推广到TissueNet中组织类型和成像平台的完全多样性,并实现了人类水平的性能。Mesmer能够自动提取关键的细胞特征,如蛋白质信号的亚细胞定位,这在以前的方法中是具有挑战性的。然后,我们对Mesmer进行了调整,以利用高度复用的数据集中的细胞谱系信息,并使用该增强版本来量化人类妊娠期间的细胞形态变化。所有代码、数据和模型都作为社区资源发布。
绪论
理解组织中存在的结构和功能关系是基础和转化研究前沿的一项挑战。多重成像的最新进展扩大了可同时定量的转录物和蛋白质的数量,为大规模分析人类组织样本开辟了新的途径。交叉结合在多方面开展,如人类肿瘤图谱网络、人类生物分子图谱计划和人类细胞图谱,正在使用这些方法来全面表征细胞在人体中的位置、功能和表型。然而,大规模分析和解释这些数据集所需的工具还不存在。最明显的例子是缺乏用于定位图像中单个细胞的通用算法。与流式细胞术或单细胞RNA测序不同,组织成像是用完整的标本进行的。因此,为了提取单细胞数据,必须在称为细胞分割的过程中将每个像素分配给一个细胞。由于通过该过程提取的特征是下游分析的基础,因此该阶段的不准确度可能对解释图像数据产生深远影响。实现精确、自动化的细胞分割的难度在很大程度上取决于不同组织类型的细胞形状、大小和密度的差异。为应对这一挑战而开发的机器学习方法,在组织成像数据方面存在不足。一个常见的缺陷是需要进行手动的、特定于图像的调整,以产生有用的分割。鉴于组织成像实验的规模越来越大,全自动化的分割算法变得尤为重要。
计算机视觉的深度学习算法越来越多地用于生物图像分析的各种任务,包括细胞核和细胞分割。这些算法能够实现高精度,但需要大量的注释训练数据。为细胞分割生成标签数据需要大量时间,因此,现有数据集一般都只有中等规模(104–105个注释)。此外,大多数公共数据集,注释的是细胞核的位置,而不是整个细胞的位置,这意味着在这些数据集上训练的模型只能执行细胞核分割,而不能进行细胞分割。因此,缺乏可用的数据,再加上在生命科学界部署预先训练的模型的困难,阻碍了全细胞分割的进展。

图1。人在环的方法能够对大型图像集合进行可扩展的像素级注释。a、 这种方法有三个阶段。在第1阶段,将从头开始创建注释以训练模型。在第二阶段,通过初步模型提供新数据以生成预测。这些预测被注释者用作校正的起点。随着更多的图像被校正,模型得到了改进,从而减少了错误数量,提高了注释新数据的速度。在第3阶段,在没有人为校正的情况下运行精确的模型。b、 TissueNet的核注释和全细胞注释比之前发布的所有数据集都多。c、 TissueNet中每个成像平台的细胞注释数量。d、 TissueNet中每种组织类型的细胞注释数。e、 创建TissueNet所需的注释时间小时数。CODEX,通过索引进行共同检测;循环免疫荧光;MIBI-TOF,按飞行时间进行多路复用离子束成像;MxIF,多重免疫荧光;IMC,成像质谱仪。
在这里,我们试图通过创建一种自动化、简单和可扩展的细胞核和全细胞分割算法来缩小这些差距,该算法可以在各种组织类型和成像平台上准确执行。开发该算法需要两项创新:(1)生成大量像素级训练数据的可扩展方法,以及(2)使用这些数据实现人类级性能的集成深度学习算法。为了应对第一个挑战,我们开发了一种众包、人在环的方法来分割细胞,其中人和算法协同工作以产生准确的注释(图第1a段)。我们使用这个方法创建了TissueNet,这是一个由>100万个配对的全细胞和细胞核注释组成的综合分割数据集。TissueNet包含的细胞核标签数量是之前所有发布数据集总和的两倍,全细胞标签数量是16倍。为了应对第二个挑战,我们开发了Mesmer,这是一种支持深度学习的算法,用于组织成像数据的可扩展、用户友好的分割。为了能够被科学界广泛使用,我们利用开源软件库集合DeepCell创建了一个使用Mesmer的网络界面,以及ImageJ和QuPath的插件。我们已经在许可证下提供了所有代码、数据和经过训练的模型作为社区资源,为将这些现代数据驱动的方法应用于广泛的研究挑战奠定了基础。
构建TissueNet的人在环方法用于细胞分割的现有注释数据集在范围和规模上受到限制(图1b)。这种限制很大程度上是由于用于构建它们的线性、时间密集型方法,这需要手动标定图像中每个细胞的边界。因此,我们实施了一种分三阶段的方法来创建TissueNet。在第一阶段,人类注释专家在80张图像中勾勒出每个细胞的边界。这些标记的图像用于训练初步模型(图1a,左侧;方法)。然后,该过程进入第二阶段(图1a,中间),在第二阶段,图像首先通过模型生成预测注释,然后发送给众包注释器以更正错误。在将校正后的注释添加到训练数据集之前,专家对其进行了最终检查。当编译了足够多的新数据后,训练了一个新的模型,并重复第二阶段。每次迭代产生更多的训练数据,从而提高了模型的准确性,减少了需要手动纠正的错误,并降低了注释的边际成本。这种良性循环一直持续到模型达到人类水平的性能。这种良性循环一直持续到模型达到人类水平的性能。在这一点上,我们过渡到了第三阶段(图1a,右)。在阶段3,模型在没有人工帮助的情况下运行,以产生高质量的预测。
人在环方法需要针对当前任务进行优化的专用软件。尽管之前的工作使用了人在环的方法来创建分割数据集,但现有的工具并没有针对众包或校正大量组织图像数据进行优化。因此,我们开发了DeepCell Label,这是一个基于浏览器的图形用户界面,用于编辑组织图像中现有的细胞注释(扩展数据图1a;方法)。DeepCell标签由可扩展的云后端支持,该后端可根据需求动态调整服务器数量(扩展数据图1b)。使用DeepCell标签,我们训练了来自多个众包平台的注释器来识别整个细胞和核边界。为了进一步简化我们的注释工作流程,我们将DeepCell Label集成到一个管道中,使我们能够准备和提交图像进行注释,让用户对这些图像进行注释并下载结果。图像和产生的标签用于训练和更新我们的模型,完成循环(扩展数据图1c;方法)。
由于我们的人在环数据标记方法的可扩展性,TissueNet大于所有先前发表的数据集的总和(图1b),具有130万个全细胞注释和120万个核注释。TissueNet包含来自六个成像平台的2D数据(图1c),九个器官(图1d),并包括组织学正常和病变组织(例如,肿瘤切除)。TissueNet还包括三个物种,分别来自人类、老鼠和猕猴。构建TissueNet需要超过4000人时,相当于近2人年的全职工作(图1e)。
Mesmer
2.1Mesmer是一种用于精确全细胞分割的深度学习算法
为了满足细胞分割对准确性和速度的要求,我们创建了Mesmer,这是一种基于深度学习的核细胞和全细胞分割算法。Mesmer的模型由连接到特征金字塔网络的ResNet50骨干组成,该网络具有四个预测头(两个用于核分割,两个用于全细胞分割),这些预测头连接到金字塔的顶部(扩展数据图2a;方法)。Mesmer的输入是定义每个细胞细胞核的细胞核图像(例如DAPI)和定义每个细胞形状的膜或细胞质图像(例如CD45或E-钙粘蛋白)(图2a)。这些输入被归一化50(以提高鲁棒性),平铺成固定大小的补丁(以允许处理任意维度的图像),并被馈送到深度学习模型。然后,模型输出被直到51,以产生对图像中每个细胞核和细胞的质心和边界的预测。质心和边界预测被用作分水岭算法的输入,以创建图像中每个细胞核和每个细胞的最终实例分割掩码(方法)。
为了评估Mesmer的准确性,我们对之前发表的预训练算法以及在TissueNet上重新训练的深度学习模型进行了全面的基准测试。这些比较使我们能够了解深度学习方法和训练数据对整体准确性的相对贡献。我们首先将Mesmer的性能与两种预处理算法进行了比较:FeatureNet26,我们之前使用它执行核分割,然后扩展分析一组乳腺癌症样本;Cellpose28,一种最近发布的显微镜数据全细胞分割算法。总体而言,我们观察到Mesmer(F1=0.82)的精度高于FeatureNet(F1=0.63)和Cellpose(F1=0.41)(图2b和扩展数据图2c–f)。
使用Dawson Trick Litzkow(DTL)纤维电极从参与者的左眼记录ERG(Dawson等人(1979);爱尔兰都柏林Di agnosys Vision有限公司)。虽然OPM传感器的数据是通过FieldLine采集系统采集的,但ERG活性是通过Elekta Triux MEG系统(MEGIN Oy,赫尔辛基,芬兰)的生物电位放大器记录的。两个测量值均以1000Hz采样。将光电二极管安装在屏幕上,用于刺激开始定义,并使用OPM和MEG系统记录数据流,以离线同步记录。
然后,我们将Mesmer的性能与在TissueNet上训练的一系列监督分割方法进行了比较。FeatureNet、RetinaMask和Ilastik即使在TissueNet上训练,也没有达到与Mesmer相当的性能(图2b和扩展数据图2c–f)。相比之下,Cellpose和StarDist在TissueNet上训练时获得了与Mesmer相当的性能(图2b)。最后,我们将Mesmer与经过训练的模型进行了比较,该模型用于执行核分割,然后进行像素扩展(现有核分割算法的一种常见方法16,55–57,用于近似整个细胞),并发现Mesmer实现了优异的性能(扩展数据图2g)。这些比较不受我们选择的指标的影响,因为我们在召回率、精确度和Jaccard指数方面观察到了类似的趋势(扩展数据图2c–f)。
除了准确性上的差异外,我们基准测试的算法在速度上也有很大差异。尽管模型容量显著增加,但Mesmer仅比FeatureNet慢13%,并且比Cellpose快20倍(图2b)。RetinaMask和Illastik的处理时间也很慢(图2b)。这些速度上的差异主要是由于各种方法之间的后处理差异,这占了大部分计算时间(扩展数据图2b)。
为了可视化Mesmer与已发表的预训练版本FeatureNet和Cellpose之间的性能差异,我们使用所有三种算法来分割结直肠癌的图像(图2c)。我们将分割预测与地面实况数据进行了比较,通过预测区域与地面实况区域的比率对每个单元进行着色(图2d)。总体而言,Mesmer更有效地捕捉到了图像中每个细胞的真实大小(图2e)。相比之下,FeatureNet很难捕捉到每个细胞的真实大小,这是预期的,因为该模型通过执行核分割和扩展来近似细胞形状。与较低的召回分数一致(扩展数据图2c),Cellpose未能识别图像中的大部分细胞(图2e),这可能是由于用于训练Cellpose的数据中组织图像相对稀缺。
接下来,我们检查了Mesmer在一系列组织类型上的分割预测(图2f)。梅斯默的错误是无偏的,有相同数量的细胞过大或过小。此外,Mesmer的误差与细胞的真实大小无关(扩展数据图2g)。相比之下,依赖于核分割和扩展的方法往往高估了大多数小细胞的大小,而低估了大多数大细胞的大小(扩展数据图2g)。总的来说,这个基准测试表明Mesmer比以前的分割方法有了显著的进步。
2.2Mesmer实现了人类级的分割性能
到目前为止,我们的结果(图2f和图3a)表明,在没有手动调整的情况下,Mesmer在所有TissueNet中都表现良好。然而,考虑到细胞形态和图像特征可能因器官部位、疾病状态和成像平台的不同而不同,与在所有TissueNet上训练的模型相比,根据单个平台或单个组织类型的数据训练专家模型可能会带来更优的性能。为了评估Mesmer的可推广性,我们将性能与使用组织或平台特定的TissueNet子集训练的模型进行了比较。我们观察到Mesmer和专家模型之间的性能相当(图第3b、c段)。接下来,我们试图评估专家模型在对训练期间未看到的数据进行评估时的表现。一般来说,当对训练期间未发现的数据类型进行评估时,专家模型的性能较差(扩展数据图第3b段)。数据集大小也起着重要作用,因为在TissueNet的小子集上训练的模型不如在整个数据集上训练的那些模型(扩展数据图3d–h)。


图2|Mesmer在组织的多路复用图像中提供精确的细胞核和全细胞分割。a、 Mesmer分割流水线中的关键步骤示意图。b、 Mesmer和先前发表的模型的速度与准确性比较,以及我们在TissueNet(TN)上重新培训的架构。准确性是通过TissueNet测试集中预测的分割和基本事实标签之间的F1分数(方法)来衡量的,其中0表示不一致,1表示完全一致。c、 结直肠癌代表性图像的彩色叠加。d、 插图显示了c中一个小区域的基本事实(顶部)和预测(中间)标签,以及分割精度的视觉表示(底部)。每个单元的预测分割由预测区域和地面实况区域之间的比率的log2着色。预测的过大的单元格为红色,而预测的过小的单元格为蓝色。e、 c中图像的地面实况分割标签,以及来自Mesmer和先前发表的模型的预测标签,每个标签都由d中的log2着色。从视觉上看,Mesmer提供了比以前的方法更好的性能。f、 梅斯默概括了组织类型、成像平台和疾病状态。为每个图像提供F1分数。DCIS,导管原位癌。比例尺,50μm。
这里使用的模型准确性指标将人工注释的数据视为金标准,但即使是专家注释者也可能相互不同意。因此,我们将Mesmer的分割预测与五位独立的专家人类标注者的预测进行了比较。我们对所有成对的人类标注者进行了评估,将一个注释器作为“金标准”,另一个作为预测。然后,我们将Mesmer的预测与这五位注释者的预测进行了比较。我们检测到人与人之间和人与梅斯默之间的F1评分之间没有显著差异(P=0.93)(图3d),表明Mesmer的表现与人类注释者不相上下。
为了进一步评估Mesmer相对于人类的表现,我们招募了四名病理学家对人类注释者和Mesmer的分段进行盲法评估。向每位病理学家展示了包含Mesmer预测和人类注释的成对图像(图第3e段)。当综合评估时,病理学家对Mesmer的预测和专家注释者的预测进行了等效的评级(图3f)。按组织类型细分评估,我们观察到病理学家的评估只有适度的差异,在某些组织中,Mesmer的表现略好于人类标注者,在其他组织中,注释器的表现稍好。总之,前面的分析表明,Mesmer可以以人类水平的性能进行全细胞分割。Cellpose和StarDist可能会获得类似的结果,因为当在TissueNet上训练时,它们的性能与Mesmer相当(图2b)。据我们所知,以前的细胞分割算法在组织数据方面没有达到与人类性能相当的效果。
为了进一步评估Mesmer相对于人类的表现,我们招募了四名病理学家对人类注释者和Mesmer的分段进行盲法评估。向每位病理学家展示了包含Mesmer预测和人类注释的成对图像(图第3e段)。当综合评估时,病理学家对Mesmer的预测和专家注释者的预测进行了等效的评级(图3f)。按组织类型细分评估,我们观察到病理学家的评估只有适度的差异,在某些组织中,Mesmer的表现略好于人类注释者,在其他组织中,人类注视者的表现稍好。总之,前面的分析表明,Mesmer可以以人类水平的性能进行全细胞分割。Cellpose和StarDist可能会获得类似的结果,因为当在TissueNet上训练时,它们的性能与Mesmer相当(图2b)。据我们所知,以前的细胞分割算法在组织数据方面没有达到与人类性能相当的效果。
为了完成我们的性能分析,我们试图通过识别Mesmer产生低质量细胞分割的图像来了解Mesmer的局限性。不准确分割的图像的特征是低信噪比、异质染色和聚焦问题(扩展数据图3i)。为了表征这些因素对模型性能的影响,我们在模糊、调整大小或添加图像噪声后评估了模型的准确性。虽然Mesmer对中等图像失真是稳健的,但随着失真幅度的增加,性能会受到影响(扩展数据图3j–l)-正如预期的那样,因为这些操作会从图像中去除信息。


图3。
2.3Mesmer能够对组织成像数据进行精确的下游分析
细胞分割是组织成像数据定量分析的第一步,也是后续单细胞分析的基础。因此,Mesmer生成全细胞和细胞核分割预测的能力应该能够实现以前的分割算法难以执行的分析。一个例子是预测蛋白质在细胞中的亚细胞定位,这可用于量化转录因子的核转位58,59或HER2的膜染色程度,以评估乳腺癌。为了探索亚细胞信号预测的准确性,我们用一组表型标记物对癌症样本进行染色,并用MIBI-TOF61对其进行成像(图4a;方法)。我们创建了一个集成的多路复用图像分析流水线-ak-analysis62-,将Mesmer的分割预测与下游分析联系起来。我们使用预测和基本事实分割掩码(方法)提取每个标记的隔室特异性表达。来自Mesmer的亚细胞定位预测与来自地面实况数据的预测一致(图第4b段)。我们主要观察到已知核标记物(例如Ki67和HH3)的核表达和膜标记物(如E-钙粘蛋白和HER2)的非核表达;图4b)。

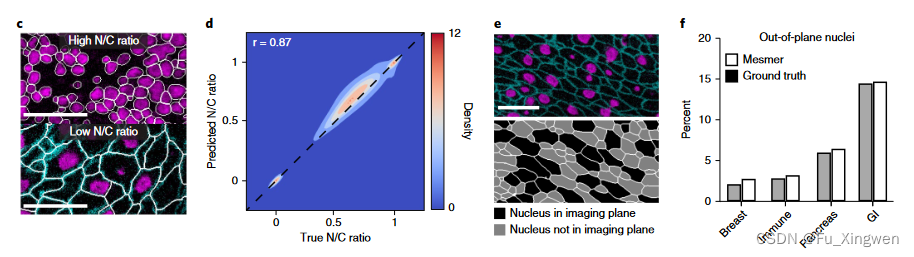

图4|Mesmer能够精确分析多路成像数据。a、 显示细胞核和非细胞核蛋白质染色模式的彩色叠加图(顶部),以及相关的细胞核和全细胞分割预测(底部)。b、 蛋白质在a中的亚细胞定位的量化,用于预测和基本事实分割。预测定位和基本事实分割之间的一致性表明,Mesmer在单细胞水平上准确地量化了蛋白质定位模式。n=1069个细胞。数据以平均值±95%置信区间表示。c、 具有高N/c比(顶部)和低N/c比的组织的示例图像(底部)。N/C比率是用于量化细胞形态的几种指标之一(方法)。d、 在TissueNet的整个测试分割中,N/C比率预测准确性的Pearson相关等值线图表明Mesmer准确地量化了细胞形态。e、 具有成像平面外许多细胞核的组织的代表性图像(顶部),以及根据细胞核是否在成像平面内而着色的相应分割。f、 在预测和基本事实分割中具有平面外细胞核的细胞数量的量化。这些细胞被Mesmer检测到,但基于核分割的方法会漏掉。g、 乳腺癌症样本中多种信息蛋白表达的代表性图像。h、 按细胞谱系着色的预测分割。i、 按细胞谱系着色的地面实况分割。j、 对基本事实和预测分割中每种细胞类型的精确度和召回率的量化表明,Mesmer产生了准确的细胞类型计数。比例尺,50μm。
由于Mesmer还提供了对每个单个细胞核和细胞之间关系的自动分析,因此它应该能够对细胞核与细胞质的比率进行自动评分,病理学家广泛使用该比率来评估恶性程度。我们使用Mesmer为TissueNet测试集中的每个细胞生成细胞核和全细胞分割。然后,我们计算了细胞核与全细胞(N/C)的比率,该比率在概念上与细胞核与细胞质的比率相似,但对于细胞质较少的细胞(例如免疫细胞;方法)具有更高的数值稳定性。Mesmer准确地捕获了具有低和高N/C比率的细胞(图4c),并且在TissueNet中所有细胞的预测N/C比率和真实N/C比率之间存在很强的相关性(Pearson’s r=0.87)(图4d)。
该分析确定了N/C比为零的细胞亚群(图4e),表明在该细胞中没有观察到细胞核。当用于获取数据的成像平面捕捉细胞质而不是细胞核时,这些细胞就会出现。我们量化了TissueNet中具有平面外细胞核的细胞在组织类型中的比例,用于预测和基本事实分割标记,并发现平面外细胞核预测率和真实率之间有很好的一致性(图4f)。平面外细胞核比例最高的发生在胃肠道组织中(图4f),可能是由于柱状上皮的伸长性质。具有平面外细胞核的细胞被基于细胞核的分割方法遗漏,但被Mesmer捕获。
细胞分类是分割后的一项常见任务。分割中的不准确可能导致图像中存在的细胞的识别和计数存在实质性偏差。为了衡量梅斯默的预测如何影响这一过程,我们分析了用Vectra平台生成的一组乳腺癌症样本。每个图像都用一组谱系定义标记物染色(图4g),我们使用这些标记物将每个细胞分类为T细胞、单核细胞、肿瘤细胞或无标记细胞。我们从三名患者中选择了两个不同的区域,并为图像中的所有细胞生成了预测分割和基本分割。我们使用相同的门控方案(方法)将预测的所有细胞分类(图4h)和基本事实(图4i)分割为这些类别。然后,我们计算了患者中每种细胞类型的精确度和召回率。我们观察到两个注释之间的一致性很强(图4j),表明Mesmer的分割预测能够对这些图像中存在的细胞的多样性进行准确分类。
2.4谱系感知分割量化形态学变化
我们已经证明,在TissueNet上训练的模型可以利用这些数据中存在的两个通道,在各种组织类型中准确地分割细胞。然而,一些组织类型具有复杂的形态,仅用两个通道的成像数据无法准确捕捉。例如,蜕膜,即子宫的粘膜,由于母体和胎儿细胞之间的相互作用,显示出细胞大小和细胞形状的显著变化。这些细胞彼此紧密并置,以及它们可以假设的非凸几何形状,使这种复杂性更加复杂,小的圆形细胞位于更大细胞的凹陷中。
这种复杂的形态学使得在使用单个膜通道时分割具有挑战性,即使对于专家注释者也是如此(图第5a段,顶部)。然而,关于每个细胞的位置和形状的信息可以通过包括细胞类型特异性的额外标记来获得(图第5a段,底部)。这些额外的标记物在模型训练期间提供了关于细胞形态的关键信息,当它们组合到单个通道中时,这些信息就会丢失。我们使用MIBI-TOF从具有六个谱系特异性标记的人类蜕膜生成多路成像数据集65,然后使用DeepCell标签从图像的子集生成谱系感知的基本事实分割。我们修改了我们的深度学习架构,以接受这六个通道的输入数据,并使用该数据集(方法)训练了一个模型。尽管这些图像中的细胞形态复杂,但最终的谱系感知分割管道准确地执行了全细胞分割(图第5b段)。
我们使用这种谱系感知的分割流水线来量化蜕膜中细胞随时间的形态学变化。我们首先定义了一系列形态学指标,以捕捉该数据集中细胞形状的多样性(图5c;方法)。人工检查证明了每个类别中细胞的准确分配(图5d)。然后,我们创建了一个自动流水线,为图像62中的每个细胞计算这些度量。我们将我们的流水线应用于该数据集,发现这些指标捕捉到了我们观察到的细胞形状的关键形态学特征(图第5e段)。然后,我们对细胞形态谱(方法)进行k均值聚类,并确定了四个不同的聚类(图5f,g)。为了确定人类蜕膜中这些细胞形态如何随时间变化,我们根据年龄将样本分为两组:孕早期(6-8周)和孕晚期(16-18周)。通过聚类对每个细胞进行染色,突出了两个胎龄组之间细胞形态的差异(图5h,i)。我们在早期观察到簇1细胞(细长)的丰度,在晚期观察到簇2细胞(大型和球状)的丰度(图第5j段)。这种转变可能反映了母体基质细胞在蜕膜过程中所经历的形态学转变。我们的分析表明,全细胞分割可以使细胞形态成为定量可观察的,将病理学家的历史知识和现代多重成像方法联系起来。



图5。
2.5DeepCell支持Mesmer在社区范围内的部署
为了促进深度学习模型的部署,我们的团队之前创建了DeepCell,这是一个用于细胞图像分析的链接开源软件库集合。在这里,我们使用DeepCell使Mesmer能够被更广泛的生物成像社区访问,并根据必须处理的数据量使用两种不同的部署解决方案(图6)。第一个解决方案面向中等数量的数据(<103百万像素图像),并以我们的门户网站为中心https://deepcell.org,承载整个Mesmer管道。用户可以通过这个门户网站直接访问Mesmer,也可以通过我们为ImageJ和QuPath这两个流行的图像分析工具制作的插件提交图像。该门户网站由可扩展的后端(由DeepCell Kiosk39创建)提供服务,该后端动态调整服务器的计算资源,以匹配提交的数据量。这种策略增加了计算资源以在高需求时期支持大量数据,同时在低需求时期减少了这些资源以降低成本。
第二个部署解决方案针对的是数据量较大(>个1百万像素图像)并且需要对所述算法的执行进行更多控制的用户。对于这些用户,我们提供了一个Docker镜像,其中包含完整的Mesmer管道。该图像在用户的计算基础设施上本地生成一个Docker容器,并且可以通过一行命令进行安装。此容器可用于启动交互式Jupyter笔记本,该笔记本可与Mesmer一起处理数据。容器也可以配置为可执行文件,从而可以将Mesmer集成到现有的图像分析工作流程中。此外,我们还开发了一个专门用于分析多路成像数据的软件包,即ark-analysis62,该软件包将基于云的分割预测与下游分析和可视化相结合。
讨论
细胞分割一直是组织成像界的主要瓶颈,因为以前的方法需要大量的手动管理和参数调整才能产生可用的结果。我们的经验表明,这些缺陷可能导致分析延迟数月。Mesmer以用户友好的格式为最广泛使用的荧光和质谱成像平台提供了单一的细胞分割统一解决方案。Mesmer在各种组织和成像模式中实现了人类水平的精度,同时不需要最终用户手动调整参数。为了使Mesmer广泛可用,我们创建了基于云的本地软件解决方案,定位各种背景的用户,为他们的数据生成准确的预测。Mesmer的速度和可扩展性应该有助于分析目前由大型联盟产生的大量多路复用成像数据。梅斯默接受了来自基于荧光和质谱的平台的组织成像数据的训练。通过先前的工作已经实现了H&E(苏木精和伊红)图像和细胞培养中细胞图像的分析;通过DeepCell提供这些功能将是未来工作的重点。
与Mesmer一起,我们展示了TissueNet的首次发布,这是一个用于组织图像的综合细胞分割数据集。TissueNet包含来自9个器官和6个成像平台的>100万个细胞的成对细胞核和全细胞注释。先前的组织数据集不够大,无法训练精确的全细胞分割模型。因此,以前生成精确组织分割模型的努力都集中在核分割上,这意味着几乎所有以前的基准测试都局限于核分割模型的评估。TissueNet将使这些有价值的努力从细胞核分割转向全细胞分割,促进一类新的组织分割算法的开发和基准测试。为了扩展TissueNet的首次发布,我们目前正在构建一个无缝机制,供研究人员添加自己的注释数据。TissueNet的未来发布可能包括来自更广泛的组织类型、成像技术和物种的更高维度数据。
构建TissueNet需要一个新的生成注释的过程。我们没有使用专家的手动注释,而是使用了一种人工循环的方法。因为这种方法中的注释器会纠正模型错误,而不是从头开始创建注释,所以注释时间与模型性能有关。随着模型性能的提高,注释的边际成本降低,提供了注释大量生物图像所需的可扩展性。在这里,我们在之前的工作的基础上,通过将人在循环注释框架与众包相集成。这种集成通过在众包劳动力库中分配工作来提高注释的执行速度,并减少深度学习方法给专家带来的注释负担。因此,众包可以帮助满足生物成像社区的数据注释需求。我们还展示了使用谱系感知分割和定制的六通道模型对具有复杂细胞形态的组织人类蜕膜进行精确分割。虽然这个谱系感知模型仅限于我们的特定数据集,但我们认为它表明了通用谱系感知分割模型的潜力。
我们开发TissueNet和Mesmer的经验提出了一个自然的问题:多少数据就足够了?我们观察到在约104–105个标签处训练数据的回报递减(扩展数据图3c)。我们认为,当准确性是首要问题时,超越这一规模所需的努力是有必要的,例如,对于跨项目或临床应用的模型,Mesmer就是这样。这项工作对于生成黄金标准数据集以对模型性能进行基准测试也是值得的。对于其他用例,较小的数据集和定制的模型可能就足够了。
未来的挑战包括对用于细胞分割的标准化、跨组织抗体面板的需求。建立这样一个小组将是一个重大进展,并将与这里介绍的工作协同增效。三维(3D)71中的全细胞分割是另一个挑战,其将随着成像吞吐量的增加而变得更加突出,以允许对这样的数据集进行常规收集。现有的用于3D实例分割的深度学习方法很有前景,但目前还不存在为未来模型提供动力的TissueNet的3D等效方法。我们在这里的工作可以作为这些工作的起点,因为它可以在组织的二维切片中产生准确的预测(扩展数据图4)。现在,社区可以获得准确的细胞分割,可以从目前生成的数据的多样性中获得许多科学见解。
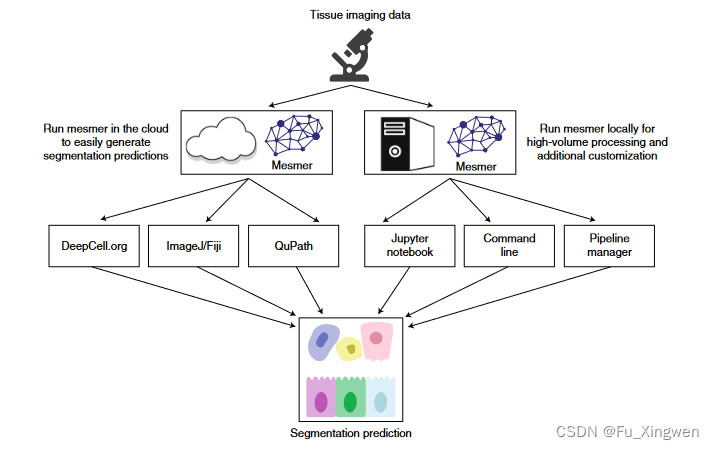
图6|云原生和本地软件促进了Mesmer的部署。集中式网络服务器,https://deepcell.org,承载Mesmer管道的一个版本。需要处理的数据量适中(<103百万像素图像)的用户可以通过门户网站访问该管道。或者,用户可以使用ImageJ和QuPath插件,将数据提交到https://deepcell.orgweb服务器并检索结果。我们还创建了一个容器化的Mesmer版本,该版本与现有的工作流程管理器兼容,因此需要处理大量数据(>1.03百万像素图像)的用户可以从我们的工作中受益。







 Mesmer是一种基于深度学习的全细胞分割算法,实现了人类级别的性能。通过TissueNet,一个包含超过100万个细胞注释的数据集,Mesmer被训练以精确分割组织图像中的细胞。该技术不仅提高了分割准确性,还能进行下游分析,如亚细胞定位和细胞形态量化。Mesmer的开源和社区资源发布促进了生物成像分析的自动化和效率。
Mesmer是一种基于深度学习的全细胞分割算法,实现了人类级别的性能。通过TissueNet,一个包含超过100万个细胞注释的数据集,Mesmer被训练以精确分割组织图像中的细胞。该技术不仅提高了分割准确性,还能进行下游分析,如亚细胞定位和细胞形态量化。Mesmer的开源和社区资源发布促进了生物成像分析的自动化和效率。

















 100
100

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








