
研究背景
组织病理学可以追溯到19世纪,它一直是病理学中使用的黄金标准诊断方法之一。如果在医学检查之后或在外科手术期间需要活组织检查,则需要从患者身上取出组织样本,然后将其切成微米薄片。这些病理学切片包含有关组织病理状态的微观信息;然而,这种薄的切片是透明的,并且在标准光学显微镜下不能提供足够的对比度。组织化学利用标本的细胞和亚细胞化学环境将特殊发色团与特定组织成分结合起来,在可见光显微镜下产生颜色对比,形成专家诊断医师和病理学家诊断组织标本异常的基础。

图1-生物化学组织染色的不确定性
在组织病理学中染色组织样品的标准过程是耗时的,因为它是劳动密集型的工作,并且需要专门的实验室环境,化学试剂和训练有素的人员,例如组织技术人员。在不同实验室和组织学技术人员的处理中,组织染色的不确定性(如图1所示)可能导致误诊,并产生许多质量把控的挑战性问题。此外,目前使用的染色方法并不能保存下原始的组织样品,每一步处理都可能会对组织样本造成不可逆转的影响(如图2所示)。这对于高级分子分析来说是一种限制,因为分析过程需要在初始染色过程之后对相同的组织样品进行。保存不下原始组织样本使得高级分子分析任务难以执行。

图2-传统染色方法造成的组织结构损伤
认识到这些瓶颈,加州大学洛杉矶分校的Ozcan研究团队提出使用深度学习方法来对无标记组织进行虚拟染色。起初,Ozcan研究团队想要找到一种强大而简单的方法来在无标记组织切片的显微图像中引入对比度。为此,Ozcan研究团队选择使用由组织内源性荧光团产生的自体荧光,这种自体荧光天然的存在于样本中。由于可用于有效激发各种组织成分,并且可以使用任何标准荧光显微镜轻松获得,Ozcan研究团队决定使用近紫外荧光激发带。在训练阶段(不成功便成仁的工作),Ozcan研究团队使用了数千个图像块,其包括精确配对的无标记组织自发荧光图像对和与其具有相同组织样本所对应的组织学染色版本的明场图像。在基于生成对抗网络概念的多阶段深度神经网络训练过程之后,Ozcan研究团队提出了一种基于深度学习的方法(如图3所示)以获取未染色(无标记)组织切片中天然存在的荧光化合物的显微图像,并将该自体荧光图像转换成与其具有相同组织样本的明场显微镜等效图像,就好像它是在标准组织染色过程之后拍摄得到的结果。也就是说,Ozcan研究团队使用深度学习方法来对无标签组织样本进行虚拟染色,以此来取代通常由医务人员执行的人工,耗时的处理和染色步骤。利用训练好的神经网络替换组织技术专家完成的大部分任务以此来节省劳动力,成本和时间。

图3-深度学习方法下的虚拟组织染色
模型介绍
在模型选择方面,Ozcan研究团队采用的是生成对抗网络(Generative Adversarial Networks , GAN)。GAN网络由深度学习大牛Goodfellow于2014年提出,包括两个部分:生成器(Generator)和判别器(Discriminator),Ozcan研究团队所使用的模型结构如图4 所示,生成器使用的是U-net模型。

图4-模型概览
生成器和判别器的损失函数被分别定义为:

其中MSE代表L2范数,TV代表总变化量,λ和α为超参数。
实验
Ozcan研究团队使用训练好的GAN模型通过对多种组织类型(如肾,肺,肝,卵巢,唾液腺和甲状腺)和三种不同的染色方法(H&E, Masson’s trichrome以及Jones’ silver)的验证,这种深度学习驱动的虚拟染色方法的被证明是成功的。使用深度学习方法进行虚拟组织染色得到的图像能清楚的展示细胞核,水肿性粘液样变化等病理学家进行病理诊断所需的病理学特性,虚拟染色结果与传统生物化学组织染色结果并无根本性二致(如图5,6,7,8所示)。

图5-唾腺组织染色

图6-肾组织染色

图7-肝组织染色

图8-肺组织染色
Ozcan研究团队对不同器官使用虚拟组织染色和传统生物化学组织染色,比较在实际病理诊断过程中所耗费的时间,虚拟染色方法显然将诊断时间从分钟级降至秒级。
Ozcan研究团队的虚拟染色结果成效由一组具有认证资格的病理学家独立地进行评估。评估内容包括:细胞核细节(ND),细胞质细节(CD),细胞外纤维化(EF)和整体染色(SQ)。其中4分代表完美,3分代表很好,2分代表可以接受,1分代表不能接受。这些病理学家对所要检查地图像起源并不知情,即病理学家不知道哪些图像实际上是被专业技术人员染色,哪些图像是通过神经网络虚拟染色生成。这项由加州大学洛杉矶分校病理学和检验医学系的W. Dean Wallace博士指导的盲测研究结论显示(表1),在染色质量和实际诊断上,两组图像的使用不存在临床意义上的不同,病理学家能够同时使用两种染色技术来识别组织病理学特征,而且两种染色技术之间的一致性很高,不需要一种明确的首选染色技术。
表1-肝组织使用虚拟染色(VS)和传统生物化学染色(HS)方法得到的图像结果评测

总结
深度学习在虚拟组织染色上的应用主要体现在图像风格迁移上,通过实验结果不难发现这种基于统计学习的方法非常的受益于各种成像模式,如荧光显微镜、非线性显微镜、全息显微镜和光学相干断层扫描等。虚拟染色方法的另一个重要优点是染色过程的标准化,因为经过训练的神经网络消除了在不同实验室和组织学技术人员的处理中可能导致的组织染色的不确定性,而这种不确定性本来将可能导致误诊或错误分类的问题。这种通过深度学习方法实现的虚拟染色方法将显著降低成本,缩短样品制备时间,同时还可节省对专业人工的消耗。由于它只需要标准荧光显微镜和简单的计算机(如笔记本电脑),因此在资源有限的环境和发展中国家的病理学需求方面尤其具有变革性。正因为在图像模式下具有如此大的潜力和魅力,深度学习方法或许将改变临床获取染色组织的流程。
其他
Ozcan在文中还介绍了迁移学习,提出在不同的器官组织之间训练模型时,可以使用前一个训练好的模型参数进行初始化,这样能够更快得到用于其他器官组织的模型,如图7所示。
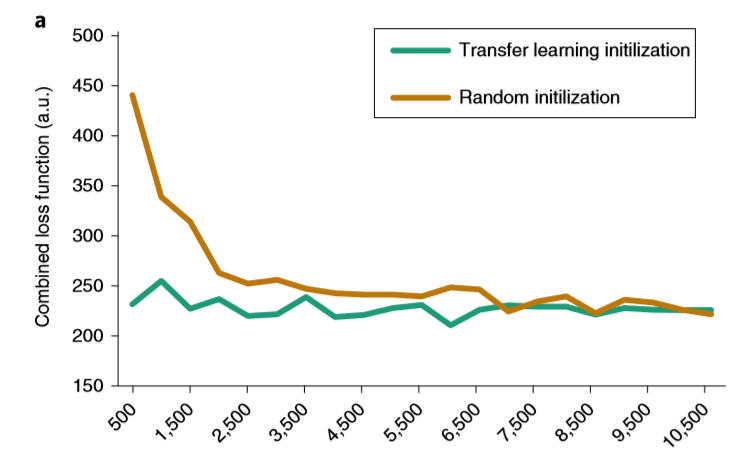
图7-迁移学习与随机初始化对比
参考资料
Rivenson Y., et al. Virtual histological staining of unlabelled tissue-autofluorescence images via deep learning. Nat. Biomed. Eng 3, pages466–477 (2019)

























 2442
2442

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










