alphafold初探
1.数据库初探
近期,alphafold这个模型又开始刷爆各种平台,其主要贡献简单说就是通过AI技术解决了复杂蛋白质结构预测的难题。这意味着,曾经分子生物学家需要花费数年实验来破译的蛋白质结构,AlphaFold只需在几分钟内就能完成。这一突破性的进步证明了人工智能对科学发现的影响。并且,alphafold开源了目前预测得到的蛋白质结构,并构建了一个可供科研目的的开源数据库【1】。

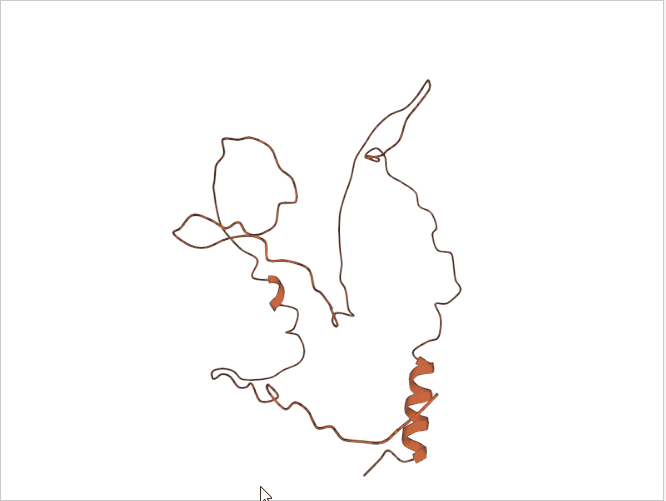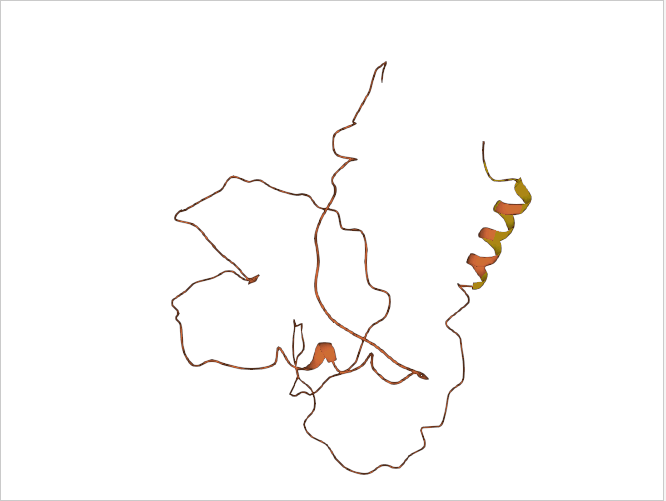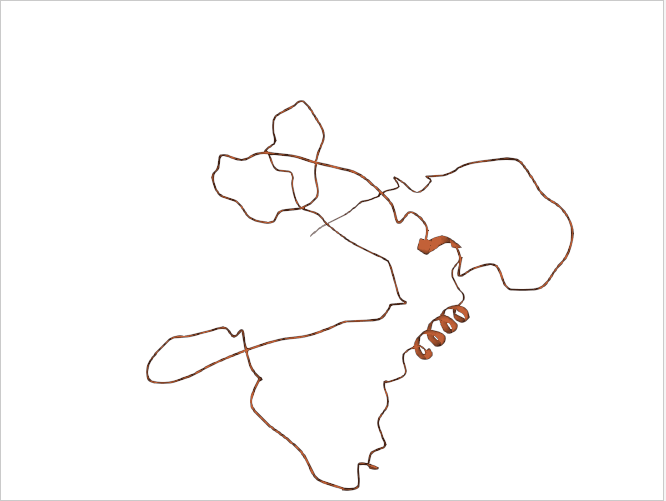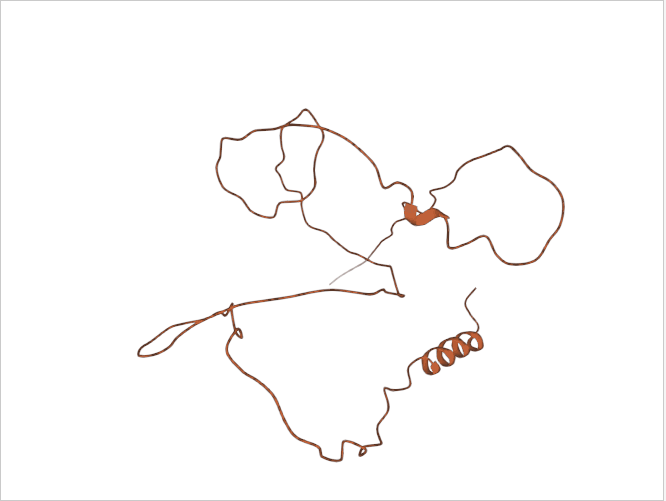
2. mmCIF文件解析与数据读取
2.1 包安装
先安装好biopython包
pip install biopython -i http://pypi.douban.com/simple/ --trusted-host pypi.douban.com
安装成功
Successfully installed biopython-1.79
2.2 肽链结构解析读取
下载一个cif文件,简单测试解析文件
from Bio.PDB.MMCIFParser import MMCIFParser
parser = MMCIFParser()
structure = parser.get_structure('AF-F4HVG8-F1-model_v3', r'C:\Users\LIU\PycharmProjects\pythonProject\Bio_protein\AF-F4HVG8-F1-model_v3.cif')
### 要解释mmCIF的额外信息, 需要MMCIF2Dict
from Bio.PDB.MMCIF2Dict import MMCIF2Dict
mmcif_dict = MMCIF2Dict(r'C:\Users\LIU\PycharmProjects\pythonProject\Bio_protein\AF-F4HVG8-F1-model_v3.cif')
print(f"mmcif_dict: {mmcif_dict}")
内部结构
mmcif_dict: {'data_': 'AF-F4HVG8-F1', '_entry.id': ['AF-F4HVG8-F1'], '_atom_type.symbol': ['C', 'N', 'O', 'S'], '_audit_author.name': ['Jumper, John', 'Evans, Richard', 'Pritzel, Alexander', 'Green, Tim', 'Figurnov, Michael', 'Ronneberger, Olaf', 'Tunyasuvunakool, Kathryn', 'Bates, Russ', 'Zidek, Augustin', 'Potapenko, Anna', 'Bridgland, Alex', 'Meyer, Clemens', 'Kohl, Simon A. A.', 'Ballard, Andrew J.', 'Cowie, Andrew', 'Romera-Paredes, Bernardino', 'Nikolov, Stanislav', 'Jain, Rishub', 'Adler, Jonas', 'Back, Trevor', 'Petersen, Stig', 'Reiman, David', 'Clancy, Ellen', 'Zielinski, Michal', 'Steinegger, Martin', 'Pacholska, Michalina', 'Berghammer, Tamas', 'Silver, David', 'Vinyals, Oriol', 'Senior, Andrew W.', 'Kavukcuoglu, Koray', 'Kohli, Pushmeet', 'Hassabis, Demis'], '_audit_author.pdbx_ordinal': ['1', '2', '3', '4', '5', '6', '7', '8', '9', '10', '11', '12', '13', '14', '15', '16', '17', '18', '19', '20', '21', '22', '23', '24', '25', '26', '27', '28', '29', '30', '31', '32', '33'], '_audit_conform.dict_location': ['https://raw.githubusercontent.com/ihmwg/ModelCIF/master/dist/mmcif_ma.dic'], '_audit_conform.dict_name': ['mmcif_ma.dic'], '_audit_conform.dict_version': ['1.3.9'], '_chem_comp.formula': ['C3 H7 N O2', 'C6 H15 N4 O2', 'C4 H8 N2 O3', 'C4 H7 N O4', 'C3 H7 N O2 S', 'C5 H10 N2 O3', 'C5 H9 N O4', 'C2 H5 N O2', 'C6 H10 N3 O2', 'C6 H13 N O2', 'C6 H13 N O2', 'C6 H15 N2 O2', 'C5 H11 N O2 S', 'C9 H11 N O2', 'C5 H9 N O2', 'C3 H7 N O3', 'C4 H9 N O3', 'C11 H12 N2 O2', 'C9 H11 N O3', 'C5 H11 N O2'], '_chem_comp.formula_weight': ['89.093', '175.209', '132.118', '133.103', '121.158', '146.144', '147.129', '75.067', '156.162', '131.173', '131.173', '147.195', '149.211', '165.189', '115.130', '105.093', '119.119', '204.225', '181.189', '117.146'], '_chem_comp.id': ['ALA', 'ARG', 'ASN', 'ASP', 'CYS', 'GLN', 'GLU', 'GLY', 'HIS', 'ILE', 'LEU', 'LYS', 'MET', 'PHE', 'PRO', 'SER', 'THR', 'TRP', 'TYR', 'VAL'], '_chem_comp.mon_nstd_flag': ['y', 'y', 'y', 'y', 'y', 'y', 'y', 'y', 'y', 'y', 'y', 'y', 'y', 'y', 'y', 'y', 'y', 'y', 'y', 'y'], '_chem_comp.name': ['ALANINE', 'ARGININE', 'ASPARAGINE', 'ASPARTIC ACID', 'CYSTEINE', 'GLUTAMINE', 'GLUTAMIC ACID', 'GLYCINE', 'HISTIDINE', 'ISOLEUCINE', 'LEUCINE', 'LYSINE', 'METHIONINE', 'PHENYLALANINE', 'PROLINE', 'SERINE', 'THREONINE', 'TRYPTOPHAN', 'TYROSINE', 'VALINE'], '_chem_comp.pdbx_synonyms': ['?', '?', '?', '?', '?', '?', '?', '?', '?', '?', '?', '?', '?', '?', '?', '?', '?', '?', '?', '?'], '_chem_comp.type': ['L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING', 'L-PEPTIDE LINKING'], '_citation.book_publisher': ['?'], '_citation.country': ['UK'], '_citation.id': ['1'], '_citation.journal_full': ['Nature'], '_citation.journal_id_ASTM': ['NATUAS'], '_citation.journal_id_CSD': ['0006'], '_citation.journal_id_ISSN': ['0028-0836'], '_citation.journal_volume': ['596'], '_citation.page_first': ['583'], '_citation.page_last': ['589'], '_citation.pdbx_database_id_DOI': ['10.1038/s41586-021-03819-2'], '_citation.pdbx_database_id_PubMed': ['34265844'], '_citation.title': ['Highly accurate protein structure prediction with AlphaFold'], '_citation.year': ['2021'], '_citation_author.citation_id': ['1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1'], '_citation_author.name': ['Jumper, John', 'Evans, Richard', 'Pritzel, Alexander', 'Green, Tim', 'Figurnov, Michael', 'Ronneberger, Olaf', 'Tunyasuvunakool, Kathryn', 'Bates, Russ', 'Zidek, Augustin', 'Potapenko, Anna', 'Bridgland, Alex', 'Meyer, Clemens', 'Kohl, Simon A. A.', 'Ballard, Andrew J.', 'Cowie, Andrew', 'Romera-Paredes, Bernardino', 'Nikolov, Stanislav', 'Jain, Rishub', 'Adler, Jonas', 'Back, Trevor', 'Petersen, Stig', 'Reiman, David', 'Clancy, Ellen', 'Zielinski, Michal', 'Steinegger, Martin', 'Pacholska, Michalina', 'Berghammer, Tamas', 'Silver, David', 'Vinyals, Oriol', 'Senior, Andrew W.', 'Kavukcuoglu, Koray', 'Kohli, Pushmeet', 'Hassabis, Demis'], '_citation_author.ordinal': ['1', '2', '3', '4', '5', '6', '7', '8', '9', '10', '11', '12', '13', '14', '15', '16', '17', '18', '19', '20', '21', '22', '23', '24', '25', '26', '27', '28', '29', '30', '31', '32', '33'], '_database_2.database_code': ['AF-F4HVG8-F1'], '_database_2.database_id': ['AlphaFoldDB'], '_entity.details': ['?'], '_entity.formula_weight': ['?'], '_entity.id': ['1'], '_entity.pdbx_description': ['Chloroplast sensor kinase, chloroplastic'], '_entity.pdbx_ec': ['?'], '_entity.pdbx_fragment': ['?'], '_entity.pdbx_mutation': ['?'], '_entity.pdbx_number_of_molecules': ['1'], '_entity.src_method': ['man'], '_entity.type': ['polymer'], '_entity_poly.entity_id': ['1'], '_entity_poly.nstd_linkage': ['no'], '_entity_poly.nstd_monomer': ['no'], '_entity_poly.pdbx_seq_one_letter_code': ['MLLSAIASQTLLSSNPNLHFSNSIPNPRPSNPSLKLLNASSSSSSSSSSSIFTRGLRYVNHTVSNEESEPGGGETMVASA\nSAIASAIRGASTTPVEFTQMIEKDHLKTKIILPSPDFQRLCLEQLDLFRQIVDPNAVLSIYVRPAGSYVMDRLELRRVTC\nYPSVNAGDVVILVGNFGIPAGLRAAEASLSSQQVELVSKHRAAVFPMVKHPFVVGFLVAELPVEAEEEEEEEEEEKPHGV\nNQFLSPEEAYALPASANTKSPRVKLPSVKVFTEEQRSYAINISRTLAMAYVMDQKTMLLQQSSWQNNVRMSKLVEQIRGP\nLSTMRTLSKMLSTHTKRNQISHDIVEDLIVQGDQIKDTLEELQDAVHLTKANIVRHNEEALKKINKTHNETRRSKYEHKD\nPIDGSQISSTRLSLGSGLDDSEMPMPPLALAPLQMHSIRPCDISNVLLDMVETVRPLALTQQRVVELGENSASLQVAVEE\nPALRQALSNLIEGALLRTHVGGKVEILSTRAPAGGSLVVIDDDGPDMRYMTQMHSLTPFGAELLSENMVEDNMTWNFVAG\nLTVAREILESYGCVIRVISPRSSDAALGAGGTRVELWLPAFPAAVSEANEA'], '_entity_poly.pdbx_seq_one_letter_code_can': ['MLLSAIASQTLLSSNPNLHFSNSIPNPRPSNPSLKLLNASSSSSSSSSSSIFTRGLRYVNHTVSNEESEPGGGETMVASA\nSAIASAIRGASTTPVEFTQMIEKDHLKTKIILPSPDFQRLCLEQLDLFRQIVDPNAVLSIYVRPAGSYVMDRLELRRVTC\nYPSVNAGDVVILVGNFGIPAGLRAAEASLSSQQVELVSKHRAAVFPMVKHPFVVGFLVAELPVEAEEEEEEEEEEKPHGV\nNQFLSPEEAYALPASANTKSPRVKLPSVKVFTEEQRSYAINISRTLAMAYVMDQKTMLLQQSSWQNNVRMSKLVEQIRGP\nLSTMRTLSKMLSTHTKRNQISHDIVEDLIVQGDQIKDTLEELQDAVHLTKANIVRHNEEALKKINKTHNETRRSKYEHKD\nPIDGSQISSTRLSLGSGLDDSEMPMPPLALAPLQMHSIRPCDISNVLLDMVETVRPLALTQQRVVELGENSASLQVAVEE\nPALRQALSNLIEGALLRTHVGGKVEILSTRAPAGGSLVVIDDDGPDMRYMTQMHSLTPFGAELLSENMVEDNMTWNFVAG\nLTVAREILESYGCVIRVISPRSSDAALGAGGTRVELWLPAFPAAVSEANEA'], '_entity_poly.pdbx_strand_id': ['A'], '_entity_poly.type': ['polypeptide(L)'], '_entity_poly_seq.entity_id': ['1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1', '1'], '_entity_poly_seq.hetero': ['n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n', 'n'], '_entity_poly_seq.mon_id': ['MET', 'LEU', 'LEU', 'SER', 'ALA', 'ILE', 'ALA', 'SER', 'GLN', 'THR', 'LEU', 'LEU', 'SER', 'SER', 'ASN', 'PRO', 'ASN', 'LEU', 'HIS', 'PHE', 'SER', 'ASN', 'SER', 'ILE', 'PRO', 'ASN', 'PRO', 'ARG', 'PRO', 'SER', 'ASN', 'PRO', 'SER', 'LEU', 'LYS', 'LEU', 'LEU', 'ASN', 'ALA', 'SER', 'SER', 'SER', 'SER', 'SER', 'SER', 'SER', 'SER', 'SER', 'SER', 'SER', 'ILE', 'PHE', 'THR', 'ARG', 'GLY', 'LEU', 'ARG', 'TYR', 'VAL', 'ASN', 'HIS', 'THR', 'VAL', 'SER', 'ASN', 'GLU', 'GLU', 'SER', 'GLU', 'PRO', 'GLY', 'GLY', 'GLY', 'GLU', 'THR', 'MET', 'VAL', 'ALA', 'SER', 'ALA', 'SER', 'ALA', 'ILE', 'ALA', 'SER', 'ALA', 'ILE', 'ARG', 'GLY', 'ALA', 'SER', 'THR', 'THR', 'PRO', 'VAL', 'GLU', 'PHE', 'THR', 'GLN', 'MET', 'ILE', 'GLU', 'LYS', 'ASP', 'HIS', 'LEU', 'LYS', 'THR', 'LYS', 'ILE', 'ILE', 'LEU', 'PRO', 'SER', 'PRO', 'ASP', 'PHE', 'GLN', 'ARG', 'LEU', 'CYS', 'LEU', 'GLU', 'GLN', 'LEU', 'ASP', 'LEU', 'PHE', 'ARG', 'GLN', 'ILE', 'VAL', 'ASP', 'PRO', 'ASN', 'ALA', 'VAL', 'LEU', 'SER', 'ILE', 'TYR', 'VAL', 'ARG', 'PRO', 'ALA', 'GLY', 'SER', 'TYR', 'VAL', 'MET', 'ASP', 'ARG', 'LEU', 'GLU', 'LEU', 'ARG', 'ARG', 'VAL', 'THR', 'CYS', 'TYR', 'PRO', 'SER', 'VAL', 'ASN', 'ALA', 'GLY', 'ASP', 'VAL', 'VAL', 'ILE', 'LEU', 'VAL', 'GLY', 'ASN', 'PHE', 'GLY', 'ILE', 'PRO', 'ALA', 'GLY', 'LEU', 'ARG', 'ALA', 'ALA', 'GLU', 'ALA', 'SER', 'LEU', 'SER', 'SER', 'GLN', 'GLN', 'VAL', 'GLU', 'LEU', 'VAL', 'SER', 'LYS', 'HIS', 'ARG', 'ALA', 'ALA', 'VAL', 'PHE', 'PRO', 'MET', 'VAL', 'LYS', 'HIS', 'PRO', 'PHE', 'VAL', 'VAL', 'GLY', 'PHE', 'LEU', 'VAL', 'ALA', 'GLU', 'LEU', 'PRO', 'VAL', 'GLU', 'ALA', 'GLU', 'GLU', 'GLU', 'GLU', 'GLU', 'GLU', 'GLU', 'GLU', 'GLU', 'GLU', 'LYS', 'PRO', 'HIS', 'GLY', 'VAL', 'ASN', 'GLN', 'PHE', 'LEU', 'SER', 'PRO', 'GLU', 'GLU', 'ALA', 'TYR', 'ALA', 'LEU', 'PRO', 'ALA', 'SER', 'ALA', 'ASN', 'THR', 'LYS', 'SER', 'PRO', 'ARG', 'VAL', 'LYS', 'LEU', 'PRO', 'SER', 'VAL', 'LYS', 'VAL', 'PHE', 'THR', 'GLU', 'GLU', 'GLN', 'ARG', 'SER', 'TYR', 'ALA', 'ILE', 'ASN', 'ILE', 'SER', 'ARG', 'THR', 'LEU', 'ALA', 'MET', 'ALA', 'TYR', 'VAL', 'MET', 'ASP', 'GLN', 'LYS', 'THR', 'MET', 'LEU', 'LEU', 'GLN', 'GLN', 'SER', 'SER', 'TRP', 'GLN', 'ASN', 'ASN', 'VAL', 'ARG', 'MET', 'SER', 'LYS', 'LEU', 'VAL', 'GLU', 'GLN', 'ILE', 'ARG', 'GLY', 'PRO', 'LEU', 'SER', 'THR', 'MET', 'ARG', 'THR', 'LEU', 'SER', 'LYS', 'MET', 'LEU', 'SER', 'THR', 'HIS', 'THR', 'LYS', 'ARG', 'ASN', 'GLN', 'ILE', 'SER', 'HIS', 'ASP', 'ILE', 'VAL', 'GLU', 'ASP', 'LEU', 'ILE', 'VAL', 'GLN', 'GLY', 'ASP', 'GLN', 'ILE', 'LYS', 'ASP', 'THR', 'LEU', 'GLU', 'GLU', 'LEU', 'GLN', 'ASP', 'ALA', 'VAL', 'HIS', 'LEU', 'THR', 'LYS', 'ALA', 'ASN', 'ILE', 'VAL', 'ARG', 'HIS', 'ASN', 'GLU', 'GLU', 'ALA', 'LEU', 'LYS', 'LYS', 'ILE', 'ASN', 'LYS', 'THR', 'HIS', 'ASN', 'GLU', 'THR', 'ARG', 'ARG', 'SER', 'LYS', 'TYR', 'GLU', 'HIS', 'LYS', 'ASP', 'PRO', 'ILE', 'ASP', 'GLY', 'SER', 'GLN', 'ILE', 'SER', 'SER', 'THR', 'ARG', 'LEU', 'SER', 'LEU', 'GLY', 'SER', 'GLY', 'LEU', 'ASP', 'ASP', 'SER', 'GLU', 'MET', 'PRO', 'MET', 'PRO', 'PRO', 'LEU', 'ALA', 'LEU', 'ALA', 'PRO', 'LEU', 'GLN', 'MET', 'HIS', 'SER', 'ILE', 'ARG', 'PRO', 'CYS', 'ASP', 'ILE', 'SER', 'ASN', 'VAL', 'LEU', 'LEU', 'ASP', 'MET', 'VAL', 'GLU', 'THR', 'VAL', 'ARG', 'PRO', 'LEU', 'ALA', 'LEU', 'THR', 'GLN', 'GLN', 'ARG', 'VAL', 'VAL', 'GLU', 'LEU', 'GLY', 'GLU', 'ASN', 'SER', 'ALA', 'SER', 'LEU', 'GLN', 'VAL', 'ALA', 'VAL', 'GLU', 'GLU', 'PRO', 'ALA', 'LEU', 'ARG', 'GLN', 'ALA', 'LEU', 'SER', 'ASN', 'LEU', 'ILE', 'GLU', 'GLY', 'ALA', 'LEU', 'LEU', 'ARG', 'THR', 'HIS', 'VAL', 'GLY', 'GLY', 'LYS', 'VAL', 'GLU', 'ILE', 'LEU', 'SER', 'THR', 'ARG', 'ALA', 'PRO', 'ALA', 'GLY', 'GLY', 'SER', 'LEU', 'VAL', 'VAL', 'ILE', 'ASP', 'ASP', 'ASP', 'GLY', 'PRO', 'ASP', 'MET', 'ARG', 'TYR', 'MET', 'THR', 'GLN', 'MET', 'HIS', 'SER', 'LEU', 'THR', 'PRO', 'PHE', 'GLY', 'ALA', 'GLU', 'LEU', 'LEU', 'SER', 'GLU', 'ASN', 'MET', 'VAL', 'GLU', 'ASP', 'ASN', 'MET', 'THR', 'TRP', 'ASN', 'PHE', 'VAL', 'ALA', 'GLY', 'LEU', 'THR', 'VAL', 'ALA', 'ARG', 'GLU', 'ILE', 'LEU', 'GLU', 'SER', 'TYR', 'GLY', 'CYS', 'VAL', 'ILE', 'ARG', 'VAL', 'ILE', 'SER', 'PRO', 'ARG', 'SER', 'SER', 'ASP', 'ALA', 'ALA', 'LEU', 'GLY', 'ALA', 'GLY', 'GLY', 'THR', 'ARG', 'VAL', 'GLU', 'LEU', 'TRP', 'LEU', 'PRO', 'ALA', 'PHE', 'PRO', 'ALA', 'ALA', 'VAL', 'SER', 'GLU', 'ALA', 'ASN', 'GLU', 'ALA'], '_entity_poly_seq.num': ['1', '2', '3', '4', '5', '6', '7', '8', '9', '10', '11', '12', '13', '14', '15', '16', '17', '18', '19', '20', '21', '22', '23', '24', '25', '26', '27', '28', '29', '30', '31', '32', '33', '34', '35', '36', '37', '38', '39', '40', '41', '42', '43', '44', '45', '46', '47', '48', '49', '50', '51', '52', '53', '54', '55', '56', '57', '58', '59', '60', '61', '62', '63', '64', '65', '66', '67', '68', '69', ...
太长了,做一个简单解析,读取肽链
structure=parser.get_structure(filename=r'C:\Users\LIU\PycharmProjects\pythonProject\Bio_protein\AF-F4HVG8-F1-model_v3.cif',structure_id=None)
chains=structure.get_chains()
print(f"chain{list(chains)}")
结果
[<Chain id=A>]
更多解析工具可以参考Biopython的wiki【2】
参考文献资料
[1] AlphaFold Protein Structure Database
[2] Biopython的API文档
























 2703
2703











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










