从三维图像中抽取样本进行2D网络训练-MONAI实战
在医学影像分析的任务中,有时需要从三维(3D)图像数据中提取二维(2D)样本进行训练。这是因为2D卷积神经网络可以更方便地处理和分析2D数据,而且使用2D样本可以减少训练时间和计算成本。
然而,从3D数据中提取2D样本并不是一件容易的事情。本教程向您展示如何使用 3D inputs 中采样2D样本训练网络。这是一个完整的2D分割教程,包含数据生成,预处理,训练以及推理
主要用到的功能函数:monai的RandSpatialCropSamplesd和PatchDataset进行2D切片的抽样。以及在推理中使用SliceInferer将2D切片分割后再组合成3D图像。所以整个端到端的流程是 input 3D volume --> model --> output 3D volume.
查看本教程前,请自行下载参考代码,边跑代码边看教程,学习效率更高哦
1.导入包
导入所有需要的包,monai版本为1.3,CPU、GPU均可训练
import os
import tempfile
from glob import glob
import shutil
import matplotlib.pyplot as plt
import monai
import nibabel as nib
import numpy as np
import torch
from monai.data import DataLoader, PatchDataset, create_test_image_3d
from monai.inferers import SliceInferer
from monai.metrics import DiceMetric
from monai.transforms import (
Compose,
EnsureChannelFirstd,
EnsureTyped,
LoadImaged,
RandRotate90d,
Resized,
ScaleIntensityd,
SqueezeDimd,
)
from monai.visualize import matshow3d
monai.config.print_config()
monai.utils.set_determinism(0)
2.生成实验数据
原教程中使用临时地址存放数据,我习惯自己创建一个地址保存数据,方便浏览数据。
root_dir = '/Users/Downloads/monai_data'
if not os.path.exists(root_dir):
os.makedirs(root_dir)
使用create_test_image_3d创建40个尺寸不同的3D images和对应的labels.
print(f"generating synthetic data to {root_dir} (this may take a while)")
for i in range(40):
# make the input volumes different spatial shapes for demo purposes
H, W, D = 30 + i, 40 + i, 50 + i
im, seg = create_test_image_3d(H, W, D, num_seg_classes=1, channel_dim=-1, rad_max=10)
n = nib.Nifti1Image(im, np.eye(4))
nib.save(n, os.path.join(root_dir, f"img{i:d}.nii.gz"))
n = nib.Nifti1Image(seg, np.eye(4))
nib.save(n, os.path.join(root_dir, f"seg{i:d}.nii.gz"))
images = sorted(glob(os.path.join(root_dir, "img*.nii.gz")))
segs = sorted(glob(os.path.join(root_dir, "seg*.nii.gz")))
并将前35个设置为训练集,后3个为测试集。
train_files = [{"img": img, "seg": seg} for img, seg in zip(images[:35], segs[:35])]
val_files = [{"img": img, "seg": seg} for img, seg in zip(images[-3:], segs[-3:])]
3.体素水平(volume-level)的预处理
假设使用3D网络,那transform 和 dataset就类似于下面代码
# volume-level transforms for both image and segmentation
train_transforms = Compose(
[
LoadImaged(keys=["img", "seg"]),
EnsureChannelFirstd(keys=["img", "seg"]),
ScaleIntensityd(keys="img"),
RandRotate90d(keys=["img", "seg"], prob=0.5, spatial_axes=[0, 2]),
EnsureTyped(keys=["img", "seg"]),
]
)
# 3D dataset with preprocessing transforms
volume_ds = monai.data.CacheDataset(data=train_files, transform=train_transforms)
# use batch_size=1 to check the volumes because the input volumes have different shapes
check_loader = DataLoader(volume_ds, batch_size=1)
check_data = monai.utils.misc.first(check_loader)
print("first volume's shape: ", check_data["img"].shape, check_data["seg"].shape)
关于这部分,可以查看我之前的教程,这里不赘述
【添加链接】
接下来使用matshow3d函数展示image 和 label
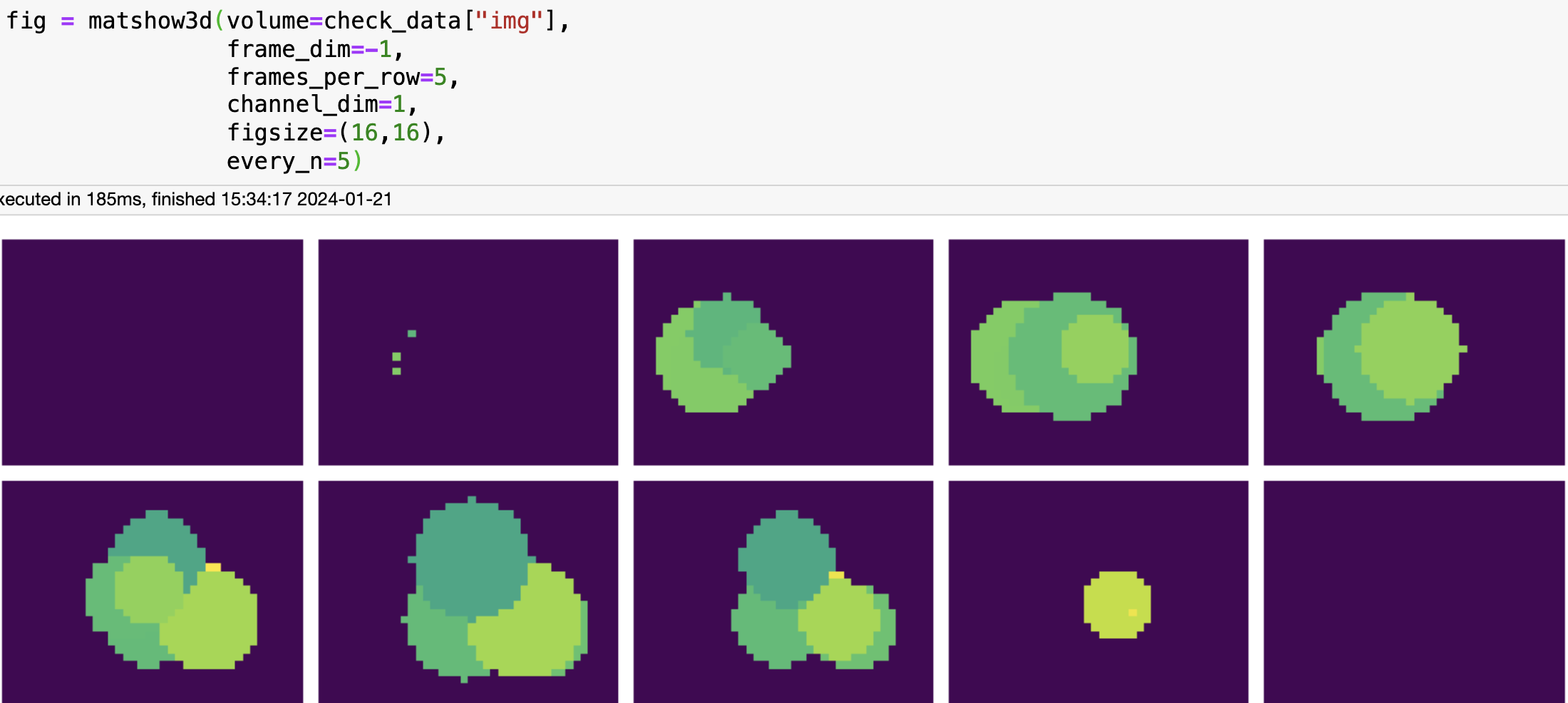
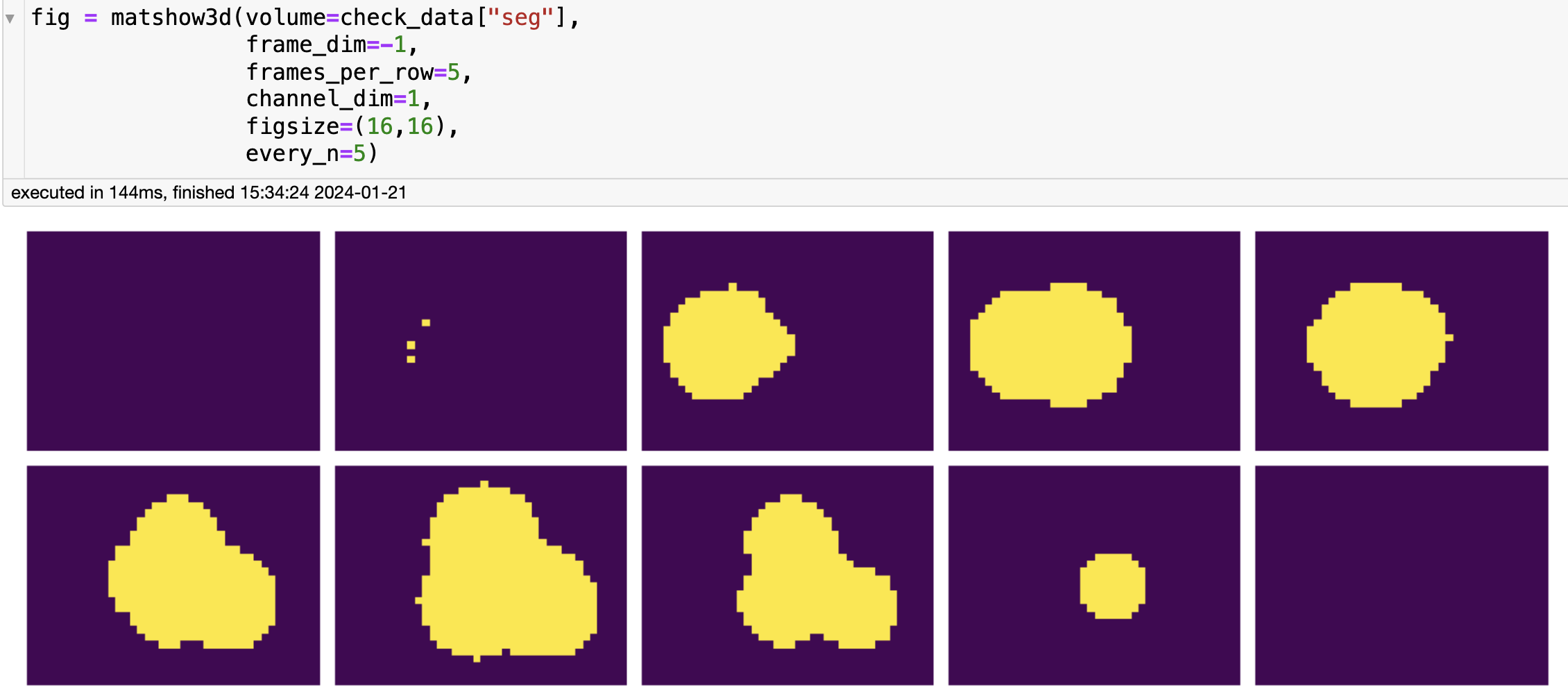
从图中可以看出,label就是image中值大于0的部分。
【添加链接】
4. 切片(patch-level)预处理
接下来就是本次的重点。如何从 3D 中切2D patch出来
num_samples = 4
patch_func = monai.transforms.RandSpatialCropSamplesd(
keys=["img", "seg"],
roi_size=[-1, -1, 1], # dynamic spatial_size for the first two dimensions
num_samples=num_samples,
random_size=False,
)
# this can also be balanced samples:
# patch_func = RandCropByPosNegLabeld(
# keys=["img", "seg"],
# label_key="seg",
# spatial_size=[-1, -1, 1], # dynamic spatial_size for the first two dimensions
# pos=1,
# neg=1,
# num_samples=num_samples,
# )
patch_func: 一个转换函数(RandSpatialCropSamplesd),从原始图像和分割掩模生成随机空间裁剪。注意这里的roi_size=[-1, -1, 1]。前面两维表示动态获取,最后一维只取1层。发现没,只要将最后一维设成1不就取到了二维图像吗?我们的数据大小是30x40x50,按这种取法就是30x50x1,至于取得是哪一层?随机取!想取几层就改num_samples
使用RandSpatialCropSamplesd随机性太强了,万一后面裁剪后取不到正样本怎么办,还可以使用RandCropByPosNegLabeld来平衡正负样本。两种方式看个人需求
patch_transform = Compose(
[
SqueezeDimd(keys=["img", "seg"], dim=-1), # squeeze the last dim
Resized(keys=["img", "seg"], spatial_size=[48, 48]),
# to use crop/pad instead of resize:
# ResizeWithPadOrCropd(keys=["img", "seg"], spatial_size=[48, 48], mode="replicate"),
]
)
patch_ds = PatchDataset(
volume_ds,
transform=patch_transform,
patch_func=patch_func,
samples_per_image=num_samples,
)
shuffle_ds = monai.data.ShuffleBuffer(patch_ds, seed=0)
train_loader = DataLoader(
shuffle_ds,
batch_size=3,
num_workers=2,
pin_memory=torch.cuda.is_available(),
)
check_data = monai.utils.misc.first(train_loader)
print("first patch's shape: ", check_data["img"].shape, check_data["seg"].shape)
-
PatchDataset: 基于现有数据集(volume_ds)和一个patch函数(patch_func)创建的数据集。它从原始样本中生成patches。 -
Compose: 应用于每个patch的transform,包括:SqueezeDimd: 压缩图像和分割掩模的最后一个维度。Resized: 将图像和分割掩模resize到[48,48]。
-
ShuffleBuffer: 一个缓冲区,将PatchDataset中的样本打乱以提供训练期间的随机顺序。这有助于提高训练过程中的效果。
创建完dataset后思考一个问题:现在的数据量怎么计算,batch_size=3是指原始图像3个,还是切好后的2D patch 3个?
这里的batch_size 指的是 patch_size. patch数量=图像数量 x num_samples
注意:使用patch的时候,volume_transform也是必要的,不能只有patch_transform
数据准备好了,现在准备训练吧
5.定义一个2D网络和loss
device = torch.device("cuda" if torch.cuda.is_available() else "cpu")
model = monai.networks.nets.UNet(
spatial_dims=2,
in_channels=1,
out_channels=1,
channels=(16, 32, 64, 128),
strides=(2, 2, 2),
num_res_units=2,
).to(device)
loss_function = monai.losses.DiceLoss(sigmoid=True)
optimizer = torch.optim.Adam(model.parameters(), 5e-3)
6.一个标准的分割训练步骤
epoch_loss_values = []
num_epochs = 5
for epoch in range(num_epochs):
print("-" * 10)
print(f"epoch {epoch + 1}/{num_epochs}")
model.train()
epoch_loss, step = 0, 0
for batch_data in train_loader:
step += 1
inputs, labels = batch_data["img"].to(device), batch_data["seg"].to(device)
optimizer.zero_grad()
outputs = model(inputs)
loss = loss_function(outputs, labels)
loss.backward()
optimizer.step()
epoch_loss += loss.item()
if step % 15 == 0:
print(f"{step}, train_loss: {loss.item():.4f}")
epoch_loss /= step
epoch_loss_values.append(epoch_loss)
print(f"epoch {epoch + 1} average loss: {epoch_loss:.4f}")
print("train completed")
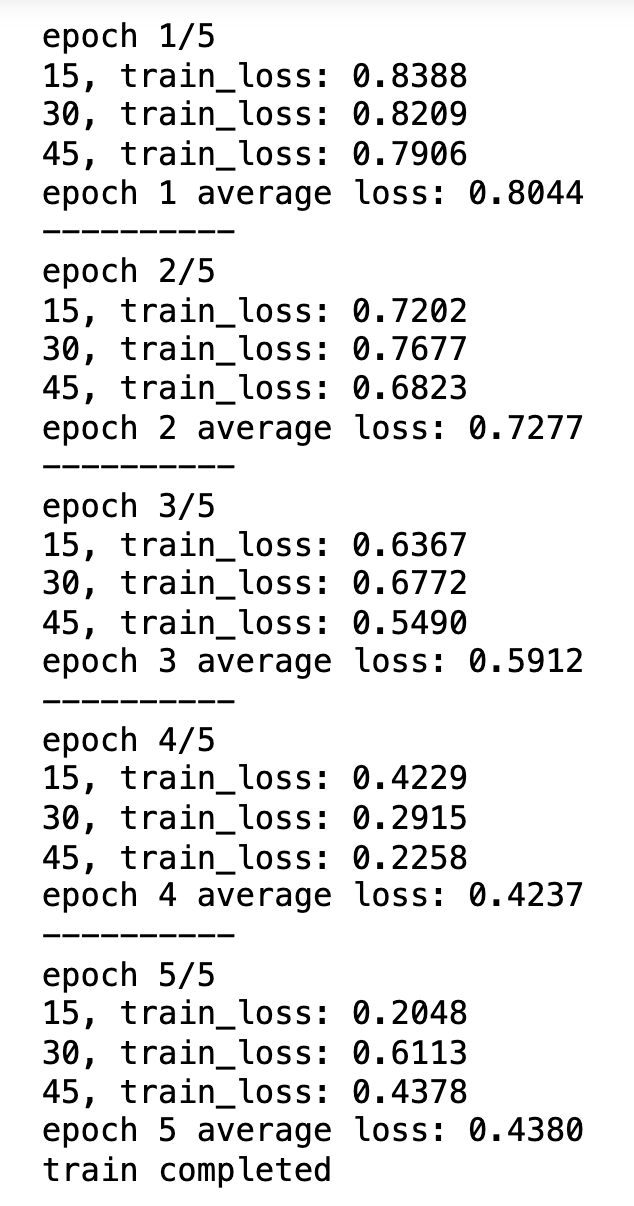
思考:像图中的step次数如何计算?
# Calculate the number of iterations
total_samples = len(patch_ds)
batch_size = 3
num_iterations = total_samples // batch_size
print(f"Number of iterations per epoch: {num_iterations}")
使用SliceInferer推理
网络是slice by slice训练的,所以推理也应该是 slice by slice, 但是我们的图像是三维的,网络要是2维输入,输出又要是一个完整的3D图像,怎么办到?
先把整个3D volume输入进来再说
val_transform = Compose(
[
LoadImaged(keys=["img", "seg"]),
EnsureChannelFirstd(keys=["img", "seg"]),
ScaleIntensityd(keys="img"),
EnsureTyped(keys=["img", "seg"]),
]
)
val_files = [{"img": img, "seg": seg} for img, seg in zip(images[-3:], segs[-3:])]
val_ds = monai.data.Dataset(data=val_files, transform=val_transform)
data_loader = DataLoader(val_ds, num_workers=1, pin_memory=torch.cuda.is_available())
dice_metric = DiceMetric(include_background=True, reduction="mean", get_not_nans=False)
使用SliceInferer
model.eval()
with torch.no_grad():
for val_data in data_loader:
val_images = val_data["img"].to(device)
roi_size = (48, 48)
sw_batch_size = 3
slice_inferer = SliceInferer(
roi_size=roi_size,
sw_batch_size=sw_batch_size,
spatial_dim=1, # Spatial dim to slice along is defined here
device=torch.device("cpu"),
padding_mode="replicate",
)
val_output = slice_inferer(val_images, model).cpu()
dice_metric(y_pred=val_output > 0.5, y=val_data["seg"])
print("Dice: ", dice_metric.get_buffer()[-1][0])
fig = plt.figure(figsize=(10, 4))
plt.subplot(1, 2, 1)
matshow3d(val_output[0], fig=plt.gca())
plt.subplot(1, 2, 2)
matshow3d(val_images[0], fig=plt.gca())
plt.show()
print(f"Avg Dice: {dice_metric.aggregate().item()}")
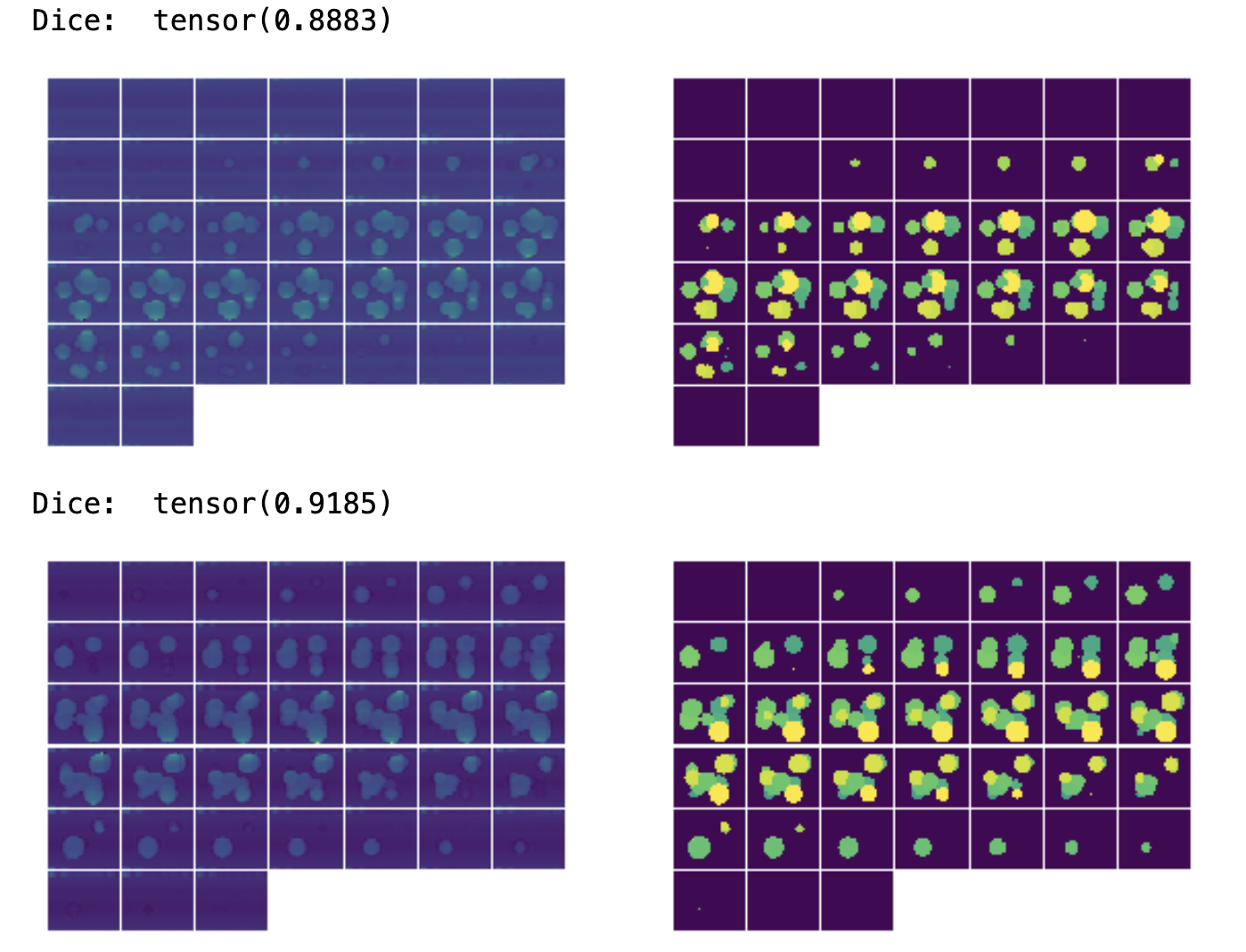
以上就是这期教程的所有内容,你学废了吗?

拓展学习:
- 更多SliceInferer的用法去官方github
- 使用
PatchIterdandGridPatchDataset也可以进行2D 切片训练,附加学习教程
文章持续更新,可以关注微公【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持以实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连










 本文介绍了如何使用MONAI库从三维医学图像中抽取2D样本,以训练2D卷积神经网络,包括数据生成、预处理、模型训练和推理过程,重点讲解了如何使用RandSpatialCropSamplesd和PatchDataset进行切片操作,以及如何通过SliceInferer将2D分割结果整合回3D图像。
本文介绍了如何使用MONAI库从三维医学图像中抽取2D样本,以训练2D卷积神经网络,包括数据生成、预处理、模型训练和推理过程,重点讲解了如何使用RandSpatialCropSamplesd和PatchDataset进行切片操作,以及如何通过SliceInferer将2D分割结果整合回3D图像。















 1709
1709











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










