使用 Monai Bundle 和 Model Zoo 对医学影像数据进行分类-全脑133个结构分割
文章目录
导读
本系列涵盖从 3D Slicer 医学图像查看器的基础使用到高级自动分割扩展程序的内容(从入门到高阶!),具体包括软件安装、基础使用教程,自动分割扩展(totalsegmentator, monai label)快速标注数据。
在本系列第三部分中,我们在工作站上安装了 MONAILabel 服务端和 MONAILabel 3D Slicer 客户端。在第四部分内容中,我们从一个简单的腹部CT脾脏分割案例开始,介绍了monai label使用的一些基础用法。在第五部分中,使用radiology app进行全脊柱的分割,并且详细介绍了radiology app所有用法。
本节重点:在这一节中,我们将尝试使用monai bundle 和model zoo里面的分割模型来分割其他结构。
该系列之前的内容如下:
【添加链接】
如果你没看过系列之前的内容,以下是monai label一个简单的介绍
[ MONAI Label 是一个服务器-客户端系统,它利用 AI 促进交互式医学图像注释。它是一个开源且易于安装的生态系统,可以在具有单个或多个 GPU 的机器上本地运行。服务器和客户端都在同一台/不同的机器上工作。它与MONAI共享相同的原理。
MONAI Label 减少了注释新数据集的时间和精力,并通过不断从用户交互和数据中学习,使 AI 能够适应手头的任务。MONAI Label 允许研究人员和开发人员以用户的方式与他们的应用程序交互,从而不断改进他们的应用程序。最终用户(临床医生、技术人员和一般注释者)受益于 AI 的不断学习,并更好地理解最终用户试图注释的内容。]
Monai Bundle 和 Model Zoo简单介绍
MONAI Model Zoo 提供了一系列由社区开发的医学影像模型,采用 Monai Bundle 格式。Monai Bundle 允许您轻松获取任何来自 MONAI Model Zoo 的模型并将其导入 MONAILabel。
Model Zoo提供的模型包括MRI脑肿瘤分割,CT肺结节检测,病理细胞核分割分类,CT胰腺分割,MRI前列腺分割,CT肾脏分割,CT脾脏分割,MRI脑切片生成等20几个模型。后面会用单独的篇幅分别介绍这些预训练模型的使用。
MONAI Bundle 是一种基于规范和文件结构的方式,用于分发经过训练的 MONAI 模型以及相关元数据、代码、文档和其他资源。这些旨在让您更轻松地以某种格式分发模型,该格式说明了模型的用途、如何使用它、如何重现您用它完成的科学研究,并将其用于 Label 和 Deploy 等其他应用程序。详细的使用教程可以去monai bundle github
接下来我们将用很多案例来演示monai bunlde在3D slicer中的用法
基于monai bundle的MRI全脑分割
模型简介
- 名称:Wholebrainseg large unest segmentation
- 概述:基于3D transformer的全脑分割模型,适用于T1W MRI图像
- 作者:范德堡大学 + MONAI团队
- 参考文献:Xin 等人. 使用3D块聚合变换器表征肾脏结构. arXiv 预印本 arXiv:2203.02430 (2022). https://arxiv.org/pdf/2203.02430.pdf
- 版本:0.2.3
模型描述
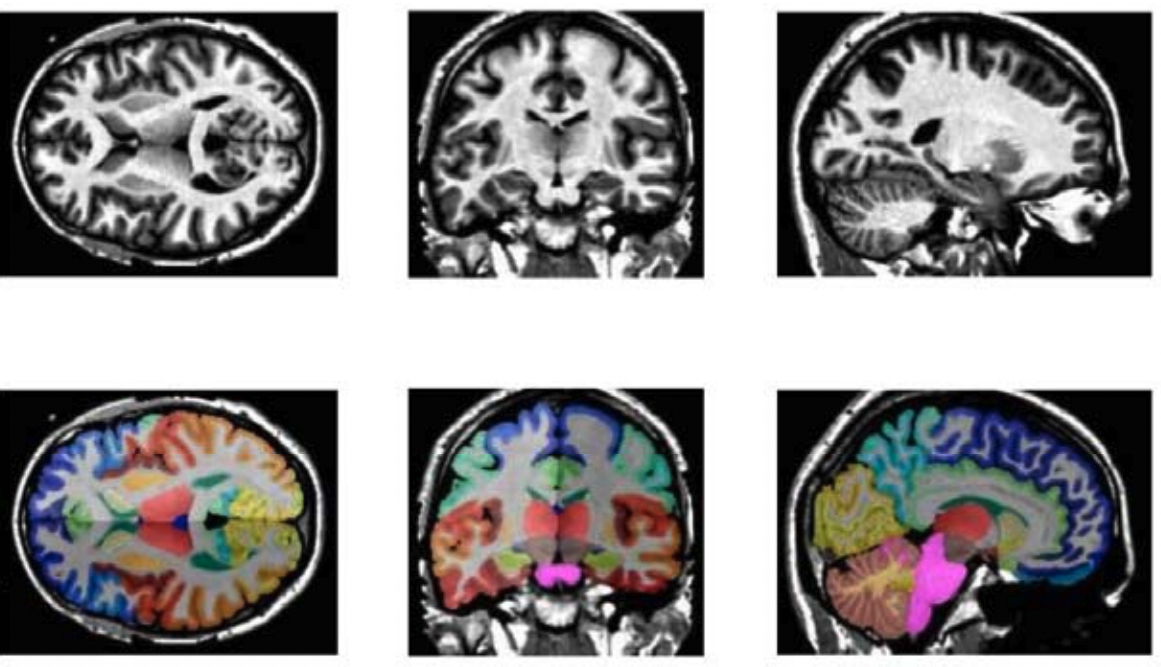
详细的全脑分割是一种重要的定量技术,在医学影像分析中提供了一种非侵入性的方法来测量临床获取的结构性磁共振成像(MRI)中的脑部区域。我们提供了一个预训练模型,用于训练和推理具有133个结构的全脑分割。提供的训练管道支持在 MONAI Label 中的主动学习和使用 bundle 进行训练
模型训练细节
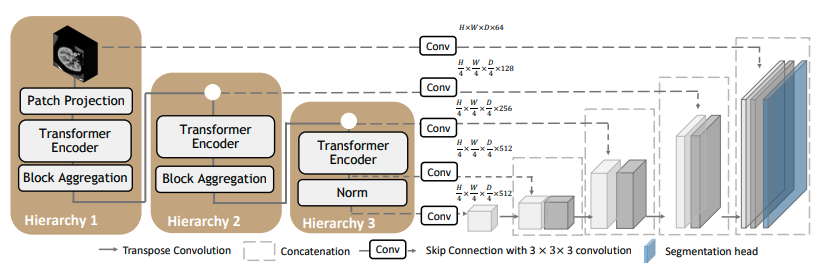
一个预训练的 UNEST base model,用于 T1w MR 图像的体积(3D)全脑分割。为了利用嵌入序列中的信息,提出了“shifted window” transformers,用于密集预测和建模多尺度特征。然而,这些旨在复杂化 self-attention 范围的尝试往往会导致高计算复杂度和数据低效。受 nested ViT 中的 aggregation function 启发,我们提出了一种新的设计,即具有 Nested Transformers (UNesT) 的 3D U-shape 医学分割模型,通过 3D block aggregation function 层次化地学习小结构或小数据集的 locality behaviors。该设计保留了原始的 global self-attention mechanism,并通过层次化堆叠 transformer encoders 实现跨 patches 的信息通信。
训练数据
训练数据来自范德堡大学和范德堡大学医疗中心,并使用了公开发布的 OASIS 和 CANDI 数据集。训练和测试数据是来自三个不同站点的 MRI T1w图像。
任务涉及 133 类全脑分割。在 OASIS 数据集中,50 个 T1w MRI 扫描中,45 个用于训练,5 个用于验证。测试队列包括 Colin27 T1w 扫描和 CANDI 数据集的 13 个 T1w MRI 扫描。所有数据均注册到 MNI 空间,并按特定方法进行预处理,输入图像随机裁剪至 96 × 96 × 96 尺寸。
注意事项
请将你的数据配准到相同空间,训练用的脑部 MRI 图像通过 NiftyReg 工具进行仿射配准,以将目标图像注册到 MNI305 模板。在推理前,数据应处于 MNI305 空间。如果图像已经在 MNI 空间,则可以跳过配准步骤。可以使用任何配准工具将图像注册到 MNI 空间,以下是一个使用 ANTs 的示例建议。
pip install antspyx
#Sample ANTS registration
import ants
import sys
import os
fixed_image = ants.image_read('<fixed_image_path>')
moving_image = ants.image_read('<moving_image_path>')
transform = ants.registration(fixed_image,moving_image,'Affine')
reg3t = ants.apply_transforms(fixed_image,moving_image,transform['fwdtransforms'][0])
ants.image_write(reg3t,output_image_path)
电脑配置
The training and inference was performed with at least one 24GB-memory GPU.
完整的133个脑结构列表
由于列表较长,我放在文末了
在3D slicer使用全脑结构分割模型
- step1: 激活环境
conda activate monailabel
- step2: 下载monaibundle app
monailabel apps --name monaibundle --download --output apps
可以前往图片上地址查看下载了什么
- step3: 下载实验数据
我直接在3D slicer 里面下载的示例数据,保存在了datasets/mrihead文件夹。
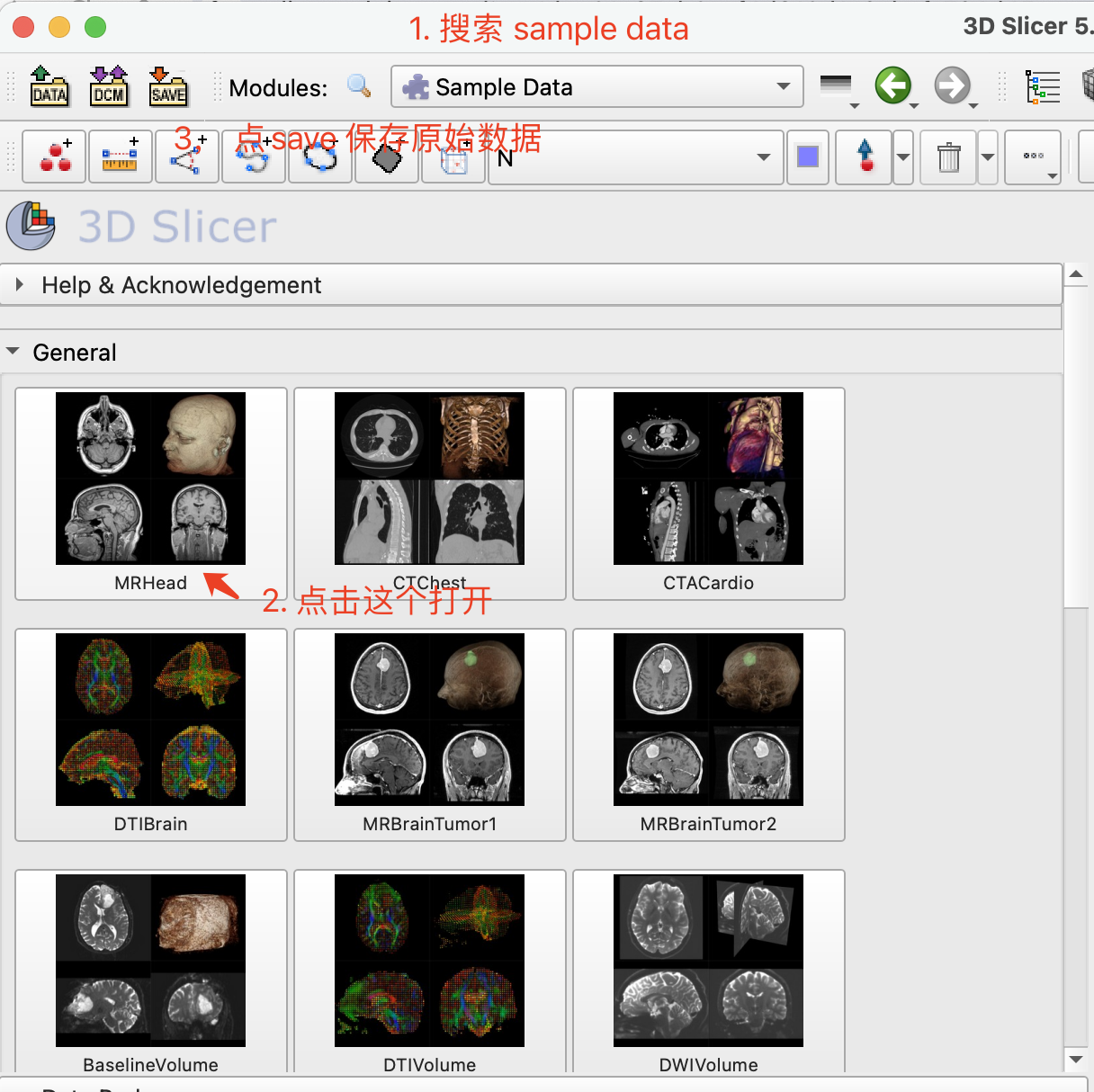
其他的T1w 数据也可以,最好是配准到了标准空间,和训练数据一致,准确度会更高。
- step4: 启动服务端
服务端启动,使用的模型为:wholeBrainSeg_Large_UNEST_segmentation,注意大小写
monailabel start_server --app apps/monaibundle --studies datasets/mrihead --conf models wholeBrainSeg_Large_UNEST_segmentation
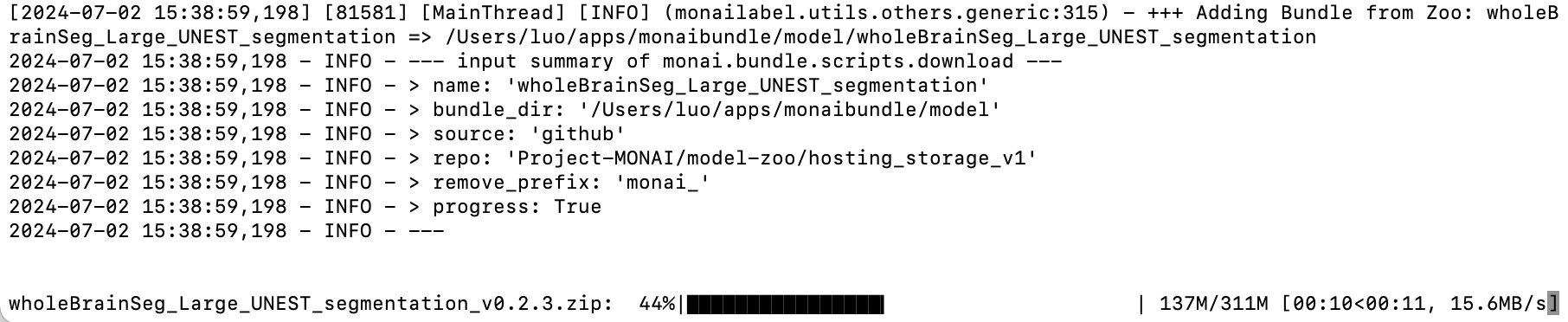
如图,第一次启动的时候会去下载预训练模型。
- step5: 打开3D slicer,连接服务端
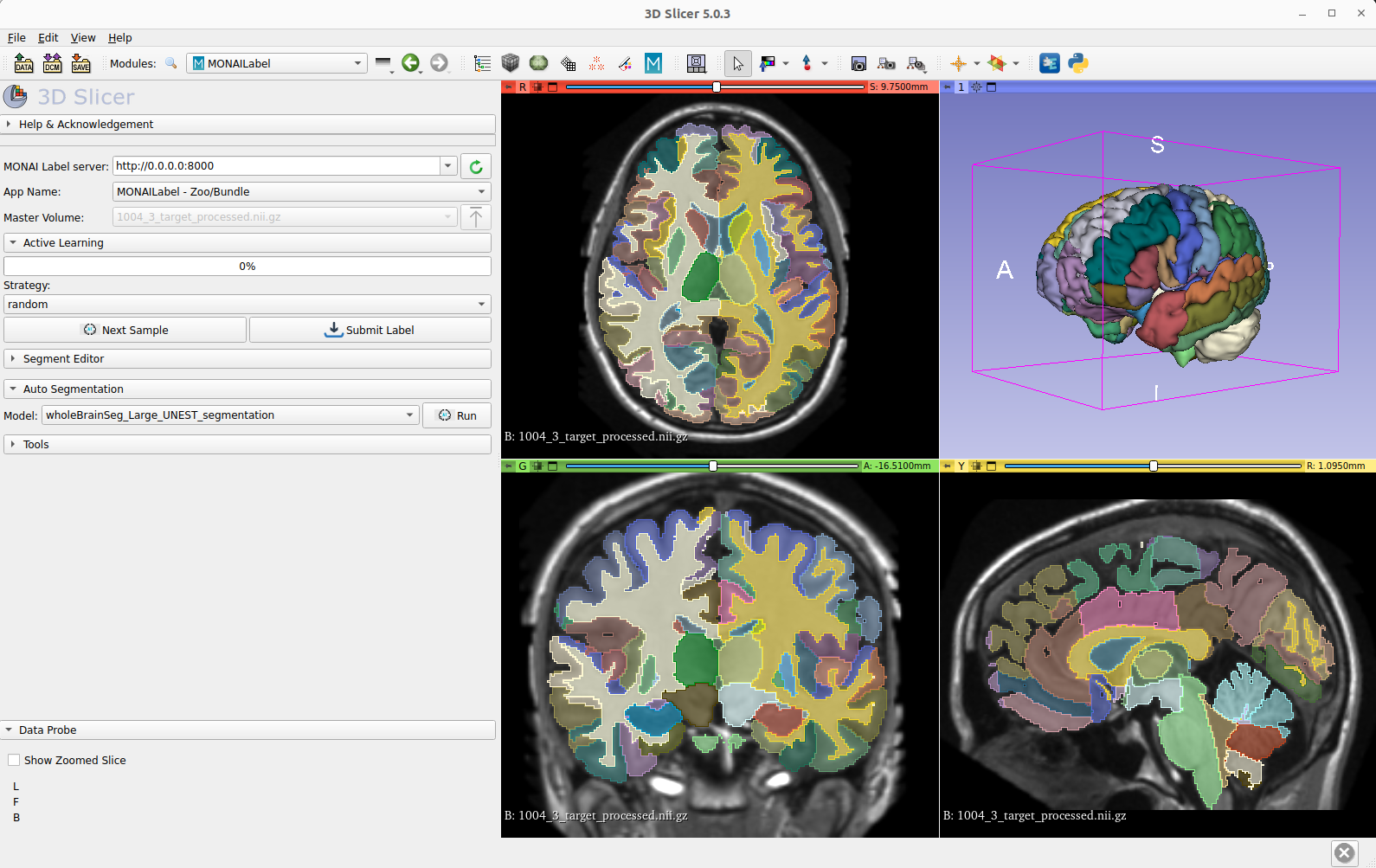
第四步启动好后,打开3D slicer,点击monailabel模块,点击绿色刷新符号就可以连接上服务端了。我们从下图可以看到使用的app和model。点击next sample加载数据,再点击run就可以自动分割了。
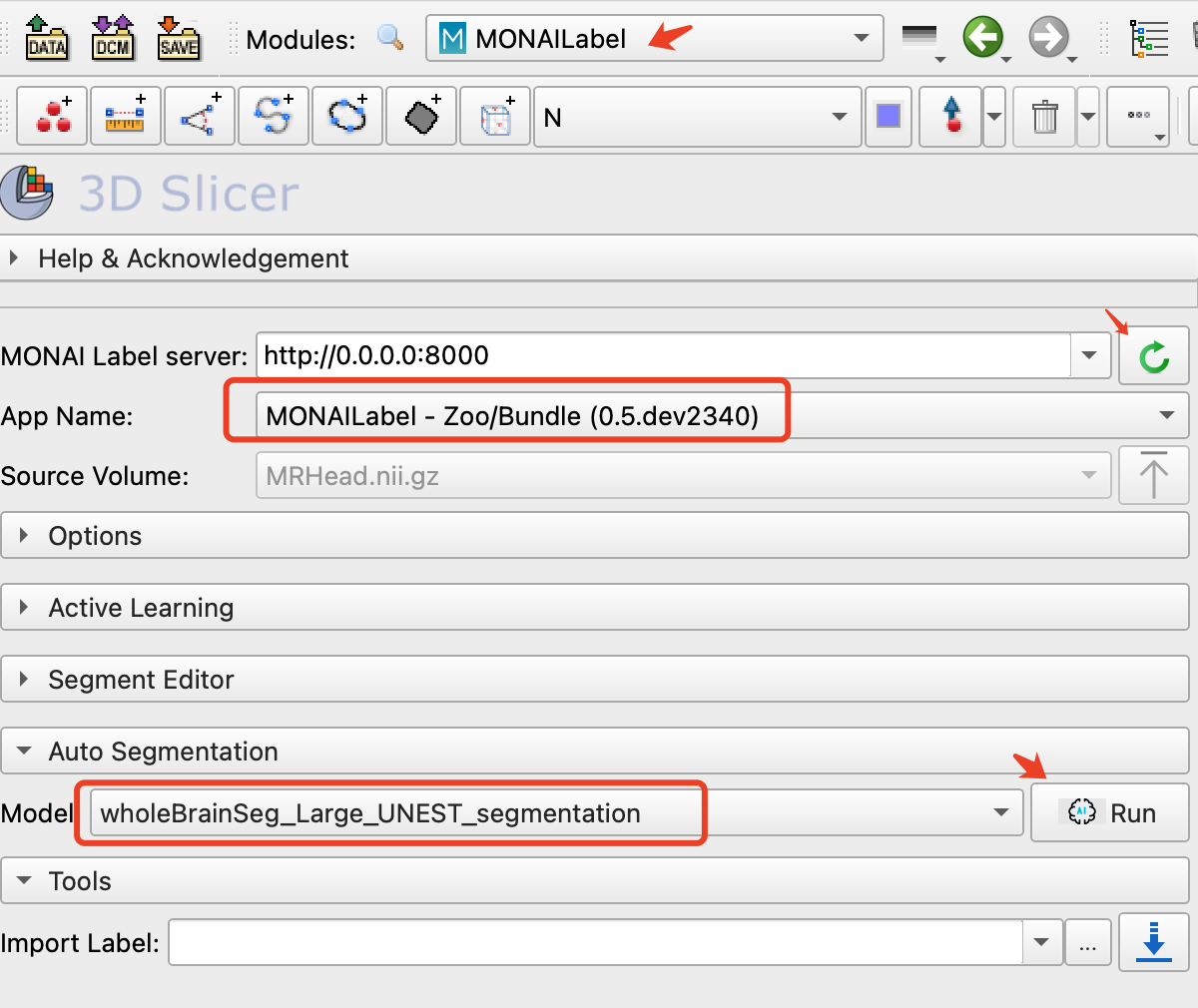
我在mac上分割花了20分钟左右,建议在GPU上运行可以加速。由于我的数据和训练数据差异太大,分割效果一般
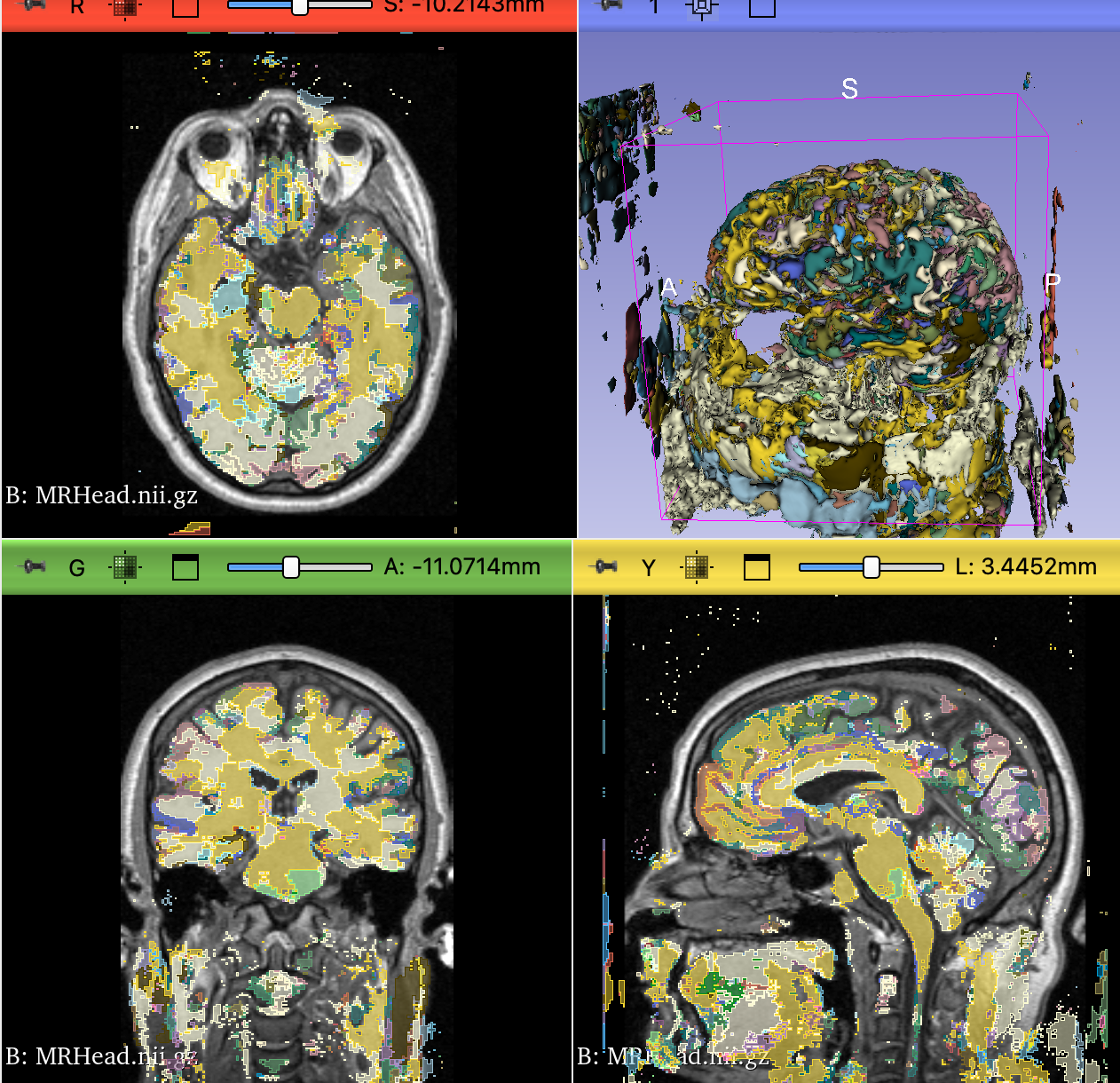
不过,对于脖子部分,可以在segment editor模块中,拿把剪刀剪了。
事实证明,这个预训练模型的泛化性能不是很好
。不过有这个需求的同学可以试试。
其余的保存步骤,训练过程等操作可以查看之前的脾脏分割教程,有详细的介绍。
Tina姐:monai bundle也可以命令行调用,本次只演示了在 3D Slicer中的应用
完整的133个脑结构列表
| 编号 | 英文描述 | 中文翻译 |
|---|---|---|
| 0 | background | 背景 |
| 1 | 3rd-Ventricle | 第三脑室 |
| 2 | 4th-Ventricle | 第四脑室 |
| 3 | Right-Accumbens-Area | 右侧伏隔核区 |
| 4 | Left-Accumbens-Area | 左侧伏隔核区 |
| 5 | Right-Amygdala | 右侧杏仁核 |
| 6 | Left-Amygdala | 左侧杏仁核 |
| 7 | Brain-Stem | 脑干 |
| 8 | Right-Caudate | 右侧尾状核 |
| 9 | Left-Caudate | 左侧尾状核 |
| 10 | Right-Cerebellum-Exterior | 右侧小脑皮层 |
| 11 | Left-Cerebellum-Exterior | 左侧小脑皮层 |
| 12 | Right-Cerebellum-White-Matter | 右侧小脑白质 |
| 13 | Left-Cerebellum-White-Matter | 左侧小脑白质 |
| 14 | Right-Cerebral-White-Matter | 右侧大脑白质 |
| 15 | Left-Cerebral-White-Matter | 左侧大脑白质 |
| 16 | Right-Hippocampus | 右侧海马 |
| 17 | Left-Hippocampus | 左侧海马 |
| 18 | Right-Inf-Lat-Vent | 右侧下外侧脑室 |
| 19 | Left-Inf-Lat-Vent | 左侧下外侧脑室 |
| 20 | Right-Lateral-Ventricle | 右侧侧脑室 |
| 21 | Left-Lateral-Ventricle | 左侧侧脑室 |
| 22 | Right-Pallidum | 右侧苍白球 |
| 23 | Left-Pallidum | 左侧苍白球 |
| 24 | Right-Putamen | 右侧壳核 |
| 25 | Left-Putamen | 左侧壳核 |
| 26 | Right-Thalamus-Proper | 右侧丘脑 |
| 27 | Left-Thalamus-Proper | 左侧丘脑 |
| 28 | Right-Ventral-DC | 右侧腹侧DC |
| 29 | Left-Ventral-DC | 左侧腹侧DC |
| 30 | Cerebellar-Vermal-Lobules-I-V | 小脑蚓部叶I-V |
| 31 | Cerebellar-Vermal-Lobules-VI-VII | 小脑蚓部叶VI-VII |
| 32 | Cerebellar-Vermal-Lobules-VIII-X | 小脑蚓部叶VIII-X |
| 33 | Left-Basal-Forebrain | 左侧基底前脑 |
| 34 | Right-Basal-Forebrain | 右侧基底前脑 |
| 35 | Right-ACgG–anterior-cingulate-gyrus | 右侧前扣带回 |
| 36 | Left-ACgG–anterior-cingulate-gyrus | 左侧前扣带回 |
| 37 | Right-AIns–anterior-insula | 右侧前岛叶 |
| 38 | Left-AIns–anterior-insula | 左侧前岛叶 |
| 39 | Right-AOrG–anterior-orbital-gyrus | 右侧前眶回 |
| 40 | Left-AOrG–anterior-orbital-gyrus | 左侧前眶回 |
| 41 | Right-AnG—angular-gyrus | 右侧角回 |
| 42 | Left-AnG—angular-gyrus | 左侧角回 |
| 43 | Right-Calc–calcarine-cortex | 右侧距状皮质 |
| 44 | Left-Calc–calcarine-cortex | 左侧距状皮质 |
| 45 | Right-CO----central-operculum | 右侧中央盖 |
| 46 | Left-CO----central-operculum | 左侧中央盖 |
| 47 | Right-Cun—cuneus | 右侧楔叶 |
| 48 | Left-Cun—cuneus | 左侧楔叶 |
| 49 | Right-Ent—entorhinal-area | 右侧内嗅区 |
| 50 | Left-Ent—entorhinal-area | 左侧内嗅区 |
| 51 | Right-FO----frontal-operculum | 右侧额盖 |
| 52 | Left-FO----frontal-operculum | 左侧额盖 |
| 53 | Right-FRP—frontal-pole | 右侧额极 |
| 54 | Left-FRP—frontal-pole | 左侧额极 |
| 55 | Right-FuG—fusiform-gyrus | 右侧颞下回 |
| 56 | Left-FuG—fusiform-gyrus | 左侧颞下回 |
| 57 | Right-GRe—gyrus-rectus | 右侧直回 |
| 58 | Left-GRe—gyrus-rectus | 左侧直回 |
| 59 | Right-IOG—inferior-occipital-gyrus | 右侧下枕回 |
| 60 | Left-IOG—inferior-occipital-gyrus | 左侧下枕回 |
| 61 | Right-ITG—inferior-temporal-gyrus | 右侧下颞回 |
| 62 | Left-ITG—inferior-temporal-gyrus | 左侧下颞回 |
| 63 | Right-LiG—lingual-gyrus | 右侧舌回 |
| 64 | Left-LiG—lingual-gyrus | 左侧舌回 |
| 65 | Right-LOrG–lateral-orbital-gyrus | 右侧外侧眶回 |
| 66 | Left-LOrG–lateral-orbital-gyrus | 左侧外侧眶回 |
| 67 | Right-MCgG–middle-cingulate-gyrus | 右侧中扣带回 |
| 68 | Left-MCgG–middle-cingulate-gyrus | 左侧中扣带回 |
| 69 | Right-MFC—medial-frontal-cortex | 右侧内侧额叶皮层 |
| 70 | Left-MFC—medial-frontal-cortex | 左侧内侧额叶皮层 |
| 71 | Right-MFG—middle-frontal-gyrus | 右侧中额回 |
| 72 | Left-MFG—middle-frontal-gyrus | 左侧中额回 |
| 73 | Right-MOG—middle-occipital-gyrus | 右侧中枕回 |
| 74 | Left-MOG—middle-occipital-gyrus | 左侧中枕回 |
| 75 | Right-MOrG–medial-orbital-gyrus | 右侧内侧眶回 |
| 76 | Left-MOrG–medial-orbital-gyrus | 左侧内侧眶回 |
| 77 | Right-MPoG–postcentral-gyrus | 右侧后中央回 |
| 78 | Left-MPoG–postcentral-gyrus | 左侧后中央回 |
| 79 | Right-MPrG–precentral-gyrus | 右侧前中央回 |
| 80 | Left-MPrG–precentral-gyrus | 左侧前中央回 |
| 81 | Right-MSFG–superior-frontal-gyrus | 右侧上额回 |
| 82 | Left-MSFG–superior-frontal-gyrus | 左侧上额回 |
| 83 | Right-MTG—middle-temporal-gyrus | 右侧中颞回 |
| 84 | Left-MTG—middle-temporal-gyrus | 左侧中颞回 |
| 85 | Right-OCP—occipital-pole | 右侧枕极 |
| 86 | Left-OCP—occipital-pole | 左侧枕极 |
| 87 | Right-OFuG–occipital-fusiform-gyrus | 右侧枕颞回 |
| 88 | Left-OFuG–occipital-fusiform-gyrus | 左侧枕颞回 |
| 89 | Right-OpIFG-opercular-part-of-the-IFG | 右侧岛状前额叶盖部 |
| 90 | Left-OpIFG-opercular-part-of-the-IFG | 左侧岛状前额叶盖部 |
| 91 | Right-OrIFG-orbital-part-of-the-IFG | 右侧岛状前额叶眶部 |
| 92 | Left-OrIFG-orbital-part-of-the-IFG | 左侧岛状前额叶眶部 |
| 93 | Right-PCgG–posterior-cingulate-gyrus | 右侧后扣带回 |
| 94 | Left-PCgG–posterior-cingulate-gyrus | 左侧后扣带回 |
| 95 | Right-PCu—precuneus | 右侧前楔叶 |
| 96 | Left-PCu—precuneus | 左侧前楔叶 |
| 97 | Right-PHG—parahippocampal-gyrus | 右侧海马旁回 |
| 98 | Left-PHG—parahippocampal-gyrus | 左侧海马旁回 |
| 99 | Right-PIns–posterior-insula | 右侧后岛叶 |
| 100 | Left-PIns–posterior-insula | 左侧后岛叶 |
| 101 | Right-PO----parietal-operculum | 右侧顶盖 |
| 102 | Left-PO----parietal-operculum | 左侧顶盖 |
| 103 | Right-PoG—postcentral-gyrus | 右侧后中央回 |
| 104 | Left-PoG—postcentral-gyrus | 左侧后中央回 |
| 105 | Right-POrG–posterior-orbital-gyrus | 右侧后眶回 |
| 106 | Left-POrG–posterior-orbital-gyrus | 左侧后眶回 |
| 107 | Right-PP----planum-polare | 右侧极平面 |
| 108 | Left-PP----planum-polare | 左侧极平面 |
| 109 | Right-PrG—precentral-gyrus | 右侧前中央回 |
| 110 | Left-PrG—precentral-gyrus | 左侧前中央回 |
| 111 | Right-PT----planum-temporale | 右侧颞平面 |
| 112 | Left-PT----planum-temporale | 左侧颞平面 |
| 113 | Right-SCA—subcallosal-area | 右侧次眶区 |
| 114 | Left-SCA—subcallosal-area | 左侧次眶区 |
| 115 | Right-SFG—superior-frontal-gyrus | 右侧上额回 |
| 116 | Left-SFG—superior-frontal-gyrus | 左侧上额回 |
| 117 | Right-SMC—supplementary-motor-cortex | 右侧辅助运动皮层 |
| 118 | Left-SMC—supplementary-motor-cortex | 左侧辅助运动皮层 |
| 119 | Right-SMG—supramarginal-gyrus | 右侧缘上回 |
| 120 | Left-SMG—supramarginal-gyrus | 左侧缘上回 |
| 121 | Right-SOG—superior-occipital-gyrus | 右侧上枕回 |
| 122 | Left-SOG—superior-occipital-gyrus | 左侧上枕回 |
| 123 | Right-SPL—superior-parietal-lobule | 右侧上顶叶 |
| 124 | Left-SPL—superior-parietal-lobule | 左侧上顶叶 |
| 125 | Right-STG—superior-temporal-gyrus | 右侧上颞回 |
| 126 | Left-STG—superior-temporal-gyrus | 左侧上颞回 |
| 127 | Right-TMP—temporal-pole | 右侧颞极 |
| 128 | Left-TMP—temporal-pole | 左侧颞极 |
| 129 | Right-TrIFG-triangular-part-of-the-IFG | 右侧三角前额叶 |
| 130 | Left-TrIFG-triangular-part-of-the-IFG | 左侧三角前额叶 |
| 131 | Right-TTG—transverse-temporal-gyrus | 右侧横颞回 |
| 132 | Left-TTG—transverse-temporal-gyrus | 左侧横颞回 |
文章持续更新,可以关注微公【医学图像人工智能实战营】获取最新动态。人手有限,文中涉及的链接前往微公对应文章查看。关注Tina姐,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连























 1089
1089

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










