今天看完这个校正方法,基本上就是一天过去了,单细胞组学数据发展迅速,尤其是现在获得的实验数据在捕获时间、操作人员、试剂批次、仪器设备甚至技术平台方面都存在差异。这些差异会导致数据中出现较大的差异或批次效应,并可能在数据整合过程中混淆感兴趣的生物变异。因此,有效消除批次效应至关重要。批次效应可能具有高度非线性,因此很难在保留关键生物变异的同时正确对齐不同的数据集。分享几种常用的批次效应校正方法,时刻提醒自己在写流程的时候要综合考虑使用哪一个校正方法对数据进行预处理。
原文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-019-1850-9#Sec13。
批次效应校正方法:
总结了最近十年的批次效应校正方法,方法包括:MNN correct、Harmony、Combat、fastMNN、limma、scGen、Seurat2、Seurat3、Scanorama、MND-ResNet、ZINB-WaVE、scMerge、LIGER、BBKNN、BUS、BEER、scVI、BERMUDA、DESC、ResPAN、fRMA、SCAN、SVA。
图中展示了使用的编程语言、批次效应校正后输出的结果、使用方法以及参考文献。
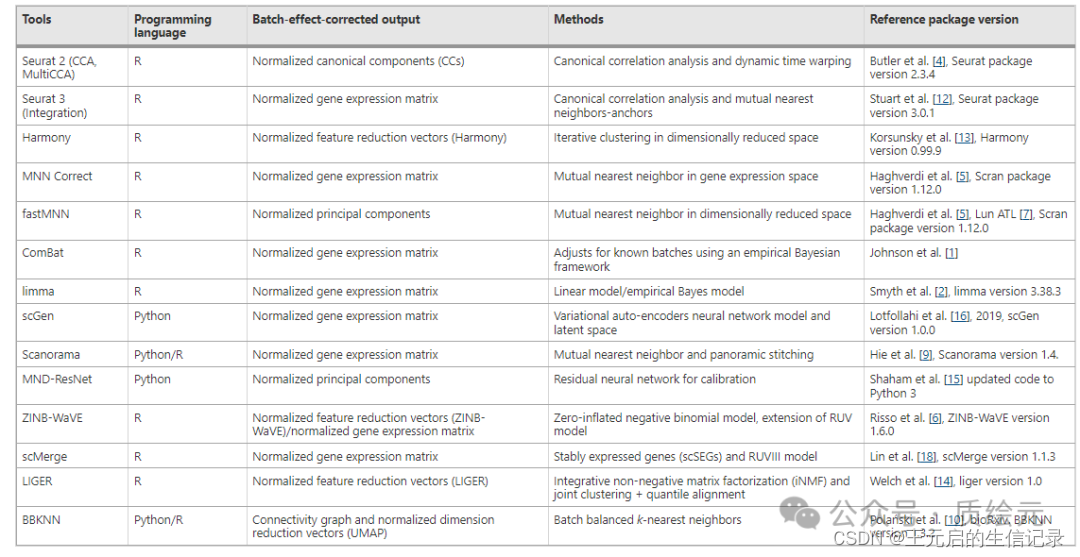
在原文的基础上补充了几个新的批次效应矫正方法

方法分类:
将上述方法进行分类,可以分为以下四类,此部分转自生信自媒体大佬曾健明,原文链接:单细胞测序最好的教程(七): 数据整合与批次效应校正 - Starlitnightly - 博客园 (cnblogs.com)。
全局模型: 源自bulk RNA-seq,将批次效应建模为所有细胞中存在的(加法/或乘法)效应。一个常见的例子是 ComBat
线性嵌入模型: 是第一个单细胞特异性批量去除方法。这些方法通常使用奇异值分解 (SVD) 的变体来嵌入数据,然后在嵌入中跨批次查找相似单元的局部邻域,并使用它们以局部自适应(非线性)方式校正批次效应。常见的例子包括最近邻MNN,Seurat,Harmony,Scanorama,FastMNN等
基于图的模型: 通常是运行速度最快的方法。使用最近邻域图来表示每个批次的数据。通过强制连接不同批次的细胞,然后修建细胞类型组成的差异的图的边缘,可以纠正批次效应。一个常见的例子是BBKNN
深度学习模型: 大多数深度学习批次效应校正方法都基于自动编码器网络,并且要么在条件变分自动编码器(CVAE)中对批量协变量进行降维,要么在嵌入空间中拟合局部线性校正。
校正评价指标:
可以根据以下几个指标对上述方法进行评估,通过原文比较,Harmony
方法在多个数据集上表现较好,其他方法自行考虑使用。

Average silhouette width (ASW): 平均轮廓宽度。这是一种用于评估聚类质量的指标,用于衡量细胞在不同批次中是否能够被正确地分开
Graph integration local inverse Simpson’s Index (graph iLISI): 图集成局部倒数Simpson指数。这似乎是一种用于比较不同图谱(可能是从不同批次收集的)之间差异的方法,可能是通过比较细胞类型或样本之间的相似性来完成的。
graph cLISI: cLISI。可能是一种用于衡量标签保守性的统计方法,可能与细胞之间的相似性有关。
k-nearest-neighbor batch effect test (kBET): k最近邻批次效应检验。这是一种用于衡量数据集中不同批次之间是否存在批次效应的统计方法。
k-nearest-neighbor (kNN) graph connectivity: k最近邻图连通性。这可能涉及构建一个基于细胞之间相似性的图,以衡量不同批次之间的连接性。
Isolated label scores: 孤立标签分数。用于评估罕见细胞身份标签的指标。
Normalized Mutual Information (NMI): 归一化互信息。用于测量两个随机变量之间关联度的度量。在这里我们测量不同batch校正后细胞类型的一致性。
Adjusted Rand Index (ARI): 调整兰德指数。一种用于比较两个数据分区的相似度的指标。
祝大家周末愉快,这周的班就先上到这里,下周再见!
对上述方法有疑问的可以联系管理员:kriswcyYQ!
























 7754
7754

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








