前面我们大量NGS相关教程视频免费发布在B站,都是使用NCBI的SRA数据库下载sra文件后转为fastq进行NGS分析流程,其实是因为我本人一直不在中国大陆,所以没有网络问题。但是学生们不一样,同样的命令他们prefetch的下载比蜗牛还慢,即使加上aspera后也会面临sra文件转为fastq的限速。所以我们在全国巡讲的答疑群给大家指点的解决方案是使用aspera从EBI下载直接fastq数据,一劳永逸。
现在把这个技巧分享给大家,让我们的讲师助教团队总结了经验如下:

使用`ascp`从EBI下载fastq数据
mkdir -p /data/project/pig_lncRNA && cd /data/project/pig_lncRNA
mkdir -p 1.raw_fq && cd 1.raw_fq
ENA主页:https://www.ebi.ac.uk/ena/browser/home
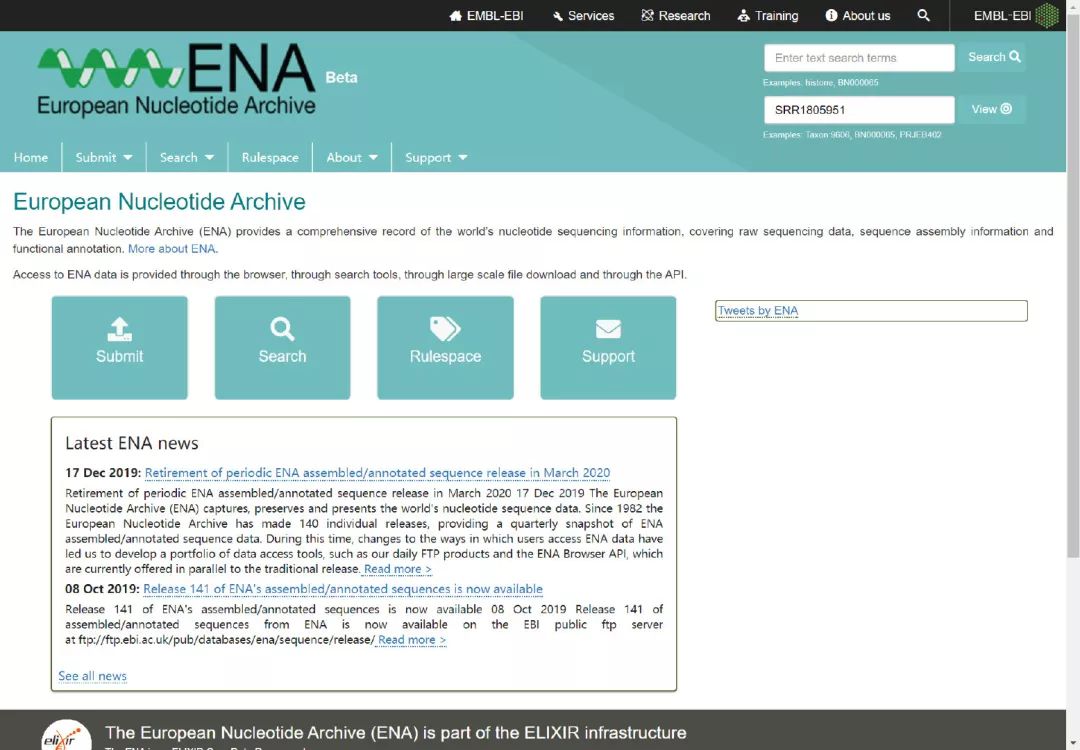
随便搜索SRR1805951
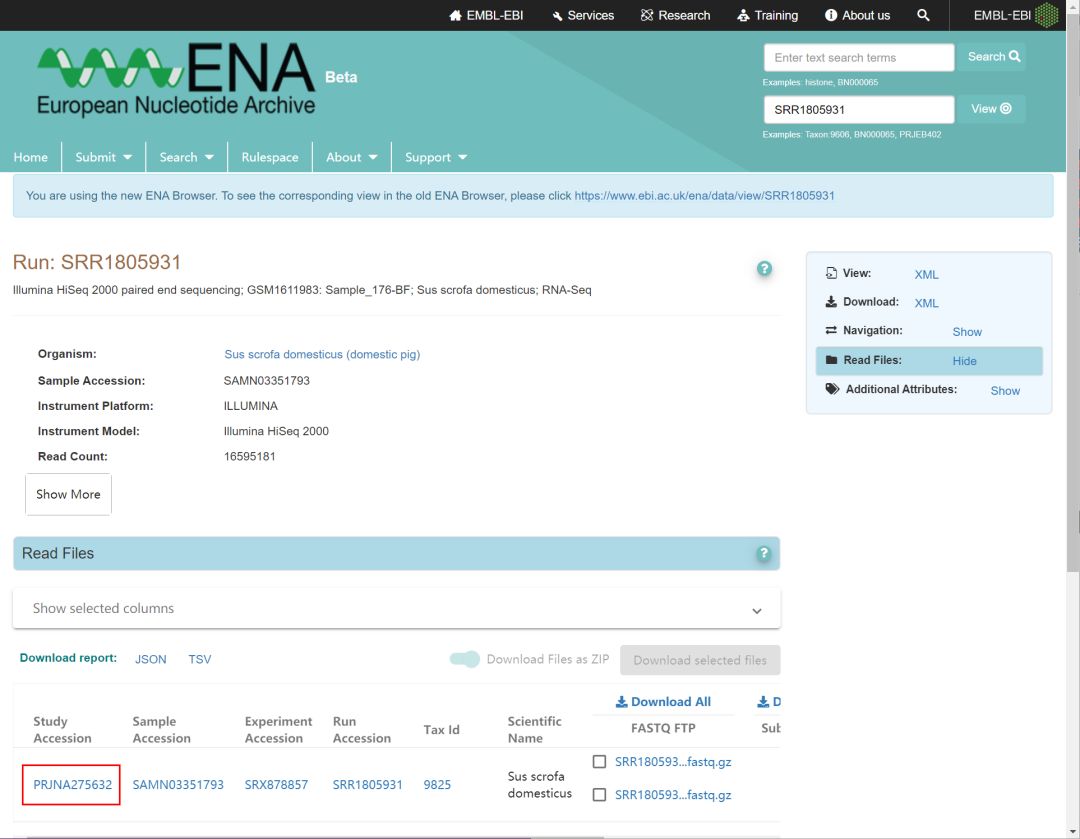
点击PRJNA275632
这里可以看到整个数据集所有样本的fastq下载地址,随便挑几个,观察一下:
ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR180/009/SRR1805929/SRR1805929_1.fastq.gzftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR180/009/SRR1805929/SRR1805929_2.fastq.gzftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR180/000/SRR1805930/SRR1805930_1.fastq.gzftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR180/000/SRR180593








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章















 392
392











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








