2018-Bioinformatics-DeepDTA: deep drug-target binding affinity prediction

Paper: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6129291/
Code: https://github.com/hkmztrk/DeepDTA
DeepDTA: 深度药物-靶点结合亲和力预测
模型
输入表示
- 蛋白质序列:40D张量(8D蛋白质二级结构特征;11D理化性质特征;21D氨基酸字符特征)
- 蛋白质口袋序列特征:40D张量(每一维的特征与蛋白质序列相同),蛋白质-配体相互作用主要取决于配体与蛋白口袋之间的结合,蛋白质口袋由一个不连续的序列组成,包括了蛋白质的一些关键氨基酸,因此将口袋作为一个整体来进行局部特征的提取。
- 配体分子SMILES字符特征:64D one-hot编码,包括了组成化合物SMILES码的64种字符。
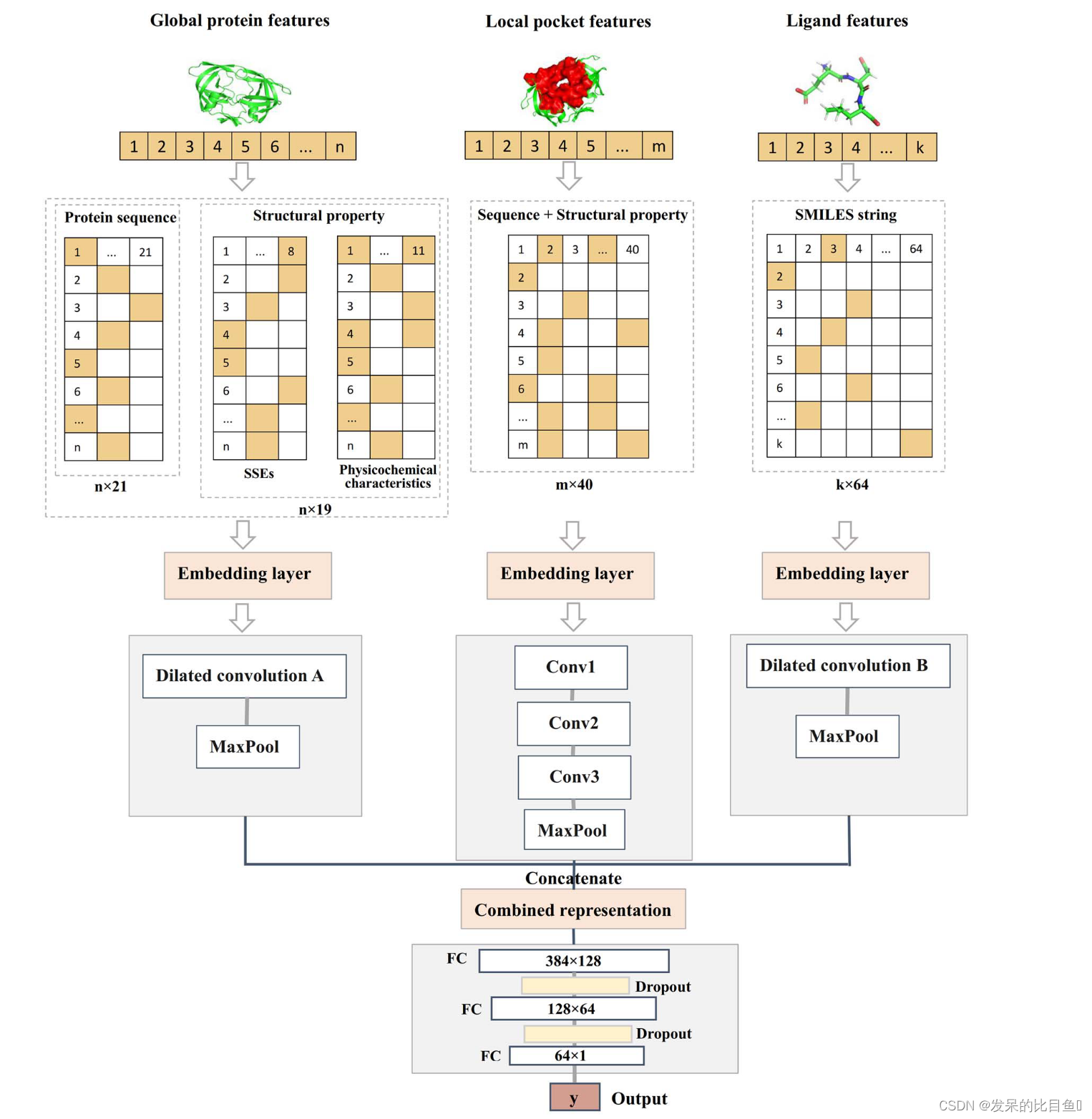
对于蛋白质序列,考虑到较长蛋白质序列的长程相互作用,使用了五层膨胀率分别为1,2,4,8,16的3x3卷积核;对于配体化合物SMILES序列,使用了四层膨胀率为1,2,4,8的3x3卷积核。即上图中的Dilated convolution A和Dilated convolution B。
数据集
Davis: 442蛋白-68配体,30056对相互作用,
k
d
=
−
l
o
g
(
k
d
1
e
9
)
kd = -log(\frac{k_d}{1e9})
kd=−log(1e9kd)
KIBA :过滤至少10对相互作用, 最终有229蛋白 - 2111药物,118254对相互作用
PDBbind: 里面包含了用-logki,-logkd和-logic50表示的经实验验证的蛋白质-配体结合亲和力数据。
结果
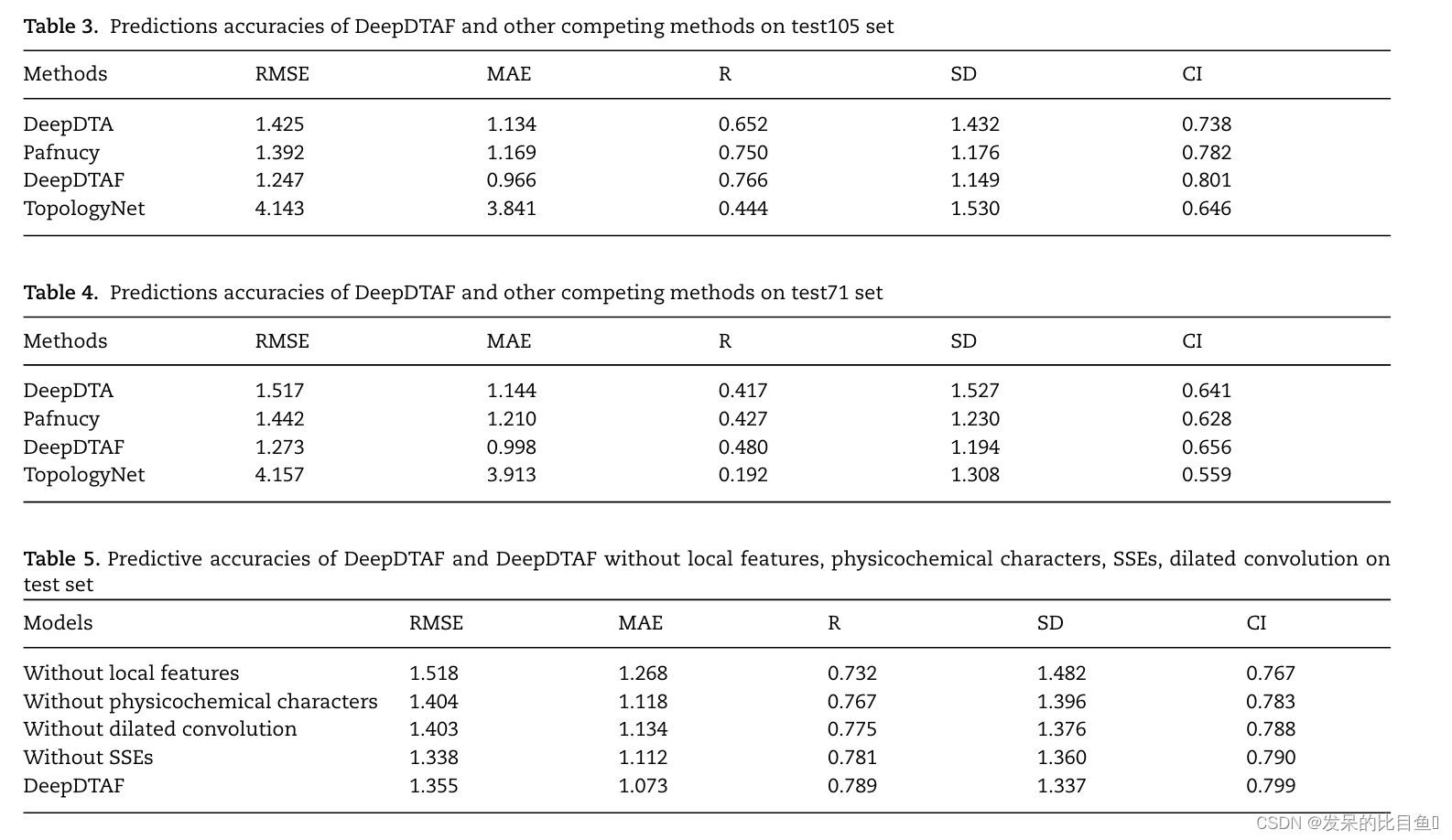
与基线模型的比较结果:























 3241
3241











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










