
TCGA | GEO | 文献阅读 | 数据库 | 理论知识
R语言 | Bioconductor | 服务器与Linux

接前文:
分子对接教程 | (6) AutoDock对接操作与对接结果解读
在介绍之前,先简单介绍一下这个软件,虽然前面我们简单的使用,但没有过多介绍,这里就简单介绍一下,具体细节上的东西,需要你不断使用,才能熟悉。下面是软件界面。

1.设定工作目录,打开文件
打开PyMOL软件,设置我们的工作目录。

或者通过命令cd F:\AutoDock来实现,这与window的dos命令行和Linux系统的cd(Change Directory)命令一样。

导入刚才下载的复合物PDB文件(1e8y.pdb)。
注意:有两种方法导入文件,一种是鼠标点击菜单栏File -> Open…,另一种是通过命令行。通过load命令加载:load 1e8y.pdb,因为这样可以同时把工作目录设置好,方便后续保存文件。
假设我们的工作目录为F:\AutoDock。注意:路径中不要有中文和空格,这是导致很多计算类软件(尤其是免费软件)出现问题的原因。
如果是从PDB网站上下载蛋白,如 1e8y.pdb, 也可以直接通过PyMOL 下载蛋白,点击菜单栏中的 File->Get PDB,或者通过fetch 命令下载,这在前面有介绍。
2.对象属性操作介绍
加载到蛋白后。
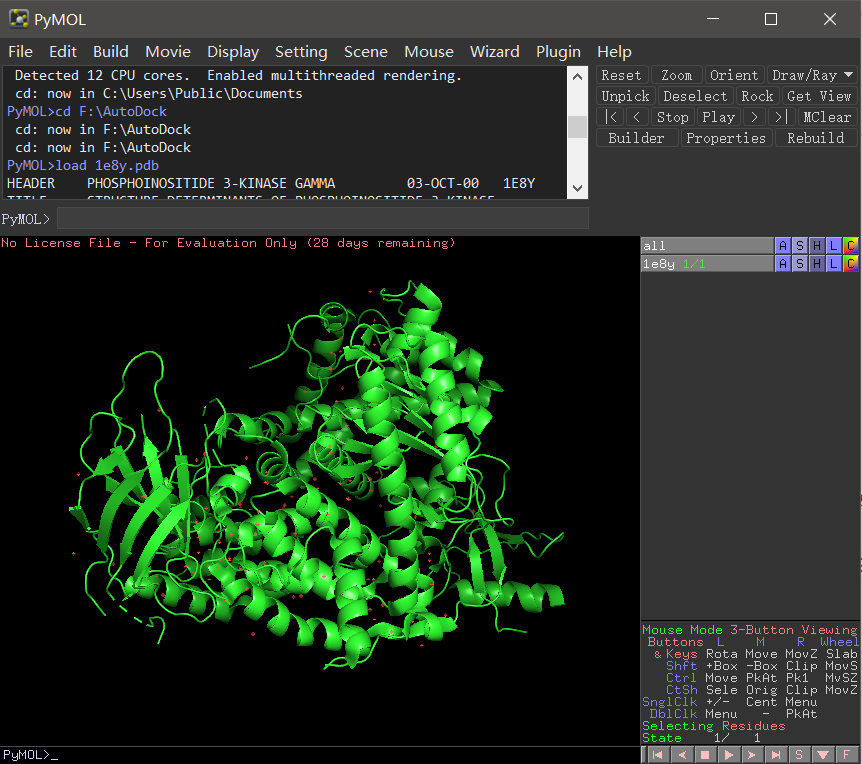
1个是all, all 不是真实的object,它代表了所有的object
1个是1e8y ,1e8y就是我们刚刚载入的蛋白
每个object 都有对应的A S H L C操作:
A:代表Action ,主要包含了对object的常用操作的集合,如 复制、删除object,对object加氢,展示Object等,
S:代表Show ,将object 渲染成cartoon 、line、stick lines sphere surface mesh dots ribbon 等模式
H:Hide 根据object的状态或者描述进行相应的掩藏
L:Label 显示object中残基、原子等名称或者属性
C:Color 对Object 进行着色
知道这些,里面的内容多试试几次就知道怎么回事啦。比如下面2种操作。
点击 A->preset->simple 显示蛋白的简单形式

点击 A->preset->ball and stick 显示球棍模型

再如下面的一些操作介绍;
L->residue 在α碳原子上标记其残基名字和编号 (常用)
L->residue name 在所有原子上标记残基名字 (不常用)
L->clear 删除该对象上所有的Label
L->element symbol 显示对象上所有原子的元素名字
L->vdw radius 看原子的范德华半径
3.可视化窗口操作
平移,按住鼠标中键不放,然后上下左右移动,进行体会,蛋白会随着鼠标而移动
旋转,按住鼠标左键不放,然后上下左右移动鼠标,蛋白会进行旋转
缩放,按住鼠标右键不放,然后上下移动,蛋白会进行缩放
切割 滚动鼠标中键, 建议将蛋白渲染成surface模式,然后滚动鼠标中键
多的不介绍了,B站有很多视频教程,这里我给大家找到了一个文本教程。http://pymol.chenzhaoqiang.com/ ,对作者的贡献表示感谢。
下面我们就简单处理一下我们前面对接的结果
4.对接结果简单处理演示
我们前面对接的结果文件,result.pdbqt,我们同样用OpenBabel这个软件转换成pdb格式。

我们用pymol打开result.pdb文件,在右下角处Selecting 处点击切换到Chains,然后选中蛋白质,在左上角sele处选择A,然后选择rename selection,然后在左上角的窗口处会弹出一行,在:后面输入名称。我这里是protein。或者通过命令set_name sele,protein实现。

我下面通过命令set_name protein,p又将protein改为p。

为了更好的区分受体和配体,我们通过C(color)来更改,根据自己喜好。

Selecting 处切换到残基。

然后选择小分子,右侧又多了一个sele,我们同样可以更改名字。

我这里把名称改成了ligand,我们选中小分子,按下图选择,让小分子显示氢键。

我们鼠标旋转,可以看见4个氢键。
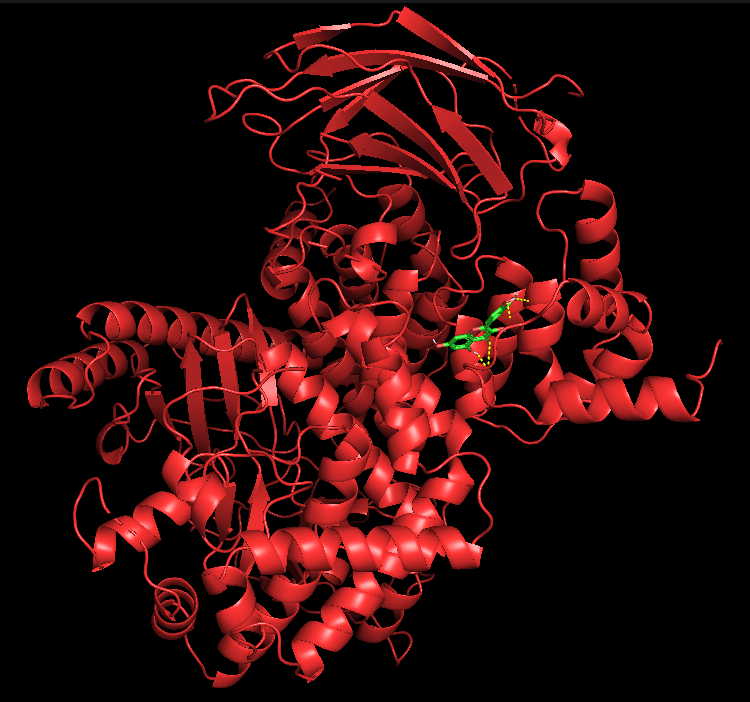
接下来我们显示这4个氢键对接在氨基酸上的那几个残基上。首先,点击蛋白质(p)的S,点击sticks。

然后点击ligand的A,点击center,将配体小分子设置为中心。后面可以通过鼠标放大缩小旋转都以它为中心。

然后我们放大(长按鼠标右键拖动),旋转到合适角度,可以看见。

然后鼠标选中对接的残基

同样将残基改一个名字,我这里是rr。
在p中的H点击sticks,将蛋白的棍状结构隐藏起来。

然后是在rr中选择S里面的sticks,单独显示残基的棍状结构。
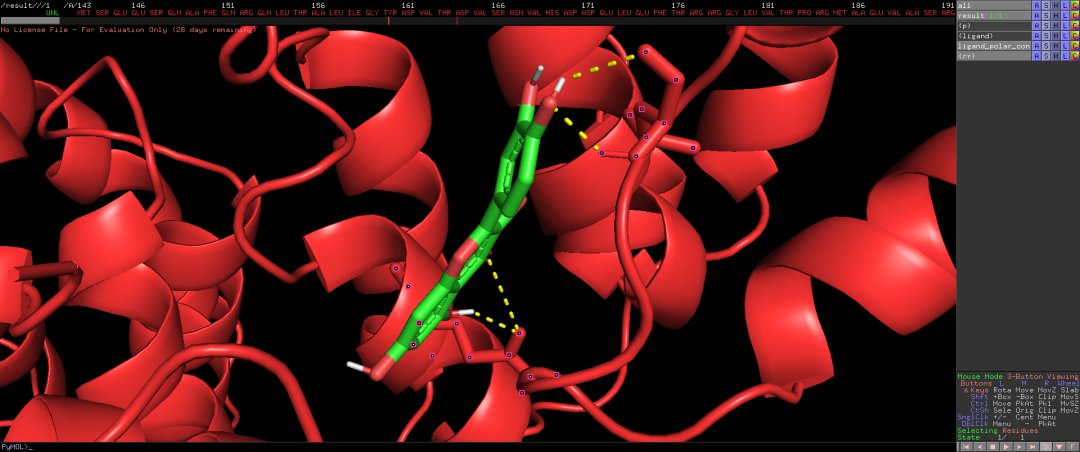
我们可以给残基换一个颜色(C)。

改变背景色,这里改为白色,同时我再把残基改成了蓝色。

可以保存文件编辑好的图片。

至于美化,这里远远不够,就得去自己好好研究这个软件怎么使用了,反正上面的教程也挺详细的,后续在介绍另外一个软件VMD。
参考:
http://pymol.chenzhaoqiang.com/
























 582
582

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








