欢迎关注”生信修炼手册”!
TwoSampleMR是MR-Base数据库开发团队提供的R包,可以调用MR-Base数据库中已有的gwas结果,来进行2SMR分析,官方文档链接如下
https://mrcieu.github.io/TwoSampleMR/
2SMR分析需要两个输入文件,第一个文件为遗传变异与暴露因素的gwas结果,第二个文件为遗传变异与结局变量的gwas结果。对于暴露因素相关的gwas结果,TwoSampleMR支持读取自定义的结果,同时也支持直接调用MR-Base中的结果;对于结局变量相关的gwas结果,仅支持调用MR-Base中的结果。
分析的pipeline示意如下
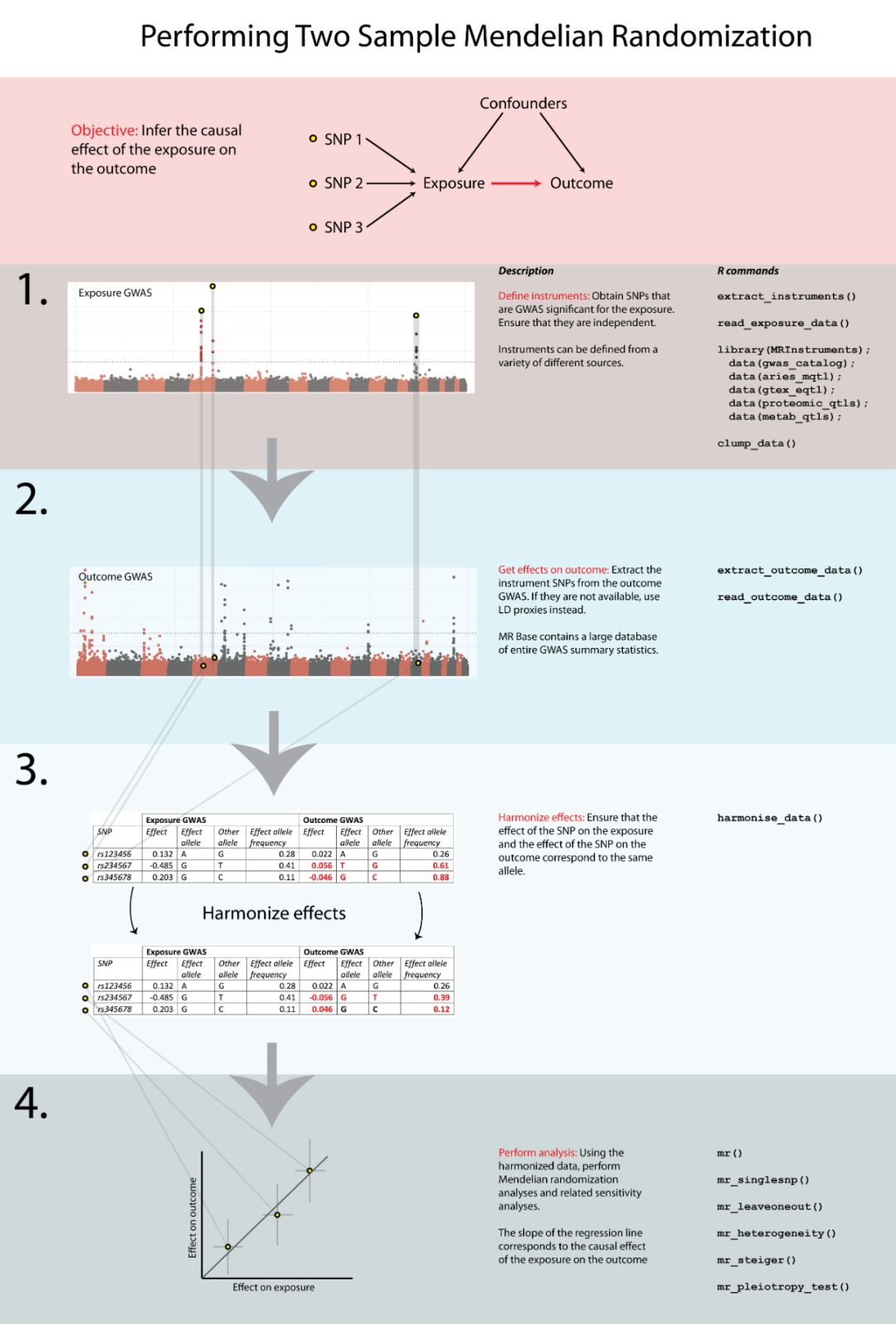
分为了以下4大步
1. read exposure data
读取暴露因素的gwas结果,支持自定义,文件内容示意如下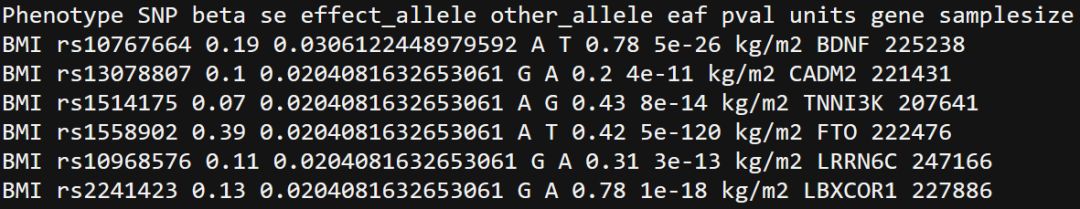
对于上述文件,读取的代码如下
exposure_dat <- read_exposure_data(exp_file)2. read outcome data
读取结局变量的gwas结果,仅支持读取MR-base数据库中的gwas结果,需要google账号,读取的代码如下
ao <- available_outcomes()
outcome_dat <- extract_outcome_data(
snps=exposure_dat$SNP,
outcomes=7)3. Harmonise data
调整暴露因素和结局变量的gwas结果,主要目的
将SNP位点统一调整成正链
根据allele和频率判断两个gwas结果中的SNP位点是否一致,不一致的进行去除
同一个位点在两个gwas结果中链的方向不一致的情况示意如下
exposure effect = 0.5
effect allele = A
other allele = G
outcome effect = -0.05
effect allele = C
other allele = T








 本文介绍了如何使用TwoSampleMR R包进行两样本孟德尔随机化(2SMR)分析。该包允许调用MR-Base数据库中的GWAS结果,进行暴露因素与结局变量的关联分析。分析过程包括读取暴露数据、结局数据,数据调和,以及运行MR分析。数据调和步骤确保SNP位点的一致性和方向性,而MR分析默认采用多种方法,如MR-Egger回归,支持异质性检验。虽然结局变量GWAS结果不支持自定义,但TwoSampleMR简化了2SMR流程,提高了分析准确性。
本文介绍了如何使用TwoSampleMR R包进行两样本孟德尔随机化(2SMR)分析。该包允许调用MR-Base数据库中的GWAS结果,进行暴露因素与结局变量的关联分析。分析过程包括读取暴露数据、结局数据,数据调和,以及运行MR分析。数据调和步骤确保SNP位点的一致性和方向性,而MR分析默认采用多种方法,如MR-Egger回归,支持异质性检验。虽然结局变量GWAS结果不支持自定义,但TwoSampleMR简化了2SMR流程,提高了分析准确性。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 1720
1720

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








