cafe5 版本 输入文件:1)基因家族在各个物种中的数目 2)超度量树 3)控制文件参数λ值
注!!!树的顺序、名字和基因文件的要对应起来啊!!!
cafe -i infile -t tree.file -o output_prefix -l fixed_lambda -P pvalue1.input文件使用之前做的orthofinder得到的Orthogroups.GeneCount.tsv
在这里~/Qxy/knowngenome/10proteins.orthofinder/OrthoFinder/Results_May29/Orthogroups
awk -v OFS="\t" '{$NF=null;print $1,$0}' Orthogroups.GeneCount.tsv |sed -E -e 's/Orthogroup/desc/' -e 's/_[^\t]+//g' >gene_families.txt
#可以用这个代码去把Orthogroups.GeneCount.tsv文件改为gene_families.txt文件,用作CAFE的输入文件之一#注!要剔除不同物种间的基因拷贝数变异特别大的基因家族。否则可能无法估计参数,运行中断报错Failed to initialize any reasonable values
awk 'NR==1 || $3<100 && $4<100 && $5<100 && $6<100 && $7<100 && $8<100 && $9<100 && $10<100 && $11<100 && $12<100 && $13<100 {print $0}' gene_families.txt >gene_families_filter.txt
#使用这个代码去除基因拷贝数变异特别大的基因家族,其实就是将每列>100的给pass掉长成这个样子!!!物种数要对,格式要对 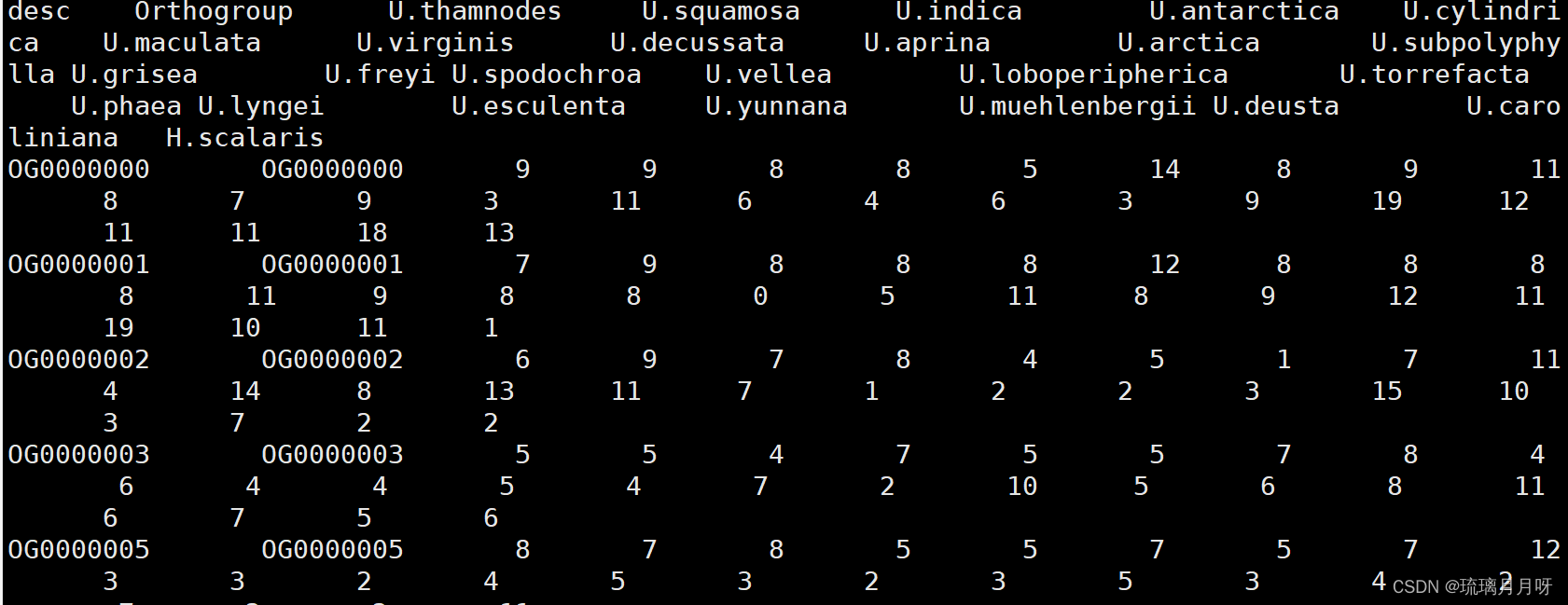
所以!!!如果是共线性比对的话,还需要手动编辑代码查找一下每组同源基因在每个物种中的数量。共线性比对结果中newgenes文件夹是以各个scaffold命名的文件,里面是每个基因的比对结果(但是最多也就是1个基因啊!!!)
2.树文件,一般都是自己写(删掉这一点,并入3里)
根据分歧时间的FigTree.tre文件改写 格式参照cafe说明书 需注意,树需要为超度量树
grep -E -v "NEXUS|BEGIN|END" FigTree.tre|sed -E -e "s/\[[^]]*\]//g" -e "s/[ \t]//g" -e "/^$/d" -e "s/UTREE/tree tree/" >tree.txt
#改代码是将FigTree.tre结果转为CAFE输入的tree.txt结果MCMCTree进行时间推断时设置的分化时间是以100个MYA为单位的,而在输入到CAFE中的时间分化树需要改为以MYA为单位。要扩大100倍!
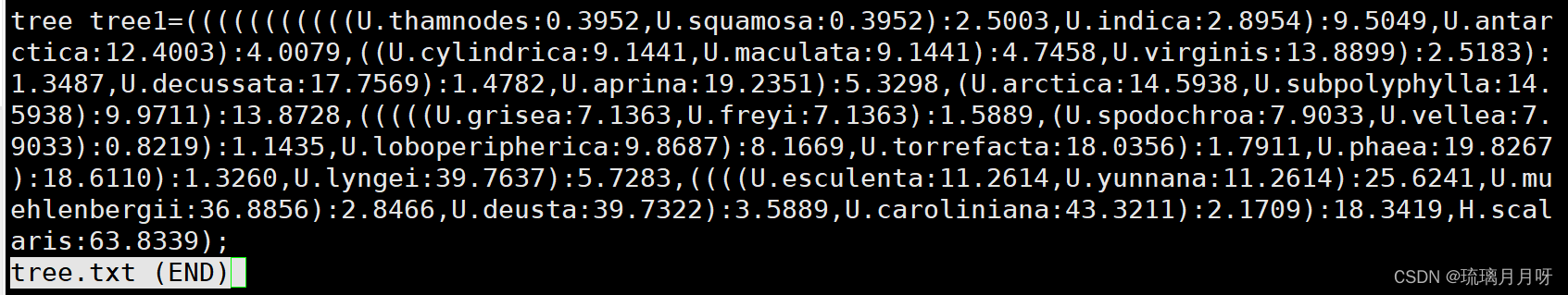
我记得当时是手动调整了一下
3. 控制文件
直接写入控制文件cafe_command2里,里面有基因家族统计文件,树文件直接粘过去,设置λ参数
#!/project/metagenome/zyr/CAFE/release/cafe
version
date
load -i gene_families_filter.txt -t 8 -p 0.01
tree (((((((((((U.thamnodes:0.3952,U.squamosa:0.3952):2.5003,U.indica:2.8954):9.5049,U.antarctica:12.4003):4.0079,((U.cylindrica:9.1441,U.maculata:9.1441):4.7458,U.virginis:13.8899):2.5183):1.3487,U.decussata:17.7569):1.4782,U.aprina:19.2351):5.3298,(U.arctica:14.5938,U.subpolyphylla:14.5938):9.9711):13.8728,(((((U.grisea:7.1363,U.freyi:7.1363):1.5889,(U.spodochroa:7.9033,U.vellea:7.9033):0.8219):1.1435,U.loboperipherica:9.8687):8.1669,U.torrefacta:18.0356):1.7911,U.phaea:19.8267):18.6110):1.3260,U.lyngei:39.7637):5.7283,((((U.esculenta:11.2614,U.yunnana:11.2614):25.6241,U.muehlenbergii:36.8856):2.8466,U.deusta:39.7322):3.5889,U.caroliniana:43.3211):2.1709):18.3419,H.scalaris:63.8339);
lambda -s -t (((((((((((1,1)1,1)1,1)1,((1,1)1,1)1)1,1)1,1)1,(1,1)1)1,(((((1,1)1,(1,1)1)1,1)1,1)1,1)1)1,1)1,((((1,1)1,1)1,1)1,1)1)1,1)
report outlambda -s 表示自动搜索λ值,但是会报一些warnning
WARNING:Calculated posterior probability for family OG0000001 = 0 Lambda : 0.01572235204309 & Score: -inf
意思是:计算出来基因家族 OG0000001 的后验概率为0,且找到的最佳参数λ值为0.01572235204309,同时评分为负无穷。
#Lambda是在模型中使用的缩放参数,用于校正生物数据的不同,以使其更好地适应模型。
#评分为负无穷,表示经过计算后得到的值小到接近于零,但又不完全等于零。实际上,对数后验概率等于负无穷,表明此家族产生的概率非常接近于零。
在运行完cafe5之后得到的.cafe文件中,会有lambda和lamtree两行,第一行是整个物种树规定的λ,第二个是各个节点的λ值,可以使用-l参数指定整个树的λ,也可以用-t 指定各个节点的λ
可以运行一次,查看一下最佳λ值是多少,直接指定,也可以修改限制XX节点是一样的λ,XX不一样
4.运行
./cafe_command
或者
python /ifs1/User/wuqi/SoftTools/CAFE/cafe/caferror.py -i cafe_command5.结果分析
~/Qxy/qxyjiaoben/parsing_cafeOut.pl out.cafe
#利用教材中的脚本去得到各节点扩张收缩基因家族信息#cafe.out结果文件解析
1)超度量时间分歧树内容
2)lambda一行显示的是λ值,基因出生率/死亡率
3)IDs of nodes 表示不同节点的编号,这里U.thamnodes是0,U.squamosa是2,U.thamnodes和U.squamosa所在节点是1,其他以此类推,直至47
# IDs of nodes:(((((((((((U.thamnodes<0>,U.squamosa<2>)<1>,U.indica<4>)<3>,U.antarctica<6>)<5>,((U.cylindrica<8>,U.maculata<10>)<9>,U.virginis<12>)<11>)<7>,U.decussata<14>)<13>,U.aprina<16>)<15>,(U.arctica<18>,U.subpolyphylla<20>)<19>)<17>,(((((U.grisea<22>,U.freyi<24>)<23>,(U.spodochroa<26>,U.vellea<28>)<27>)<25>,U.loboperipherica<30>)<29>,U.torrefacta<32>)<31>,U.phaea<34>)<33>)<21>,U.lyngei<36>)<35>,((((U.esculenta<38>,U.yunnana<40>)<39>,U.muehlenbergii<42>)<41>,U.deusta<44>)<43>,U.caroliniana<46>)<45>)<37>,H.scalaris<48>)<47>
Gamma_asr.tre ## 每个基因家族的树文件
Gamma_branch_probabilities.tab ## 每个分支计算的概率
Gamma_category_likelihoods.txt
Gamma_change.tab ## 每一个基因家族在每个节点的收缩与扩增数目
Gamma_clade_results.txt ##每个节点基因家族的扩增/收缩数目
Gamma_count.txt ## 每一个基因家族在每个节点的数目
Gamma_family_likelihoods.txt
Gamma_family_results.txt ## 基因家族变化的p值和是否显著的结果
Gamma_results.txt ## 模型,最终似然值,最终Lambda值等参数信息。
4)Average Expansion #平均每个基因家族中扩张的基因数目,负数表示基因家族收缩
5)Expansion #发生了扩张的基因家族数目
6)nRemain #没有发生改变的基因家族数目
7)nDecrease #发生了收缩的基因家族数目
8)每个基因家族具体扩张和收缩的情况
第一列对应输入基因家族的编号;第二列是Newick的进化树,U.thamnodes_9:0.3952中的9表示该基因家族在U.thamnodes中有9个基因;
第三列是Family-wide P-value,基因家族扩张收缩的pvalue,用于表明该基因家族是否是显著性的扩张或是收缩,p值小于0.01时,表示发生显著差异变化
第四列是每个分枝的基因家族扩张的显著性,表明哪个分支的基因家族发生了变化,有变化用数字XX具体表示,无变化的用-表示
9)使用脚本生成的文件
#cafe_out.out每个物种在每个基因家族上的收缩或扩张情况
#cafe_out.cafe 与out.cafe类似,但是更详细一些
#cafe_out.stats 每个节点的扩张和收缩情况
10)作图
python /ifs1/User/wuqi/SoftTools/CAFE5/docs/tutorial/cafe5_draw_tree.py -i cafe_out.stats -y Expansions -d out1.cafe -o clade_results1.png由于服务器上作图有点失败,只能显示收缩或者扩张,所以第一次是自己手动P的,后期也可以尝试一下R进行作图。
后期1020跑CAFE分析也失败了,手动总结了一下每个节点基因扩张、收缩数目,后期AI处理
安装了CAFE_fig在/ifs1/User/wuqi/Qxy/knowngenome/CAFE目录下,使用时运行代码:
python3 ../CAFE_fig/CAFE_fig.py out1020.cafe -pb 0.05 -pf 0.05 --dump test/ -g pdf --count_all_expansions会生成一个总的pdf文件和每个基因家族的pdf文件,传输到本地查看(但是我搞出来全是蓝色的,且树被压缩的很短不知道咋回事)
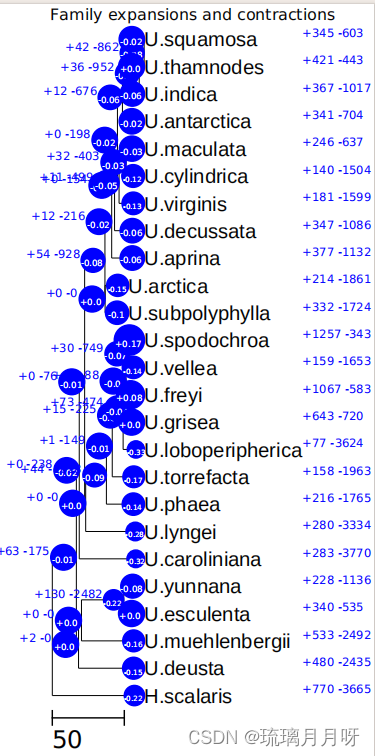
注意:CAFE_fig.py中对pixels_per_mya进行修正。源码中是1.0,如果想让长度变成5倍则更正为5.0即可
查到一个这个,可以试试改一下CAFE_fig.py中的代码,但是出bug了,跑不了了





















 2743
2743











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








