The gradual establishment of complex coumarin biosynthetic pathway in Apiaceae
伞形科中复杂香豆素生物合成途径的逐步建立

摘要:复杂香豆素(CCs)是伞形科植物中的特征性代谢产物,具有重要的药用价值。它们的重要功能可能是作为病原体防护剂并调节响应环境刺激。利用包括我们新近测序的前胡(Peucedanum praeruptorum)在内的34种伞形科植物的基因组和转录组,我们进行了全面的系统发育分析,以重建伞形科中CC生物合成途径的详细进化过程。我们的结果显示,三个关键酶——对香豆酰CoA 2'-羟化酶(C2’H)、C-异戊烯转移酶(C-PT)和环化酶——通过不同的基因重复方式(异位和串联重复)在伞形科的不同进化节点上依次起源。新功能化赋予这些酶在CC合成中所需的新功能,从而完成了该途径。候选基因被克隆用于异源表达,并通过体外酶活性检测来验证我们关于关键酶起源的假设,结果精确地验证了我们的进化推论。在这三种酶中,C-PTs可能是CC结构多样性的主要决定因素(线性/角形),因为它们进化出不同的活性以靶向伞形酮的不同位置(C-6或C-8)。一个关键的氨基酸变异(Ala161/Thr161)被识别并证明在改变酶活性中起着至关重要的作用,这可能导致酶与底物之间不同的结合形式,从而产生不同的产物。总之,本研究提供了伞形科中CC生物合成途径建立和进化的详细轨迹,解释了为什么只有部分而非所有伞形科植物能够产生CC,并揭示了不同伞形科植物中CC结构多样性的机制。
引言
为了应对与侵害者的军备竞赛,植物进化出生产次生代谢产物的能力。一些次生代谢产物在人类中表现出特定的治疗活性,并被我们的祖先巧妙地应用于治疗人类疾病,这些祖先发展了阿育吠陀医学、阿拉伯医学、传统中医药以及其他古老的治疗方法。香豆素是植物中主要的代谢产物类别之一。除了作为植物病原体的防护剂并响应生物和非生物压力外,香豆素还表现出多种医学活性,如抗菌、抗肿瘤、抗氧化、抗凝血和抗炎等特性。
这些多样化的生物活性可能源于香豆素的结构多样性。香豆素可分为五种类型:简单香豆素(SCs)、线型呋喃香豆素(FCs)、角型FCs、线型吡喃香豆素(PCs)和角型PCs;后四者统称为复杂香豆素(CCs)。相对而言,CCs是具有潜在药用价值的主要成分,是由SCs进一步生物合成的产物。生物合成过程涉及三种不同类型的酶——对香豆酰CoA 2'-羟化酶(C2’H)、C-异戊烯转移酶(C-PT)和环化酶——并通过三个独特的步骤完成。CCs的详细生物合成过程(如图1所示)始于C2’H催化对香豆酰CoA生成伞形酮。在此步骤中,C2’H与催化东莨菪素(F6’H)生物合成的酶竞争底物,而东莨菪素不会继续生成CCs。由于严格的底物特异性,只有伞形酮能被下游的C-PTs识别,并通过加戊烯基化继续生成去甲莨菪素或欧前胡素,而O-异戊烯转移酶(O-PTs)则将反应引向6',7'-环氧甲氧基香豆素或6',7'-二羟基佛手柑内酯,而不再继续生成CCs。最后,环化酶催化吡喃或呋喃环的环化,形成PCs或FCs,这些环化酶最近才被鉴定出来,填补了CCs生物合成途径的最后一块缺失环节。

PAL苯丙氨酸解氨酶,C4H肉桂酸4-羟化酶,4CL 4-香豆酸:辅酶A连接酶,HCT/HQT羟基肉桂酰CoA莽草酸/奎宁酸羟基肉桂酰转移酶,CCoAOMT咖啡酰-CoA O-甲基转移酶,C3’H肉桂酰酯3'-羟化酶,C2’H对香豆酰CoA 2'-羟化酶,F6’H阿魏酰-CoA 6'-羟化酶,COSY香豆素合酶,U6PT伞形酮6-异戊烯转移酶,U8PT伞形酮8-异戊烯转移酶,DC去甲莨菪素环化酶,OC欧前胡素环化酶。图片中的植物是新近测序的前胡(Peucedanum praeruptorum),其富含所有类型的香豆素,特别是高含量的角型复杂香豆素(CCs)。
在自然界中,尽管简单香豆素(SCs)在被子植物中普遍存在,主要的活性成分CCs据报道主要积累在433个被子植物科中的四个科:伞形科、桑科、芸香科和豆科,这四个科在系统发育上彼此相距较远。CCs在被子植物系统发育中的这种分散分布暗示了CCs生物合成在这四个科中具有多次独立的起源。巧合的是,分子证据也支持了CCs生物合成在不同被子植物谱系中的独立进化。例如,对异戊烯转移酶(PTs)的系统发育分析表明,伞形科、桑科和芸香科的PTs是通过趋同进化从不同的祖先中衍生出来的。负责环化的酶也分别属于不同的CYP450家族:桑科中的CYP76F和伞形科中的CYP736A。
尽管伞形科因其丰富的CC代谢物而闻名,但事实是并非所有伞形科植物都能产生CCs。一些著名的中草药,如前胡(Peucedanum praeruptorum)、当归、白芷、防风等在根部积累CCs。相反,其他植物,例如大多数蔬菜和香料——胡萝卜、芹菜、水芹和香菜——则不积累CCs。此外,一些其他著名的药用植物如积雪草和柴胡也不积累CCs。这些观察结果暗示CCs的生物合成在伞形科中可能经历了多变的进化模式。如今,随着测序技术的快速进展,伞形科已经积累了丰富的基因组和转录组数据资源。我们还新近测序了前胡,一种富含所有类型香豆素,特别是高含量角型CCs的伞形科植物。这些数据为阐明CCs生物合成的起源和多样化产物的分子机制提供了宝贵的基础信息。利用进化基因组学研究策略,我们的工作力图重建伞形科CCs生物合成途径建立和进化的详细过程。
结果
伞形科物种的系统发育提供了研究CC生物合成进化的框架
为了为我们对伞形科CC生物合成的分析提供一个稳定的物种关系框架,我们首先利用该科的所有可用基因组和转录组数据以及来自其他分类群的18种植物作为外类群(参见补充表1),重建了伞形科的高分辨率系统发育树。在使用的分类群中,我们新近测序的前胡(Peucedanum praeruptorum)使用了Hifi和HiC数据(大小为1.74 Gb,scaffold N50 = 157.14 Mb,注释了33,420个蛋白编码基因,我们的注释捕获了94.9%的陆地植物BUSCO (odb10)基因,其中82.6%为单拷贝基因,12.3%为重复基因,详细信息见补充图1-3、补充表2-9和补充注释),作为一个富含多样化CC产物的代表。
根据OrthoFinder (v2.5.5)分类的直系同源基因家族,我们提取了总共1708个低拷贝直系同源基因家族(OGs),并使用方法部分描述的标准生成了精细化的单拷贝基因(RSCGs)。然后处理RSCGs,并构建了氨基酸和相应编码序列的连接数据集。核苷酸和氨基酸数据集都恢复了伞形科系统发育的一致拓扑结构,并且具有强大的支持(见图2,补充图4)。在伞形科中,前胡被解析为防风的姐妹群,而蛇床、白芷和芹菜依次被恢复为姐妹群,均属于伞形亚科。除芹菜与欧防风的姐妹关系外,所有关系均获得了100%的bootstrap支持,而该姐妹关系在氨基酸数据集中获得了98%的支持。因此,进一步使用基因共alescent数据集进行了分析,获得了一致的结果,支持其姐妹关系,而不完全的谱系排序(见图2,补充图5)可能影响了bootstrap值。伞形亚科是伞形科所有亚科中采样最密集的,因为据报道,伞形科植物中含有CC代谢物的植物主要属于这一分支。所有13种伞形亚科植物聚集成一个单系群,直刺变豆菜、Azorella atacamensis和积雪草依次代表Saniculoideae、Azorelloideae和Mackinlayoideae。分子钟估计伞形科冠群的出现大约在4900万年前(MYA),而伞形科与其他伞形目谱系的分化可以追溯到5300万年前。前胡与其最近亲属防风的分化时间约为550万年前(见补充图6,补充表10),这可能代表了前胡属和防风属的分化时间。

系统发育分析和分歧时间估计基于来自34种被子植物的1708个精细化低拷贝直系同源基因组。香豆素的类型和分布信息来源于多篇文献。‘-’表示无报道,SCs表示简单香豆素,LFs表示线型呋喃香豆素,LPs表示线型吡喃香豆素,AFs表示角型呋喃香豆素,APs表示角型吡喃香豆素,WGD表示全基因组复制。相关分析请见补充图26–28。星号表示100%支持。源数据已作为源数据文件提供。
通过将我们收集的伞形科植物中香豆素积累的信息映射到系统发育主干上,值得注意的是,伞形科中积累CC的植物仅限于集中在伞形亚科的一些族或属中,而不是均匀分布在整个伞形科中(见图2)。因此,完整的CC生物合成途径很可能首先在伞形亚科的后期进化中建立,这可以解释为什么其他伞形科亚科缺乏CCs。同时,不同的植物倾向于积累不同的CCs,大多数伞形亚科植物无法生产所有类型的CCs,除了前胡,这表明伞形科CC生物合成途径可能处于一种活跃的进化模式。
伞形科C2'H通过异位重复在伞形亚科早期进化中起源
鉴于被子植物中上游苯丙氨酸代谢途径的保守性,共享性,下游的C2'H、C-PT和环化酶对于探索CC生物合成在伞形科中建立和进化的机制至关重要。CC生物合成由C2'H靶向对香豆酰CoA启动,关键的触发酶C2'H由2-氧代戊二酸依赖双加氧酶(2-OGD)基因家族编码。为探讨伞形科C2'H的起源和进化,我们使用从154种植物基因组和17种转录组(涵盖42个被子植物目)中提取的所有2-OGDs构建了一个系统发育树。选择的基因组和转录组旨在提供充足且平衡的物种采样,重点关注伞形科,作为系统发育分析的数据来源(参见补充表11、12)。系统发育树可以分为三个单系群,可能是由于早期被子植物祖先中的重复事件。伞形科C2'H位于亚群I(见图3a),而其他两个亚群分别包含拟南芥的东莨菪素8-羟化酶(S8H)和大豆的阿魏酰-CoA 6'-羟化酶(F6'H)。通过将基因树与物种树进行比较,并检查伞形科C2'H在亚群I中的位置,可以识别出伞形科特有的两轮重复,产生了三个伞形科特有的分支,之前已被鉴定的C2'H(欧防风和前胡的C2'H)嵌套在分支A中(见图3b)。可以观察到,三个分支的成员仅限于伞形亚科,且包括所有伞形亚科物种,因此我们推测产生C2'H的重复发生在伞形亚科早期进化过程中,这得到了伞形科基因重复事件调和分析的支持(见补充图7)。我们的功能表征显示,通过在大肠杆菌中异源表达,分支C中的Pp2.7733也表现出C2'H的酶活性(见图3c),表明C2'H的功能可能通过早期的重复事件起源。由于在两个重复事件中产生的基因未观察到在染色体上成簇或显示共线性(见补充图8),伞形科C2'H可能通过异位重复起源并扩展。这两个异位重复可能追溯到伞形亚科的早期祖先。由于后来的串联重复事件,前胡在分支A中有四个拷贝,位于第9染色体上,这些重复基因(Pp9.2445、Pp9.2502、Pp9.2515和Pp9.2536)都表现出C2'H活性,尽管在定量活性上存在一些差异(见补充图11)。然而,分支B中的Pp6.1129未表现出酶活性(见图3c和3d)。在检查Pp6.1129的序列时,我们发现该蛋白的N末端缺失了十个氨基酸/有变异(见补充图12),我们推测这可能导致了功能丧失的突变。由于测试的蛋白质(除了Pp6.1129)还表现出F6'H活性(见图3d),而F6'H在其他物种和亚群中具有更广泛的系统分布,我们推测F6'H应该是原始功能,而伞形科C2'H可能由F6'H衍生而来。这个假设也得到了C2'H在系统发育树上单系群的支持,表明C2'H是一种后期进化出的活性,相比之下F6'Hs分布更广泛。在序列水平上,我们通过多序列比对发现,C2'H具有更多保守的氨基酸残基,而F6'Hs则显示出更多样化的残基类型(见补充图13、补充数据1)。

a 从154个植物基因组和17个转录组中分析了2-OGD基因家族的系统发育,详细的系统发育信息见补充图29。GenBank登录号分别为:PsC2'H, APP94171.1; PpC2'H, ASR80916.1; AtS8H, NP_187896.1 (AT3G12900.1); GmF6'H, AHY03267.1; AtF6'H1, NP_187970.1 (AT3G13610.1); AtF6'H2, NP_175925.1 (AT1G55290.1)。b 伞形科家族F6'H和C2'H基因的系统发育树提取图。绿色圆点表示C2'H功能的起源。红色和紫色圆点分别表示相应基因具有C2'H和F6'H功能。橙色五角星表示已发表的功能性基因。c 选定的伞形科家族2-OGD基因的C2'H活性测定。d 选定的伞形科家族2-OGD基因的F6'H活性测定。图中展示了标准品的液相色谱图及其相应的化学结构。伞形花内酯和木犀草素的保留时间分别为20.75和19.49分钟。源数据作为源数据文件提供。
串联重复导致了伞形科家族C-PTs的产生及U6-和U8PT的活性分化。
虽然C-PT在生物合成路径中作用于C2'H之后,但它首次通过伞形花内酯C-6和C-8位的异戊二烯化作用生成了去甲基胡椒酚和阿米朵素,从而产生了产品的多样性(图1)。这两种产物分别是线型和角型香豆素的前体,使C-PT成为阐明线型和角型香豆素产物配置机制的关键。利用相同的154个基因组和17个额外的转录组资源(补充表11、12)对C-PT进行了进化分析。系统发育分析表明,C-PT基因可能经历了复杂的进化过程。尽管C-PT是一个小的基因家族(每个被子植物物种平均有十几个成员),但推测具有C-C活性的PTs的进化历程涉及多达七次伞形科特有的重复事件(图4a、b),其中串联重复应是主要机制。例如,前胡的十个PT基因(PpPTs)中有七个聚集在一个单系群中,表明它们的进化关系密切,而且它们都位于第9号染色体上的同一位点,形成了一个基因簇(补充图8)。这些结果为PpPTs的新兴机制提供了有力证据:通过串联重复生成。三个已鉴定的前胡 C-PTs(PpPT1-3)7也位于该基因簇中,表明伞形科家族的C-PT酶可能来源于串联重复事件。具体来说,这些串联重复事件不应仅限于前胡,因为七个簇状PpPTs分布在五个系统发育分支上,其中包括来自多个植物的直系同源基因。通过与物种树(图2)比较,可以看到已鉴定的C-PTs(PpPT1-3)及其直系同源基因仅存在于Selineae(前胡、A. dahurica、C. monnieri和S. divaricata)、Tordylieae(P. sativa)、Coriandreae(C. sativum)、Sinodielsia(A. sinensis)和Apieae(A. graveolens)(图4b),这些部落/谱系起源于早期Apioideae进化中的一个共同祖先(图2)29,30。因此,分支A、B和C的产生的串联重复事件应该发生在这些部落的共同祖先中(补充图14),因此这些分支中的基因通过新功能化进化出了C-PT活性。来自三个不同分支的基因之间的氨基酸序列同一性较高(至少超过75%),A和B之间的中位数为84.43%,A和C之间的中位数为82.35%,B和C之间的中位数为81.56%(补充图15),这表明这些来自不同分支的基因关系密切,也暗示它们的功能分化可能仅由少量序列变异引起。

a 从154个植物基因组中分析了PT基因家族的系统发育,详细的系统发育信息见补充图25。 b 提取的伞形科(伞形科)PTs系统发育树。 c 通过高效液相色谱法(HPLC)对C-PTs进行酶活性测定。 d 通过高效液相色谱法(HPLC)对O-PTs进行酶活性测定。 e 伞形科中U6PTs和U8PTs之间的六个显著氨基酸差异位点。 f 通过高效液相色谱法(HPLC)对具有特定位点突变的PpPT1和PpPT2进行酶活性测定。DMAPP代表异戊二烯焦磷酸,DMS代表去甲基胡椒酚。 g PpPT1和PpPT2的三维蛋白质结构,以及U6PT和U8PT催化伞形花内酯异戊二烯化过程的假设。关键位点(Thr161/Ala161)以颜色显示。源数据作为源数据文件提供。
为了验证我们的假设,随后进行了功能性实验。选择了来自不同分支的基因在大肠杆菌中异源表达,并通过高效液相色谱法(HPLC)和质谱法(MS)分析其产物。通过将我们的实验结果(图4c、d,补充图16、17)与之前表征的酶映射到PT系统发育树(图4b),可以观察到只有在A、B和C分支中的基因(仅存在于Selineae、Tordylieae、Coriandreae、Sinodielsia和Apieae中)在伞形花内酯C-6(U6PT,以绿色点表示)或C-8(U8PT,以粉红色点表示)处具有C-C键活性,而其他分支中的基因被测试为具有O-PT活性而非C-PT活性(以蓝色点表示)。这些实验结果与我们的进化推断一致,即C-PTs在后期分化的Apioideae谱系中起源较晚。此外,证明了C-PT活性是通过新功能化从O-PT基因进化而来的。毕竟,产生A、B和C分支的重复事件发生相对较晚(补充图14)。
代表U6PT和U8PT的分支在系统发育关系上非常接近。在前胡中,U6PT(PpPT1)和U8PT(PpPT2)位于同一个基因簇中(补充图8),表明它们是串联重复的产物,并经历了新功能化或亚功能化。对A和C分支中基因编码的蛋白质序列进行比较,揭示了六个位点(Leu102-Phe102、Thr161-Ala161、Ile195-Val195、Phe262-Tyr262、Gly335-Ala335和Lys388-Gln388,以PpPT1为参考)具有显著的序列差异(图4e,补充图18),可能解释了U6PT和U8PT的功能分化。为了确定这些位点的关键作用,我们通过特定位点突变改变了氨基酸,以测试相应的功能变化。突变的PpPT1s和PpPT2的酶活性测定表明,位于161位的氨基酸是从U6PT活性向U8PT活性转变的关键,Thr161决定了U6PT活性,而Ala161决定了U8PT活性,因为具有Thr161-to-Ala161(T161A)突变的PpPT1显示出U8PT活性,而具有Ala161-to-Thr161(A161T)突变的PpPT2显示出U6PT活性,而其他突变没有显示出明显的活性变化(图4f,补充图19)。通过Alphafold231建模了PpPT1和PpPT2的三维蛋白质结构,并通过分子对接推测它们与伞形花内酯和异戊二烯焦磷酸(DMAPP)形成复合物。我们推断,PpPT1由于其亲水性Thr161,可能倾向于与伞形花内酯的亲水侧结合,使DMAPP只能与C-6结合,生成线型产物。而PpPT2由于其疏水性Ala161,更倾向于与伞形花内酯的疏水侧结合,因此DMAPP只能靶向C-8,从而形成角型构象(图4g)。
伞形科环化酶通过异位重复起源
负责伞形科中CC生物合成最后一步的环化酶属于CYP450超家族。为了探索伞形科环化酶的起源和进化,我们从18个植物基因组和17个转录组中提取了所有CYP450蛋白质(使用较少的基因组是因为植物基因组中的CYP450基因数量过多,补充表12、13),并进行了系统发育分析(图5a)。根据功能,伞形科中的环化酶可以分为两种类型,即去甲基胡椒酚环化酶(DC)和阿米朵素环化酶(OC),分别催化线型和角型产物7,11。在前胡中,最近表征的PpDC(分支A中的Pp7.4765)和PpOC(分支C中的Pp9.2436-37)具有非常相似的序列7,并且在系统发育树上与另外两个CYP450基因(分支B中的Pp4.4512和Pp4.4519)被归类为近亲(图5b)。虽然Pp4.4512和Pp4.4519被测试为具有极弱的环化酶活性7,但同一分支的S. divaricate基因(DN7479)被证明具有更强的DC活性(图5c,补充图20、21),这表明这两个前胡基因可能存在功能丧失的情况。由于A和B分支的基因均表现出DC活性(图5c,补充图20),DC的起源可以追溯到这两个分支的共同祖先。这个祖先作为OC谱系(分支C)的姐妹群,表明DC和OC谱系同时分化。先前的研究报告指出,来自其他分支(A、B和C分支之外)的基因没有环化酶活性7。因此,环化酶活性仅限于A、B和C分支,包括仅来自Selineae、Apieae、Sinodielsia和Tordylieae的基因,环化酶因此应该起源于这些谱系的共同祖先。相对而言,伞形科环化酶起源于C2’H之后,并且也不早于C-PTs,最终建立了完整的CC生物合成路径。在同时拥有DC和OC的伞形科基因组(如前胡和A. sinensis)中,DC和OC基因之间没有检测到基因组内同线性,这表明伞形科 DC和OC可能来源于异位重复(补充图22)。此外,推测独立的基因丧失事件发生频繁,因为所有三个分支都被推测经历了基因丧失事件,其中OC分支被推测经历了最多的基因丧失(补充图23)。
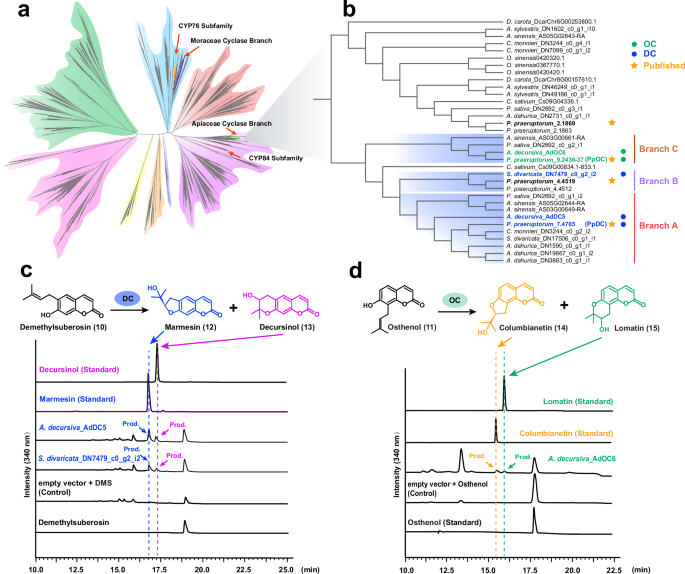
a 从18个植物基因组和17个植物转录组中分析了CYP450基因家族的系统发育,详细的系统发育信息见补充图30。 b 提取的伞形科(伞形科)环化酶的系统发育树。 c 通过高效液相色谱法(HPLC)对DC(去甲基胡椒酚环化酶)进行酶活性测定。在16.7分钟和17.2分钟处的峰分别为marmesin和decursinol。 d 通过高效液相色谱法(HPLC)对OC(阿米朵素环化酶)进行酶活性测定。在15.4分钟和16.0分钟处的峰分别为columbianetin和lomatin。源数据作为源数据文件提供。
CC生物合成路径的建立和崩溃与编码酶的基因起源和丧失相关
总之,伞形科的C2'H、C-PT和环化酶是伞形科内特定重复事件的结果(图3b、4b和5b),这表明这些酶基因的严格直系同源体仅存在于伞形科中。更具体地说,它们是通过不同的重复事件(串联重复和异位重复)在Apioideae的不同系统发育节点上产生的。对基因重复事件的全面整合有助于追溯这三个关键酶基因的详细进化历史(补充图7、14、23),展示了伞形科 CC生物合成工具箱的逐步完成(图6)。尽管C2'H在Apioideae中起源较早,其他两种酶随后相继出现。直到Daucinae-Scandicinae谱系分化时,环化酶才最终完成CC生物合成的最后一步。理论上,所有Apieae、Sinodielsia、Tordylieae、Coriandreae和Selineae植物都应该拥有完整的CC生物合成工具箱,并能够产生CCs。然而,二次丧失应该已经发生(图6,补充图7、14、23),导致某些酶的缺失,可能影响CC产物的多样性,甚至导致生物合成路径的崩溃;例如,芹菜和香菜失去了所有的环化酶,因此完全无法产生CCs。这个重建的历史解释了当前伞形科植物中CC的存在或缺失,并与这些物种中已知的代谢产物一致,反映了基因型与表型的一致性。

伞形科C2’H至少可以追溯到伞形科亚科的共同祖先,而伞形科的C-PTs和环化酶可能在Daucinae-Scandicinae谱系分化后不久起源。之后,这些关键酶的二次丧失现象很常见,尤其是对负责角型CCs(香豆素)的U8PTs和OCs的丧失更为普遍。H1表示C2’H;U6表示U6PT;U8表示U8PT。
一个可能解释前胡中高含量角型CCs的CC基因簇
虽然许多伞形科亚科植物具有完整的CC生物合成工具箱,但其产物的含量和类型差异显著,表明不同类群之间生物合成效率存在差异。相比之下,前胡进化出了比其他伞形科植物更强的生物合成能力,主要积累角型产物。生物合成基因簇(BGCs)是一种涉及各种代谢途径的基因的特定而常见的组织形式,例如,苯并恶嗪类、烯桃素、菟丝子素、多炔保护素,涵盖了植物、真菌和细菌等多种生物。BGCs的重要性被推测与生物合成的效率有关:上游和下游酶之间的紧密物理距离可能在转录期间保持同步,并有助于途径中中间体的快速处理。我们检查了前胡中CC途径基因的基因组组织。通过将所有PpC2’Hs、PpPTs、PpDC和PpOC锚定到染色体上(图7,补充图8),我们观察到所有四个PpC2’Hs、七个PpPTs(包括PpPT1-3)和PpOC都位于第9号染色体上的同一位点,显示出彼此之间非常近的物理距离,形成了一个典型的BGC;而PpDC位于另一条染色体(第7号染色体)上,与PpC2’Hs和PpPTs相距较远(补充图8)。因此,我们推测,这种聚集的基因排列应有助于角型CC生物合成的相应代谢过程,从而增强了前胡中角型CCs的生物合成效率。根据我们前述的进化推断,这个BGC通过多轮PpC2’Hs和PpPTs的串联重复及PpOC的异位重复插入产生。基因组间同线性分析在A. sinensis的第3号染色体上检测到一个类似的BGC,其中包含相同的三个酶基因(图7)。而其他伞形科物种,如A. graveolens、C. sativum、D. carota和O. sinensis,要么有不完整的BGC,要么完全缺乏BGC(补充图24),这与这些类群中仅检测到少量CCs或SCs(苄胺类)的事实相符。

PpC2’Hs、PpPTs和PpOC位于前胡基因组第9号染色体的一个基因簇中,而PpDC位于第7号染色体,不属于该基因簇。根据基因系统发育树,可以识别出涉及PpC2’Hs和PpPTs的多轮串联重复。A. sinensis具有与前胡在同线性基因组区块中包含C2’H、C-PT和环化酶的类似基因簇。
讨论
解密伞形科CC生物合成路径的进化
CCs是伞形科的特征代谢物,具有很高的药用价值和广阔的临床潜力。最近,三种关键酶(C2’H、C-PT和环化酶)被分离出来,从而揭示了伞形科中完整的CC生物合成路径。然而,值得注意的是,并非所有伞形科类群都能检测到CCs的积累,不同物种通常还会积累不同的CCs产物。为了理解伞形科中CCs的有无及多样性的分子基础,我们对所有可用的伞形科植物基因组和转录组进行了全面的进化研究,其中包括我们新组装的前胡,这是一种伞形科植物,含有所有类型的香豆素,并且角型CCs含量较高。基于系统发育分析,我们重建了三种关键酶基因(C2’Hs、C-PTs和环化酶)的进化历史。然后,结合功能性实验,我们揭示了这些酶的后起源和二次丧失应该可以解释伞形科内含有CCs的类群分布有限以及CCs产物多样性的原因。
这三种酶都是CCs生物合成不可或缺的,其中C-PTs预计对产物多样性做出了优先贡献,因为通过U6PT和U8PT的亚功能化/新功能化活动,分别生成了线型和角型前体。已表征的161位点(Ala/Thr)被证明对U6PT和U8PT之间的酶特性具有重要作用。同样,PpDC中的Glu303和PpOC中的Asp301定义了它们各自的催化功能,分别继续进行U6PT和U8PT下游的反应。因此,C-PT序列的多样性应该是产物多样性的决定性因素。然而,我们的实验只测试了单一氨基酸的变异,尚未扩展到不同候选位点/氨基酸的组合,因此同时在多个位点发生的某些突变也可能改变这些酶的活性。
先前的研究还提出了一个假设,即角型CCs是由线型CCs衍生的后期进化代谢物,因为角型CCs总是与线型产物共存。然而,支持这一假设的证据是间接的。根据我们的进化分析,角型CCs的出现不应晚于线型CCs,因为环化酶系统发育树表明,伞形科的DC和OC是同时出现的(在系统发育上互为姐妹群),并且它们的外群基因没有表现出DC或OC活性(没有先前进化的DC或OC功能)。
总之,我们的研究重建了伞形科CC生物合成的详细进化轨迹。这一重建增强了我们对伞形科植物中香豆素多样性背后分子机制的理解,并应激发在其他植物谱系中识别与CC生物合成相关的关键基因。
被子植物中复杂香豆素生物合成路径的平行进化
除了伞形科外,CCs还在三个系统发育上相距较远的被子植物谱系中积累,即芸香科、桑科和豆科。然而,到目前为止,伞形科是唯一一个已解码完整生物合成路径的谱系。由于该路径是由伞形科特有的基因重复事件衍生的,我们推测其他三个植物谱系中的三种酶基因不可能是伞形科基因的直系同源基因,最多可能是旁系同源基因。C2’H、PT和环化酶基因的系统发育提供了我们假设的进化证据。例如,被子植物PTs分化为三个亚家族,而大多数物种在亚家族I和III中保留了PTs,而在亚家族II中丧失了它们的成员。然而,伞形科和芸香科在亚家族I和III中仅保留了少量基因拷贝,但表现出亚家族II基因的显著扩展,这应该为伞形科和芸香科PTs的基因新功能化提供了丰富的材料。因此,伞形科亚家族II基因开发了包括U6和U8处的C-C键以及C-O键在内的多种活性;并且发现芸香科也在亚家族II中开发了具有C-O活性的基因。因此,可以合理推测,芸香科的C-PT基因也可能位于其大幅扩展的亚家族II成员中,因为在其他两个亚家族中,芸香科没有太多可用的选择。与伞形科和芸香科相比,其他两个类群——豆科和桑科在亚家族II中完全丧失。因此,豆科和桑科的C-PTs不可能来源于亚家族II,而必须位于亚家族I或III中,而这两个类群在这些亚家族中恰好扩展。事实上,大豆在亚家族I中有15个基因(包括功能性表征的GmDTs),而榕树在亚家族III中有38个基因(包括FcPTs),完美支持了我们的推论。总之,伞形科、豆科、芸香科和桑科必须通过不同PT亚家族的谱系特异性重复事件开发出功能性C-PTs,这表明C-PT活性在被子植物中的平行进化。
同样,环化酶基因也应该在不同被子植物谱系中平行出现。伞形科环化酶基因属于CYP84的姐妹亚家族。除了伞形科环化酶外,在其他植物谱系(桑科)中仅鉴定出一种额外的环化酶(F. carica中的CYP76F112),并且它属于不同的系统发育分支,与伞形科环化酶有较远的关系。基因系统发育的这种分散分布表明,伞形科和桑科的环化酶可能在平行进化中独立起源。
综上所述,谱系独立的基因重复事件可能为新功能化提供了材料。长期的进化选择可能引导了功能的趋同,最终导致在相距较远的被子植物类群中CC生物合成的反复起源。
方法
植物材料采集、基因组测序、组装与注释
本研究中使用的前胡样品采集自中国药科大学药用植物园(北纬31°54’,东经118°54’)。植物叶片首先用75%酒精清洗,然后用纯水清洗以进行DNA提取。基因组DNA采用十六烷基三甲基溴化铵(CTAB)法提取,并使用PacBio Sequel平台上的单分子实时(SMRT)DNA文库进行DNA测序。为了构建HiC DNA文库,DNA样品经过超声破碎、末端修复、加A尾、接头添加、纯化、PCR扩增,然后在Illumina Hiseq平台上进行双端读长测序(每端150 bp)。最终,共获得了188.37 Gb的PacBio数据和178.09 Gb的HiC数据,用于后续基因组组装(补充表3)。用于转录组测序的RNA从前胡的叶片和根部提取。
首先,使用Falcon (v3.1.0)中的自校正程序42校正原始PacBio数据,校正后的读数随后由Smartdenovo (v1.0.0)43处理以组装基因组。接着,我们使用200 kb作为窗口计算了组装后的基因组序列的GC含量和平均深度,以评估测序数据中是否存在GC偏差以及样品中是否存在污染。然后,基于Illumina测序读数,使用Pilon (v1.20)44进行三轮抛光,以修正由SNP、插入缺失和间隙引起的组装错误。使用Purge haplotigs (v1.1.0)45精炼组装并折叠同源区域,参数设置为:“-l 10 -m 63 -h 195”用于“purge_haplotigs contigcov”(“purge_haplotigs hist”和“purge_haplotigs purge”按默认参数执行)。为了获得染色体水平的基因组,约178 Gb的HiC数据用于结合ALLHiC (v0.9.8)46执行HiC染色体构象。在进行修剪、分区、救援、优化和构建五个步骤后,生成的支架水平基因组通过3D-DNA (v170123)47和Juicer (v1.6)48转换为染色质接触矩阵,并通过Juicebox (v1.11.08)49进一步可视化。经过手动调整,包括修正倒位错误和重新连接contig,选择了11个最长的支架,总长度为1.735 Gb(占总基因组组装的99.68%),推测对应于前胡单倍体基因组的11条染色体。最后,使用BUSCO (v1.0.0)50和LAI (v2.9.8)51评估组装的完整性和连续性。
我们采用了一种结合同源预测、从头预测和基于转录组预测的方法进行基因和功能预测。在同源预测过程中,我们将来自五个已发表植物基因组(拟南芥、甜橙、大豆、芫荽和胡萝卜)的蛋白质序列与前胡基因组进行了比对。在基于RNA-seq的预测过程中,为了优化基因组注释,使用Hisat2 (v2.0.4)52将各种组织的转录组数据比对到组装后的基因组上,以识别外显子区域和剪接位点。最后,使用EvidenceModeler (v1.1.1)53基于上述三种方法生成了共识基因集。
系统发育重建与分歧时间估算
在伞形科的系统发育重建过程中,我们采用了结合三种不同方法的策略。对于基于氨基酸序列的系统发育重建,我们收集了包括新生成的前胡基因组在内的17个基因组。此外,我们使用Trinity (v2.9.1)54从公共数据库中获取的转录组测序读数组装了17个转录组,随后通过Transdecoder (https://github.com/TransDecoder/TransDecoder)进行注释,将组装结果与公共蛋白质数据库进行比对。最终,使用cd-hit (v4.8.1)55获得了非冗余的最长转录本。然后,将这些氨基酸序列输入OrthoFinder (v2.5.5)26中,使用默认参数识别低拷贝直系同源家族(OGs)。为了生成精细的单拷贝基因(RSCGs),我们首先过滤出在单个物种中拷贝数少于三个的低拷贝直系同源家族。随后,我们进行了额外筛选,以确保每个低拷贝基因家族中至少90%的物种有超过一个基因拷贝。经过两轮过滤后,使用Python脚本对每个低拷贝基因家族进行去冗余处理,确保每个家族中的直系同源基因是每个物种中最长的拷贝。分别构建了多物种串联核苷酸和氨基酸数据集。最后,使用iqtree (v2.2.2.3)56,57,58基于这些串联数据推导最大似然树。在基于基因树推导物种树的过程中,使用iqtree (v2.2.2.3)57和Astral (v5.7.1)59分别推导最大似然树,并以默认设置总结共祖物种树和四分体支持(-t 2)。
使用PAML (v4.10.0)60,61,62中的MCMCtree软件包估算伞形科植物的分歧时间。选择了17个化石(补充表15)校准伞形科植物的时间树。为确保采样的合理性,我们将burn-in、sampfreq和nsample的值分别设置为400,000、10和100,000。
酶基因家族的系统发育分析
为了探索被子植物中PT和C2’H基因的进化轨迹,我们收集了154个植物基因组和17个伞形目植物转录组。随后,我们使用Orthofinder (v2.5.5)26识别同源基因。去除无效序列后,使用iqtree (v2.2.2.3)57重建基因树。对于环化酶基因,我们选择了来自五个科的18个基因组和17个转录组(补充表12, 13)。通过Blastp (v2.13.0)63和Hmmsearch (v3.3.2)64使用基于同源性的搜索方法识别同源基因。为了进一步追踪伞形科中这三个基因的重复事件,我们从功能验证的基因所在的系统发育拓扑结构中提取了单系。随后,我们基于转录本确保序列的准确性和完整性,并将其对齐后输入iqtree (v2.2.2.3)57。
总RNA提取与cDNA获取
使用EASYspin Universal Plant RNA Kit(Aidlab,北京,中国)从根部和叶片中提取总RNA。提取的RNA随后进行琼脂糖凝胶电泳以检查污染情况。随后,使用紫外分光光度计测定其纯度和浓度。提取的RNA立即使用TransScript All-in-One First-Strand cDNA Synthesis SuperMix for PCR(TransGen Biotech,北京,中国)反转录为cDNA。生成的cDNA存储在-20°C下以备后续实验使用。
重组PpC2’Hs的表达与纯化
使用PrimeSTAR® Max(Takara Bio Inc., 日本草津)和特定引物(补充表16),以cDNA为模板扩增四个PpC2’H基因的开放阅读框(ORF)。随后,基因用NdeI和EcoRI酶切,并连接到pET28a质粒中生成pET28a-C2’H,从而允许表达带有N端组氨酸标签的融合蛋白。重组质粒随后转化到E. coli BL21 (DE3)中。阳性转化子在Luria-Bertani培养基中于37°C和220 rpm下培养,直到OD600达到0.6-0.8。通过添加0.5
重组PpC2’Hs的表达与纯化
使用PrimeSTAR® Max(Takara Bio Inc., Kusatsu, Japan)和特定引物(补充表16)以cDNA为模板扩增四个PpC2’H基因的开放阅读框(ORF)。随后,这些基因经NdeI和EcoRI消化后连接到pET28a质粒中生成pET28a-C2’H,从而允许N端带有组氨酸标签的融合蛋白表达。重组质粒随后转化至E. coli BL21 (DE3)中。阳性转化子在37°C和220 rpm的Luria-Bertani培养基中培养,直到OD600达到0.6–0.8。培养物用0.5 mM异丙基-β-d-1-硫代半乳糖苷(IPTG)诱导12小时,在16°C下培养,然后以4000 rpm在4°C下离心15分钟。将细胞重悬于缓冲液(20 mM HEPES, 20 mM咪唑, 500 mM NaCl, 10%甘油, pH 7.5)中,并在冰上超声裂解15分钟。随后,在4°C下以12000 rpm离心60分钟以收集上清液以进行进一步纯化。使用Ni-NTA柱(Smart-Lifesciences, Changzhou, China)纯化重组蛋白,吸附的蛋白通过洗脱缓冲液(20 mM HEPES, 300 mM咪唑, 500 mM NaCl, 10%甘油, pH 7.5)洗脱。通过SDS-PAGE分析纯化的蛋白,并使用微量分光光度计(KAIAO, 北京)测定蛋白浓度。最后,将纯化的蛋白储存于−80°C以供未来研究。在酶反应中(200 μL),将20 μg纯化的蛋白与100 mM Tris HCl(pH 7.0)、0.5 mM FeSO4、2 mM抗坏血酸钠、2 mM 2-氧代戊二酸和200 μM底物(对香豆酸、对香豆酰CoA、阿魏酸和阿魏酰CoA)混合。反应混合物在20°C和300 rpm下孵育30分钟,然后通过加入200 μL甲醇终止反应。使用HPLC和MS检测反应产物。
PTs的克隆与功能验证
候选PTs在大肠杆菌中的异源表达按照先前的报告进行。使用特定引物(补充表16)扩增PpPTs的开放阅读框。PCR产物通过无缝克隆和组装试剂盒(TransGen Biotech, 北京, 中国)克隆到pETDuet-1载体的BamHI和EcoRI限制位点中。重组质粒随后转化至E. coli DH5α菌株,并对具有阳性测序结果的单克隆进行进一步培养以提取质粒。所有重组质粒分别插入大肠杆菌感受态细胞,并在37°C和220 rpm的Luria-Bertani培养基中培养,直到OD600达到0.6–0.8。随后用0.4 mM IPTG诱导并在25°C和120 rpm下孵育12-16小时。收获细菌细胞,并使用重组蛋白进行相关的活性验证实验。生物信息学研究表明,PTs是膜蛋白,这使得异源表达的AdPT蛋白难以纯化。因此,使用粗蛋白分析其活性。酶反应在220 rpm和25°C下的摇床中进行5小时。反应液由100 μL粗酶、200 μM伞形花内酯、200 μM MgCl2和100 μM DMAPP在200 μL的50 mM Tris-HCl(pH 8.0)中组成。上清液在15000 rpm下离心10分钟,并将10 μL的上清液通过HPLC和MS进行分析。
CYP450s的克隆与功能验证
CYP450s在酵母中的异源表达和体外测定按照我们之前的报告进行。使用特定引物(补充表16)扩增CYP450s的开放阅读框。随后,将基因克隆到酵母表达载体pYES2.0的BamHI/EcoRI位点中。重组质粒随后转化至酿酒酵母WAT11菌株中。在含20 g/L葡萄糖的固体SC-U(不含尿嘧啶的SC缺失培养基)上筛选阳性转化子,然后在液体SC-U培养基中培养,直到OD600达到2-3。随后,将细胞以4000 rpm离心10分钟,并至少用ddH2O清洗三次以去除葡萄糖残留。将细胞沉淀转移至含20 g/L半乳糖的SC-U培养基中以诱导目标蛋白的表达。为了初步活性筛选,将osthenol和去甲基胡椒酚以100 µM的最终浓度加入培养液中。在29 °C下孵育4小时,加入等量的甲醇终止反应。收集反应上清液进行HPLC-MS分析。
LC/MS分析
所有反应均使用C-18色谱柱(4.6 × 250 mm;2.5 μm)进行分析。对于AsC2′H,梯度包含溶剂A(0.1%甲酸的超纯水)和溶剂B(甲醇),方法如下:0分钟,10% B;5分钟,15% B;15分钟,60% B;22分钟,60% B。流速保持在0.5 mL/min。对于PTs和CYPs,梯度包含溶剂A(0.1%甲酸的超纯水)和溶剂B(甲醇),方法如下:0分钟:70:30(v/v),5分钟:35:65(v/v),12分钟:95:5(v/v),14分钟:95:5(v/v),16分钟:70:30(v/v),22分钟:70:30(v/v)。流速保持在0.5 mL/min。使用Shimadzu LC-2010AT在340 nm处检测对香豆酰CoA、伞形花内酯、DMS、osthenol、marmesin和columbianetin的检测波长。对于MS分析,使用Agilent Poroshell 120 SB-Aq(3.0 × 150 mm, 2.7 μm),流动相由0.1% HCOOH水溶液和甲醇组成。线性洗脱条件如下:0分钟:85:15(v/v),3分钟:85:15(v/v),8分钟:20:80(v/v),12分钟:5:95(v/v),16分钟:85:15(v/v),18分钟:85:15(v/v)。ESI源的条件如下:干燥气体(N2)流速,8.0 L/min;碰撞能量,35 eV;喷雾电压,3.5 kV;毛细管温度,320 °C;辅助气体加热温度,300 °C;鞘气流速,35 arb;辅助气流速,15 arb;扫气流速,5 arb。所有使用的气体,包括辅助气体、鞘气和扫气,均为高纯度氮气。所有操作和数据分析均在正离子模式下进行。
PpPT1和PpPT2的定点突变
根据对接结果和活性位点分析设计了用于定点突变的特定引物(补充表16)。使用KOD-plus-neo通过PCR方法进行定点突变。从E. coli DH5α中提取突变体用于测序,随后将阳性质粒转移到E. coli BL21(DE3)中进行蛋白表达和活性测试。最后,功能验证将检测突变对DMS和osthenol产量的影响。
统计与重复性
使用GraphPad Prism 9.5软件进行常规统计分析。两尾Student's t检验用于计算样本或组之间的显著差异。除非特别说明,所有反应均进行三次生物学重复(n = 3)。由相应空载体产生的酶或粗蛋白用于阴性对照。





























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








