「写在前面」
学习一个软件最好的方法就是啃它的官方文档。本着自己学习、分享他人的态度,分享官方文档的中文教程。软件可能随时更新,建议配合官方文档一起阅读。推荐先按顺序阅读往期内容:
文献篇:
1.文献阅读:Squidpy: 一个可扩展的空间组学分析框架
目录
-
1 AnnData 中的空间坐标 -
2 AnnData 中的组织图像
官网教程:
https://squidpy.readthedocs.io/en/stable/notebooks/tutorials/tutorial_read_spatial.html
本教程介绍如何在 anndata.AnnData 中存储空间数据集。
空间分子数据有多种不同的格式,迄今为止,还没有一种通用的解决方案可以用 Python 读取空间数据。Scanpy 已经通过 scanpy.read_visium() 函数为 Visium 空间转录组数据提供了解决方案,但基本上就是这样。在 Squidpy 中,我们确实通过 squidpy.datasets 模块提供了一些预处理(和预格式化)的数据集,但对于需要导入自己数据的用户来说并不是很有用。
在本教程中,我们将展示空间数据如何存储在 anndata.AnnData 中。为此,我们将使用模拟数据集,并通过示例展示您应该注意的重要细节,以便利用 AnnData-Scanpy-Squidpy 生态系统的全部功能。
from numpy.random import default_rng
import matplotlib.pyplot as plt
import scanpy as sc
import squidpy as sq
from anndata import AnnData
sc.logging.print_header()
print(f"squidpy=={sq.__version__}")
## scanpy==1.9.2 anndata==0.8.0 umap==0.5.3 numpy==1.23.5 scipy==1.9.3 pandas==1.5.1 scikit-learn==1.1.3 statsmodels==0.13.2 python-igraph==0.10.2 pynndescent==0.5.7 squidpy==1.2.3
1 AnnData 中的空间坐标
首先,让我们生成一些数据。我们会需要:
-
an array of features (e.g. counts). -
an array of spatial coordinates. -
an image array (e.g. the tissue image).
rng = default_rng(42)
counts = rng.integers(0, 15, size=(10, 100)) # feature matrix
coordinates = rng.uniform(0, 10, size=(10, 2)) # spatial coordinates
image = rng.uniform(0, 1, size=(10, 10, 3)) # image
我们首先从创建 anndata.AnnData 对象开始。我们首先将使用计数矩阵和空间坐标。将 anndata.AnnData.obsm 的 key 指定为 'spatial' 并不是绝对必要的,但会节省您大量的输入,因为它是 Squidpy 和 Scanpy 的默认值。
adata = AnnData(counts, obsm={"spatial": coordinates})
接下来,让我们运行标准的 Scanpy 聚类和 umap 工作流程。
sc.pp.normalize_total(adata)
sc.pp.log1p(adata)
sc.pp.pca(adata)
sc.pp.neighbors(adata)
sc.tl.umap(adata)
sc.tl.leiden(adata)
adata
## WARNING: n_obs too small: adjusting to `n_neighbors = 6`
## AnnData object with n_obs × n_vars = 10 × 100
## obs: 'leiden'
## uns: 'log1p', 'pca', 'neighbors', 'umap', 'leiden'
## obsm: 'spatial', 'X_pca', 'X_umap'
## varm: 'PCs'
## obsp: 'distances', 'connectivities'
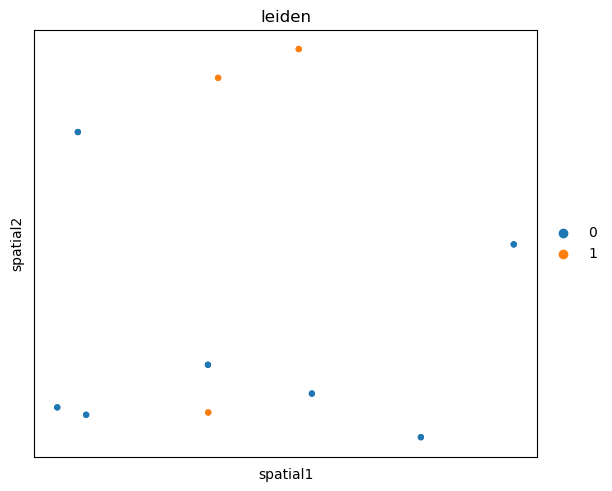
我们可以在空间中可视化虚拟类群注释 adata.obs['leiden']。
sq.pl.spatial_scatter(adata, shape=None, color="leiden", size=50)
2 AnnData 中的组织图像
对于没有组织图像的用例,这就是您开始使用 Scanpy/Squidpy 进行分析所需的全部内容。例如,您可以使用 squidpy.gr.spatial_neighbors() 基于固定邻居半径计算空间图,该半径在给定您的实验设置的情况下提供信息。
sq.gr.spatial_neighbors(adata, radius=3.0)
sq.pl.spatial_scatter(
adata,
color="leiden",
connectivity_key="spatial_connectivities",
edges_color="black",
shape=None,
edges_width=1,
size=3000,
)

如果您确实有组织图像(或多张,分辨率不同),则需要了解这些信息才能将其正确存储在 AnnData 中。首先,让我们可视化之前的模拟图像。
plt.imshow(image)

图像及其 metadata 存储在 anndata.AnnData 的 uns 插槽中。具体来说,在 adata.uns['spatial'][<library_id>] 插槽中,其中 library_id 是引用组织图像的任何唯一 key。
现在,我们假设只有一张图像。这是必要的 metadata:
-
tissue_hires_scalef- 这是空间坐标单位和图像像素之间的比例因子。对于 Visium,该值通常约为 0.17。在这种情况下,我们假设空间坐标与像素具有相同的比例,因此我们将该值设置为 1。 -
spot_diameter_fullres- 这是每次观察的捕获区域的直径。对于 Visium,我们通常将它们称为"spots",并且该值设置为约 89。
在这里,我们将其设置为 0.5。
spatial_key = "spatial"
library_id = "tissue42"
adata.uns[spatial_key] = {library_id: {}}
adata.uns[spatial_key][library_id]["images"] = {}
adata.uns[spatial_key][library_id]["images"] = {"hires": image}
adata.uns[spatial_key][library_id]["scalefactors"] = {
"tissue_hires_scalef": 1,
"spot_diameter_fullres": 0.5,
}
我们(尚)不提供更改此类 keys 值的灵活性。这些是 10x Genomics Visium 的 Space Ranger 输出提供的 keys,因此是第一个被采用的 keys。将来,我们可能会采用一种截然不同的结构。但目前,如果所有这些 keys 都是正确的,squidpy.pl.spatial_scatter() 可以开箱即用。
sq.pl.spatial_scatter(adata, color="leiden")

您可以修改设置以查看发生了什么变化。例如,让我们将 tissue_hires_scalef 更改为之前值的一半。
adata.uns[spatial_key][library_id]["scalefactors"] = {
"tissue_hires_scalef": 0.5,
"spot_diameter_fullres": 0.5,
}
sq.pl.spatial_scatter(adata, color="leiden", size=2)
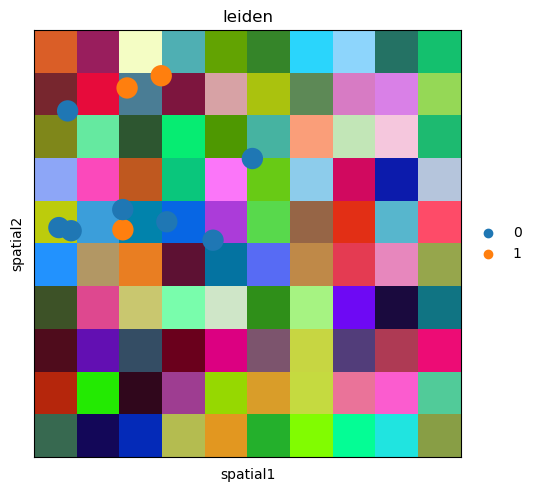
正如你所看到的,空间坐标被缩小了,图像被“放大”了。
当然,您可能想“分析”这样的图像。squidpy.im.ImageContainer 来救援!只需实例化一个新对象,它就可以开箱即用。
img = sq.im.ImageContainer(image)
img.show()


本文由 mdnice 多平台发布

























 1004
1004

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










