「写在前面」
学习一个软件最好的方法就是啃它的官方文档。本着自己学习、分享他人的态度,分享官方文档的中文教程。软件可能随时更新,建议配合官方文档一起阅读。推荐先按顺序阅读往期内容:
文献篇:
1.文献阅读:(Seurat V1) 单细胞基因表达数据的空间重建
2.文献阅读:(Seurat V2) 整合跨越不同条件、技术、物种的单细胞转录组数据
3.文献阅读:(Seurat V3) 单细胞数据综合整合
4.文献阅读:(Seurat V4) 整合分析多模态单细胞数据
5.文献阅读:(Seurat V5) 用于集成、多模态和可扩展单细胞分析的字典学习
教程篇:
1.Seurat Tutorial 1:常见分析工作流程,基于 PBMC 3K 数据集
2.Seurat Tutorial 2:使用 Seurat 分析多模态数据
3.Seurat Tutorial 3:scRNA-seq 整合分析介绍
4.Seurat Tutorial 4:映射和注释查询数据集
5.Seurat Tutorial 5:使用 reciprocal PCA (RPCA) 快速整合
6.Seurat Tutorial 6:整合大型数据集的技巧
7.Seurat Tutorial 7:整合 scRNA-seq 和 scATAC-seq 数据
8.Seurat Tutorial 8:多模态参考映射
目录
-
1 WNN analysis of CITE-seq, RNA + ADT -
2 WNN analysis of 10x Multiome, RNA + ATAC
官网教程:
https://satijalab.org/seurat/articles/weighted_nearest_neighbor_analysis
多模态的同时测量,称为多模态分析,代表了单细胞基因组学的一个令人兴奋的前沿领域,并且需要能够基于多种数据类型定义细胞状态的新计算方法。每种模态的信息内容不同,甚至在同一数据集中的细胞之间也是如此,这对多模态数据集的分析和整合来说是一个紧迫的挑战。在(Hao* et al, Cell 2021)中,我们引入了 'weighted-nearest neighbor'(WNN) 分析,这是一种无监督框架,用于学习每个细胞中每种数据类型的相对效用,从而实现多种模态的整合分析。
本小节介绍了用于分析多模态单细胞数据集的 WNN 工作流程。工作流程由三个步骤组成:
-
每种模态单独进行独立预处理和降维 -
学习细胞-特异性模态“权重”,并构建一个 WNN graph 整合模态 -
WNN graph 的下游分析(即,可视化、聚类等)
我们演示了 WNN 分析在两种单细胞多模态技术中的应用:CITE-seq 和 10x multiome。我们根据两种模态而不是任何一种单独的模态来定义细胞状态。
1 WNN analysis of CITE-seq, RNA + ADT
我们使用来自 (Stuart*, Butler* et al, Cell 2019) 的 CITE-seq 数据集,该数据集包含 30,672 个 scRNA-seq 谱,与一组来自骨髓的 25 种抗体一起测量。该对象包含两种 assays:RNA 和 antibody-derived tags (ADT)。
要运行此小节,请安装 CRAN 上提供的 Seurat v4 和 GitHub 上提供的 SeuratData。
install.packages("Seurat")
library(Seurat)
library(SeuratData)
library(cowplot)
library(dplyr)
#InstallData("bmcite")
bm <- LoadData(ds = "bmcite")
我们首先对两种 assays 独立进行预处理和降维。我们使用标准 normalization,但您也可以使用 SCTransform 或任何替代方法。
DefaultAssay(bm) <- 'RNA'
bm <- NormalizeData(bm) %>% FindVariableFeatures() %>% ScaleData() %>% RunPCA()
DefaultAssay(bm) <- 'ADT'
# we will use all ADT features for dimensional reduction
# we set a dimensional reduction name to avoid overwriting the
VariableFeatures(bm) <- rownames(bm[["ADT"]])
bm <- NormalizeData(bm, normalization.method = 'CLR', margin = 2) %>%
ScaleData() %>% RunPCA(reduction.name = 'apca')
对于每个细胞,我们根据 RNA 和 protein 相似性的加权组合计算数据集中其最近的邻居。细胞特定模态权重和多模态邻居在单个函数中计算,在该数据集上运行大约需要 2 分钟。我们指定每种模态的维度(类似于指定 scRNA-seq 聚类中包含的 PCs 数量),但您可以改变这些设置,以查看微小的变化对整体结果的影响最小。
# 识别多模态邻居。这些将被存储在 neighbors slot 中,
# 并且可以使用 bm[['weighted.nn']] 访问
# WNN graph 可以通过 bm[["wknn"]] 访问,
# 并且用于聚类的 SNN graph 在 bm[["wsnn"]] 中
# 细胞特异性模态权重可以通过 bm$RNA.weight 访问
bm <- FindMultiModalNeighbors(
bm, reduction.list = list("pca", "apca"),
dims.list = list(1:30, 1:18), modality.weight.name = "RNA.weight"
)
我们现在可以使用这些结果进行下游分析,例如可视化和聚类。例如,我们可以基于 RNA 和 protein 数据的加权组合创建数据的 UMAP 可视化。我们还可以执行 graph-based 聚类,并在 UMAP 上可视化这些结果以及一组细胞注释。
bm <- RunUMAP(bm, nn.name = "weighted.nn", reduction.name = "wnn.umap", reduction.key = "wnnUMAP_")
bm <- FindClusters(bm, graph.name = "wsnn", algorithm = 3, resolution = 2, verbose = FALSE)
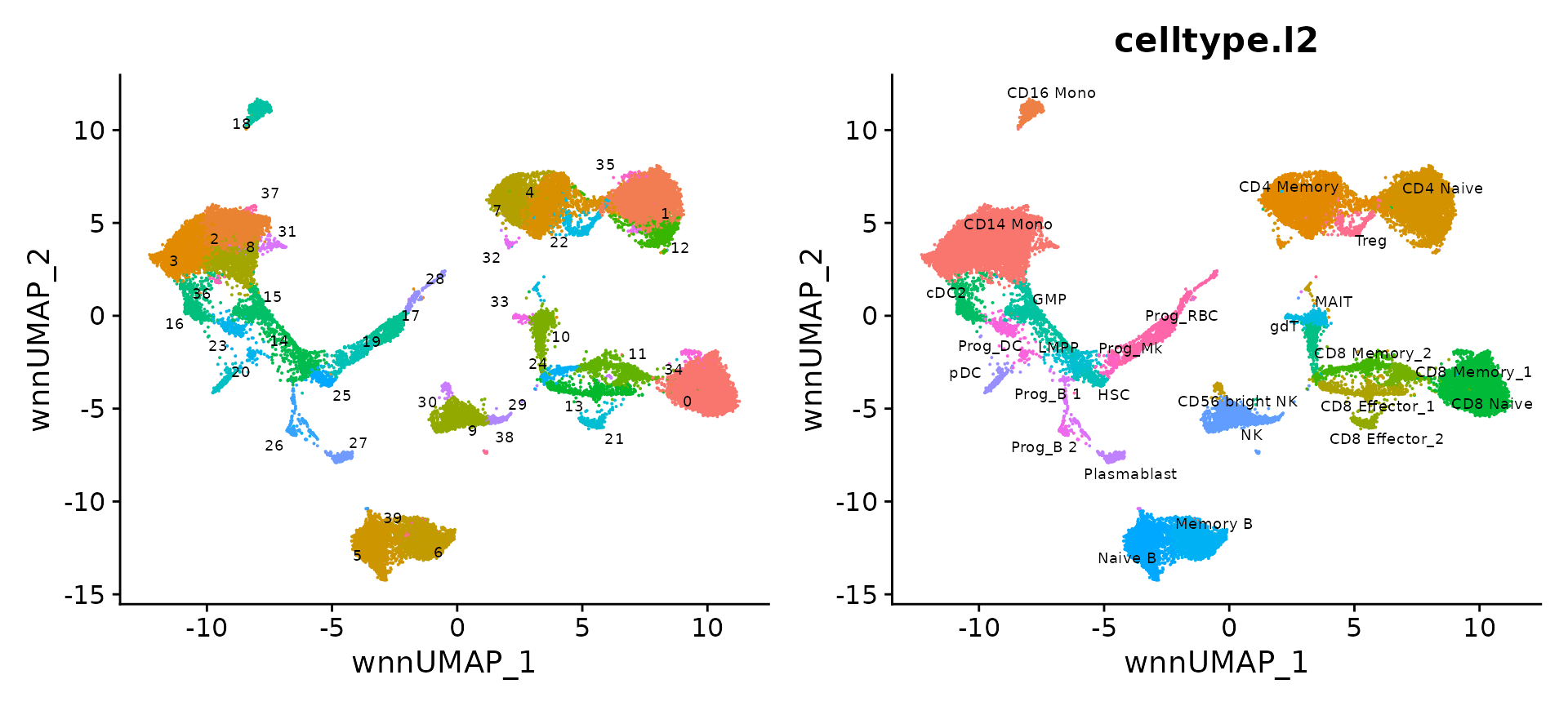
p1 <- DimPlot(bm, reduction = 'wnn.umap', label = TRUE, repel = TRUE, label.size = 2.5) + NoLegend()
p2 <- DimPlot(bm, reduction = 'wnn.umap', group.by = 'celltype.l2', label = TRUE, repel = TRUE, label.size = 2.5) + NoLegend()
p1 + p2
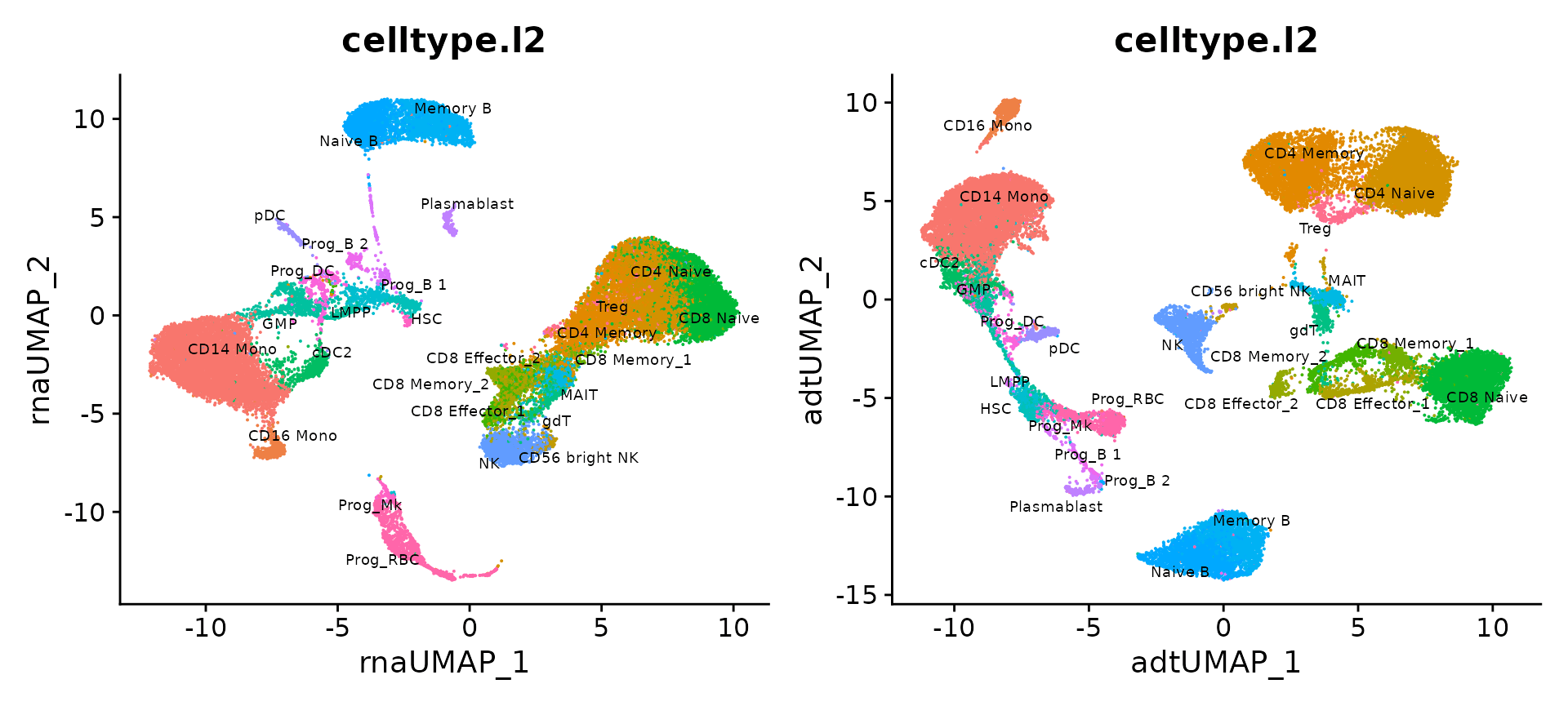
我们还可以仅基于 RNA 和 protein 数据计算 UMAP 可视化并进行比较。我们发现,在识别祖细胞状态方面,RNA 分析比 ADT 分析提供更多信息(ADT panel 包含分化细胞的标记),而 T 细胞状态则相反(ADT 分析优于 RNA)。
bm <- RunUMAP(bm, reduction = 'pca', dims = 1:30, assay = 'RNA',
reduction.name = 'rna.umap', reduction.key = 'rnaUMAP_')
bm <- RunUMAP(bm, reduction = 'apca', dims = 1:18, assay = 'ADT',
reduction.name = 'adt.umap', reduction.key = 'adtUMAP_')
p3 <- DimPlot(bm, reduction = 'rna.umap', group.by = 'celltype.l2', label = TRUE,
repel = TRUE, label.size = 2.5) + NoLegend()
p4 <- DimPlot(bm, reduction = 'adt.umap', group.by = 'celltype.l2', label = TRUE,
repel = TRUE, label.size = 2.5) + NoLegend()
p3 + p4
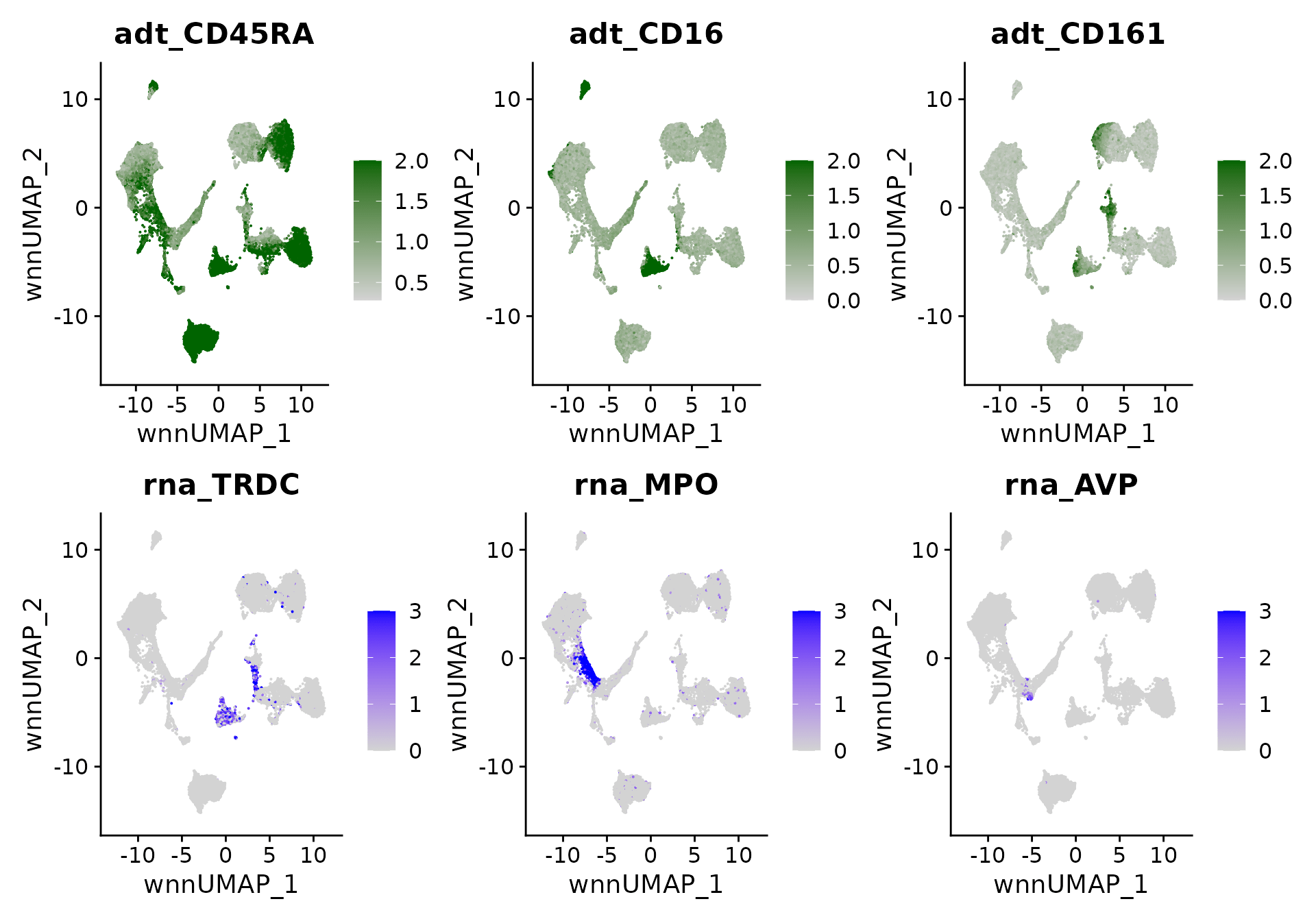
我们可以在多模态 UMAP 上可视化规范标记基因和蛋白质的表达,这可以帮助验证所提供的注释:
p5 <- FeaturePlot(bm, features = c("adt_CD45RA","adt_CD16","adt_CD161"),
reduction = 'wnn.umap', max.cutoff = 2,
cols = c("lightgrey","darkgreen"), ncol = 3)
p6 <- FeaturePlot(bm, features = c("rna_TRDC","rna_MPO","rna_AVP"),
reduction = 'wnn.umap', max.cutoff = 3, ncol = 3)
p5 / p6
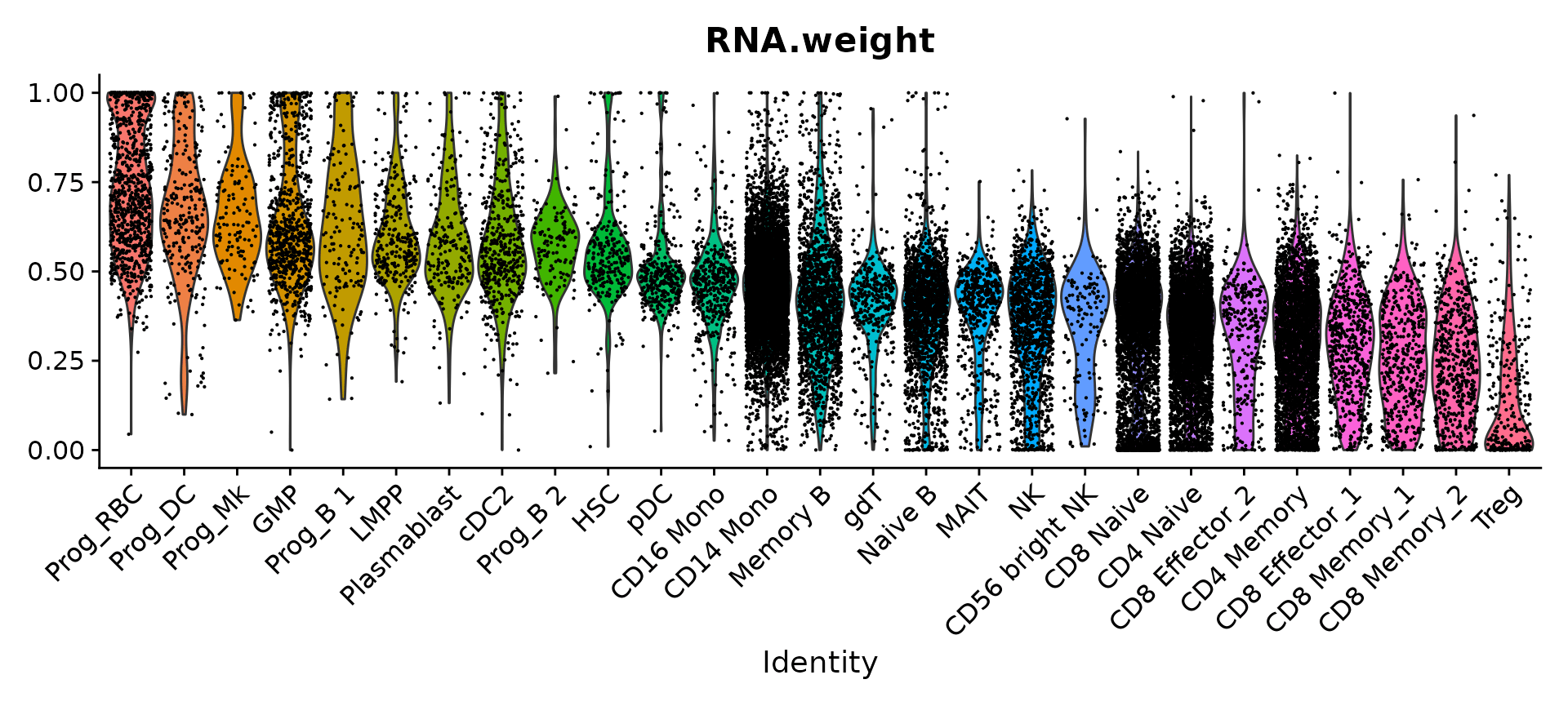
最后,我们可以可视化为每个细胞学习的模态权重。每个具有最高 RNA 权重的群体代表祖细胞,而具有最高 protein 权重的群体代表 T 细胞。这符合我们的生物学预期,因为抗体组不包含可以区分不同祖细胞群的标记。
VlnPlot(bm, features = "RNA.weight", group.by = 'celltype.l2', sort = TRUE, pt.size = 0.1) +
NoLegend()
2 WNN analysis of 10x Multiome, RNA + ATAC
在这里,我们演示了 WNN 分析在第二种多模式技术,10x multiome RNA+ATAC kit 中的使用。我们使用 10x 网站上公开的数据集,其中在 10,412 个 PBMCs 中测量了配对 transcriptome 和 ATAC-seq 谱。
我们使用与上一个小标题中使用的相同的 WNN 方法,其中我们将整合多模态分析应用于 CITE-seq 数据集。在此示例中,我们将演示如何:
-
创建具有配对 transcriptome 和 ATAC-seq 谱的多模态 Seurat 对象 -
对单细胞中的 RNA+ATAC 数据执行加权邻居聚类 -
利用这两种模态来识别不同细胞类型和状态的假定调节因子
您可以从此处的 10x Genomics 网站下载数据集。请确保下载以下文件:
-
Filtered feature barcode matrix (HDF5) -
ATAC Per fragment information file (TSV.GZ) -
ATAC Per fragment information index (TSV.GZ index)
最后,为了运行小节,请确保安装了以下软件包:
-
Seurat v4 -
Signac for the analysis of single-cell chromatin datasets -
EnsDb.Hsapiens.v86 for a set of annotations for hg38 -
dplyr to help manipulate data tables
library(Seurat)
library(Signac)
library(EnsDb.Hsapiens.v86)
library(dplyr)
library(ggplot2)
我们将根据基因表达数据创建 Seurat 对象,然后添加 ATAC-seq 数据作为第二个 assay。您可以浏览 Signac getting started vignette,了解有关 ChromatinAssay 对象的创建和处理的更多信息。
# the 10x hdf5 file contains both data types.
inputdata.10x <- Read10X_h5("../data/pbmc_granulocyte_sorted_10k_filtered_feature_bc_matrix.h5")
# extract RNA and ATAC data
rna_counts <- inputdata.10x$`Gene Expression`
atac_counts <- inputdata.10x$Peaks
# Create Seurat object
pbmc <- CreateSeuratObject(counts = rna_counts)
pbmc[["percent.mt"]] <- PercentageFeatureSet(pbmc, pattern = "^MT-")
# Now add in the ATAC-seq data
# we'll only use peaks in standard chromosomes
grange.counts <- StringToGRanges(rownames(atac_counts), sep = c(":", "-"))
grange.use <- seqnames(grange.counts) %in% standardChromosomes(grange.counts)
atac_counts <- atac_counts[as.vector(grange.use), ]
annotations <- GetGRangesFromEnsDb(ensdb = EnsDb.Hsapiens.v86)
seqlevelsStyle(annotations) <- 'UCSC'
genome(annotations) <- "hg38"
frag.file <- "../data/pbmc_granulocyte_sorted_10k_atac_fragments.tsv.gz"
chrom_assay <- CreateChromatinAssay(
counts = atac_counts,
sep = c(":", "-"),
genome = 'hg38',
fragments = frag.file,
min.cells = 10,
annotation = annotations
)
pbmc[["ATAC"]] <- chrom_assay
我们根据每种模态检测到的分子数量以及线粒体百分比进行基本的质量控制。
VlnPlot(pbmc, features = c("nCount_ATAC", "nCount_RNA","percent.mt"), ncol = 3,
log = TRUE, pt.size = 0) + NoLegend()
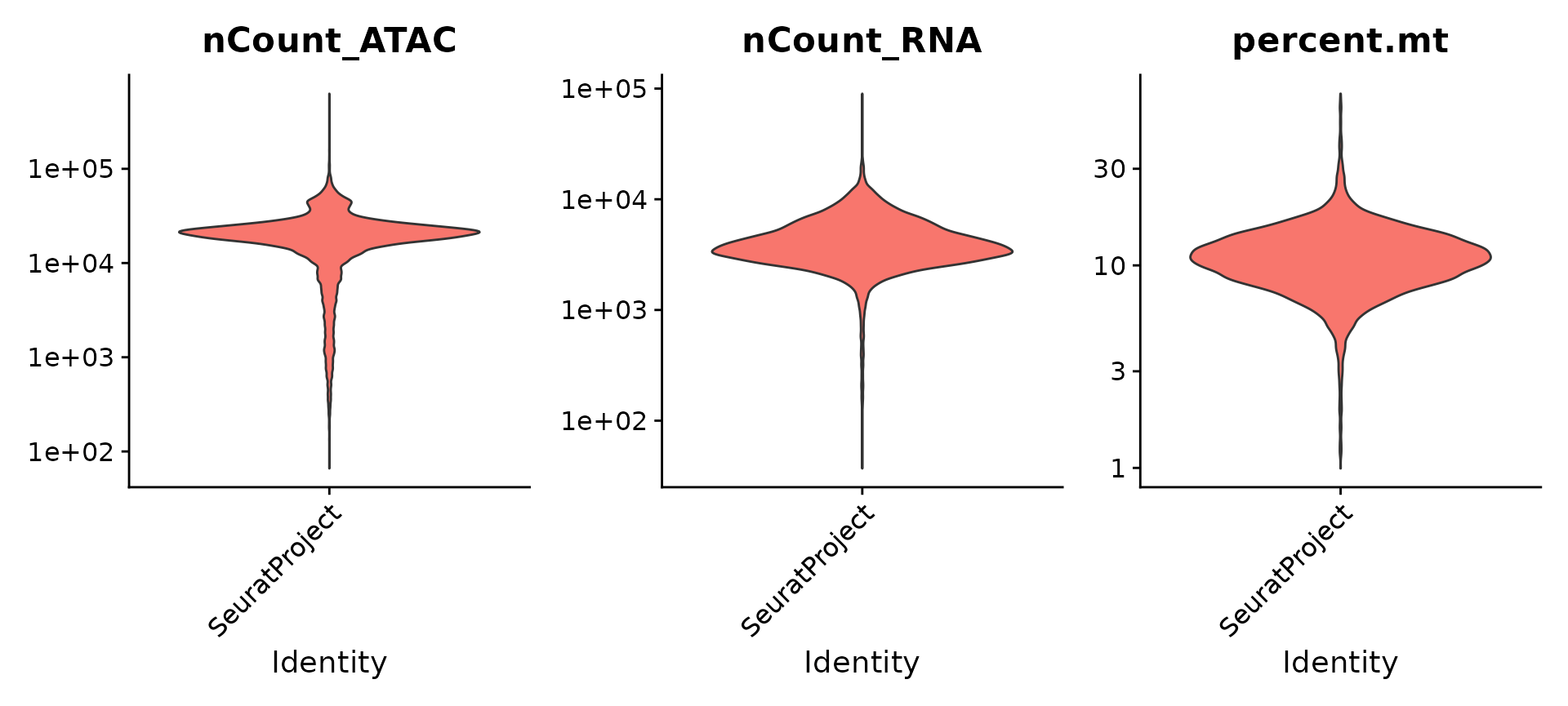
#| eval: false
pbmc <- subset(
x = pbmc,
subset = nCount_ATAC < 7e4 &
nCount_ATAC > 5e3 &
nCount_RNA < 25000 &
nCount_RNA > 1000 &
percent.mt < 20
)
接下来,我们使用 RNA 和 ATAC-seq 数据的标准方法独立地对两种 assays 进行预处理和降维。
# RNA analysis
DefaultAssay(pbmc) <- "RNA"
pbmc <- SCTransform(pbmc, verbose = FALSE) %>% RunPCA() %>% RunUMAP(dims = 1:50, reduction.name = 'umap.rna', reduction.key = 'rnaUMAP_')
# ATAC analysis
# We exclude the first dimension as this is typically correlated with sequencing depth
DefaultAssay(pbmc) <- "ATAC"
pbmc <- RunTFIDF(pbmc)
pbmc <- FindTopFeatures(pbmc, min.cutoff = 'q0')
pbmc <- RunSVD(pbmc)
pbmc <- RunUMAP(pbmc, reduction = 'lsi', dims = 2:50, reduction.name = "umap.atac", reduction.key = "atacUMAP_")
我们计算一个 WNN graph,代表 RNA 和 ATAC-seq 模态的加权组合。我们使用该 graph 进行 UMAP 可视化和聚类
#| eval: false
pbmc <- FindMultiModalNeighbors(pbmc, reduction.list = list("pca", "lsi"), dims.list = list(1:50, 2:50))
pbmc <- RunUMAP(pbmc, nn.name = "weighted.nn", reduction.name = "wnn.umap", reduction.key = "wnnUMAP_")
pbmc <- FindClusters(pbmc, graph.name = "wsnn", algorithm = 3, verbose = FALSE)
我们对下面的 clusters 进行注释。请注意,您还可以使用我们的 supervised mapping pipelines 对数据集进行注释,使用我们的 vignette 或 automated web tool, Azimuth 。
# perform sub-clustering on cluster 6 to find additional structure
pbmc <- FindSubCluster(pbmc, cluster = 6, graph.name = "wsnn", algorithm = 3)
Idents(pbmc) <- "sub.cluster"
# add annotations
pbmc <- RenameIdents(pbmc, '19' = 'pDC','20' = 'HSPC','15' = 'cDC')
pbmc <- RenameIdents(pbmc, '0' = 'CD14 Mono', '9' ='CD14 Mono', '5' = 'CD16 Mono')
pbmc <- RenameIdents(pbmc, '10' = 'Naive B', '11' = 'Intermediate B', '17' = 'Memory B', '21' = 'Plasma')
pbmc <- RenameIdents(pbmc, '7' = 'NK')
pbmc <- RenameIdents(pbmc, '4' = 'CD4 TCM', '13'= "CD4 TEM", '3' = "CD4 TCM", '16' ="Treg", '1' ="CD4 Naive", '14' = "CD4 Naive")
pbmc <- RenameIdents(pbmc, '2' = 'CD8 Naive', '8'= "CD8 Naive", '12' = 'CD8 TEM_1', '6_0' = 'CD8 TEM_2', '6_1' ='CD8 TEM_2', '6_4' ='CD8 TEM_2')
pbmc <- RenameIdents(pbmc, '18' = 'MAIT')
pbmc <- RenameIdents(pbmc, '6_2' ='gdT', '6_3' = 'gdT')
pbmc$celltype <- Idents(pbmc)
我们可以基于基因表达、ATAC-seq 或 WNN 分析可视化聚类。差异比之前的分析更加微妙(您可以探索权重,权重比我们的 CITE-seq 示例中的分布更均匀),但我们发现 WNN 提供了最清晰的细胞状态分离。
p1 <- DimPlot(pbmc, reduction = "umap.rna", group.by = "celltype", label = TRUE, label.size = 2.5, repel = TRUE) + ggtitle("RNA")
p2 <- DimPlot(pbmc, reduction = "umap.atac", group.by = "celltype", label = TRUE, label.size = 2.5, repel = TRUE) + ggtitle("ATAC")
p3 <- DimPlot(pbmc, reduction = "wnn.umap", group.by = "celltype", label = TRUE, label.size = 2.5, repel = TRUE) + ggtitle("WNN")
p1 + p2 + p3 & NoLegend() & theme(plot.title = element_text(hjust = 0.5))
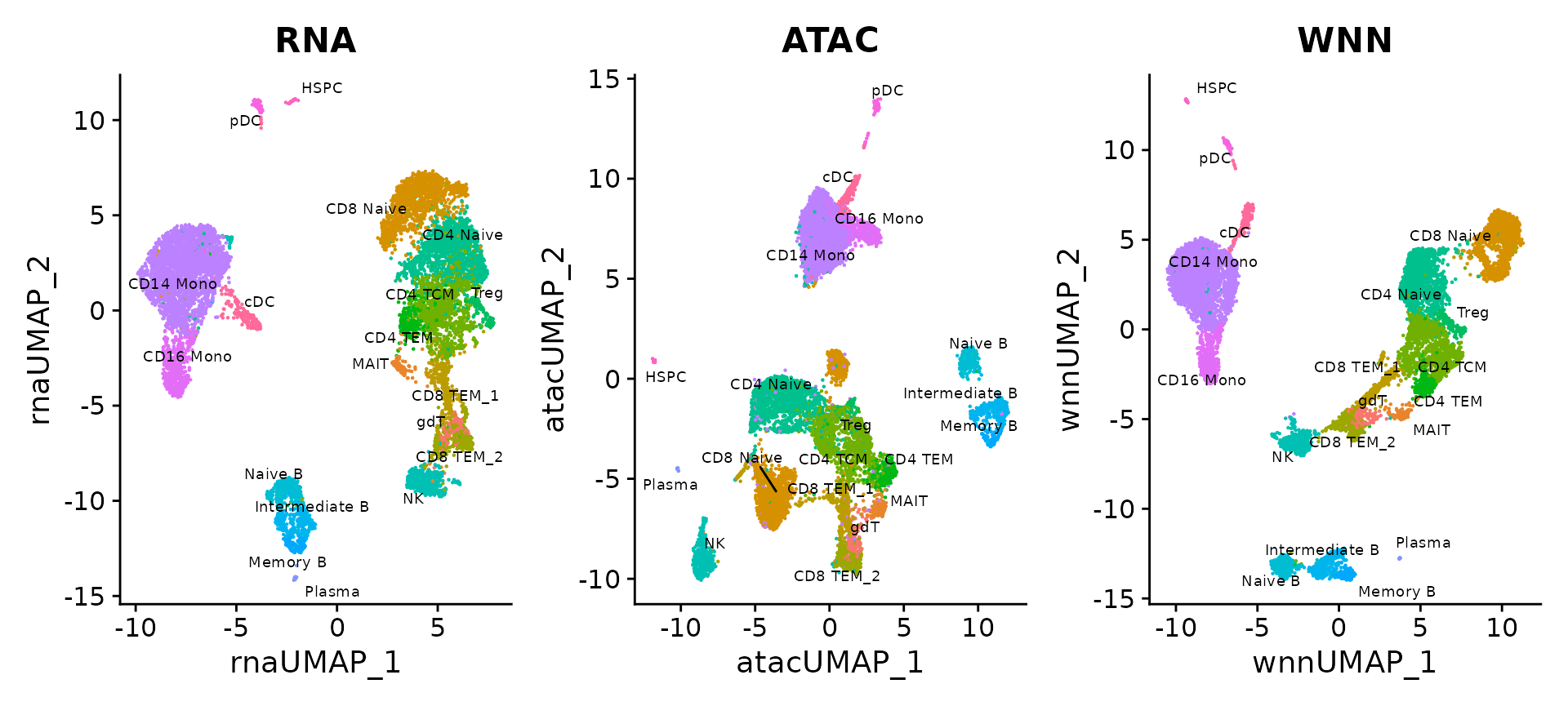
例如,ATAC-seq 数据有助于分离 CD4 和 CD8 T 细胞状态。这是由于存在多个位点,这些位点在不同 T 细胞亚型之间表现出不同的可及性。例如,我们可以使用 Signac visualization vignette 中的工具,将 CD8A 基因座的“pseudobulk”轨迹与基因表达水平的小提琴图一起可视化。
## to make the visualization easier, subset T cell clusters
celltype.names <- levels(pbmc)
tcell.names <- grep("CD4|CD8|Treg", celltype.names,value = TRUE)
tcells <- subset(pbmc, idents = tcell.names)
CoveragePlot(tcells, region = 'CD8A', features = 'CD8A', assay = 'ATAC', expression.assay = 'SCT', peaks = FALSE)
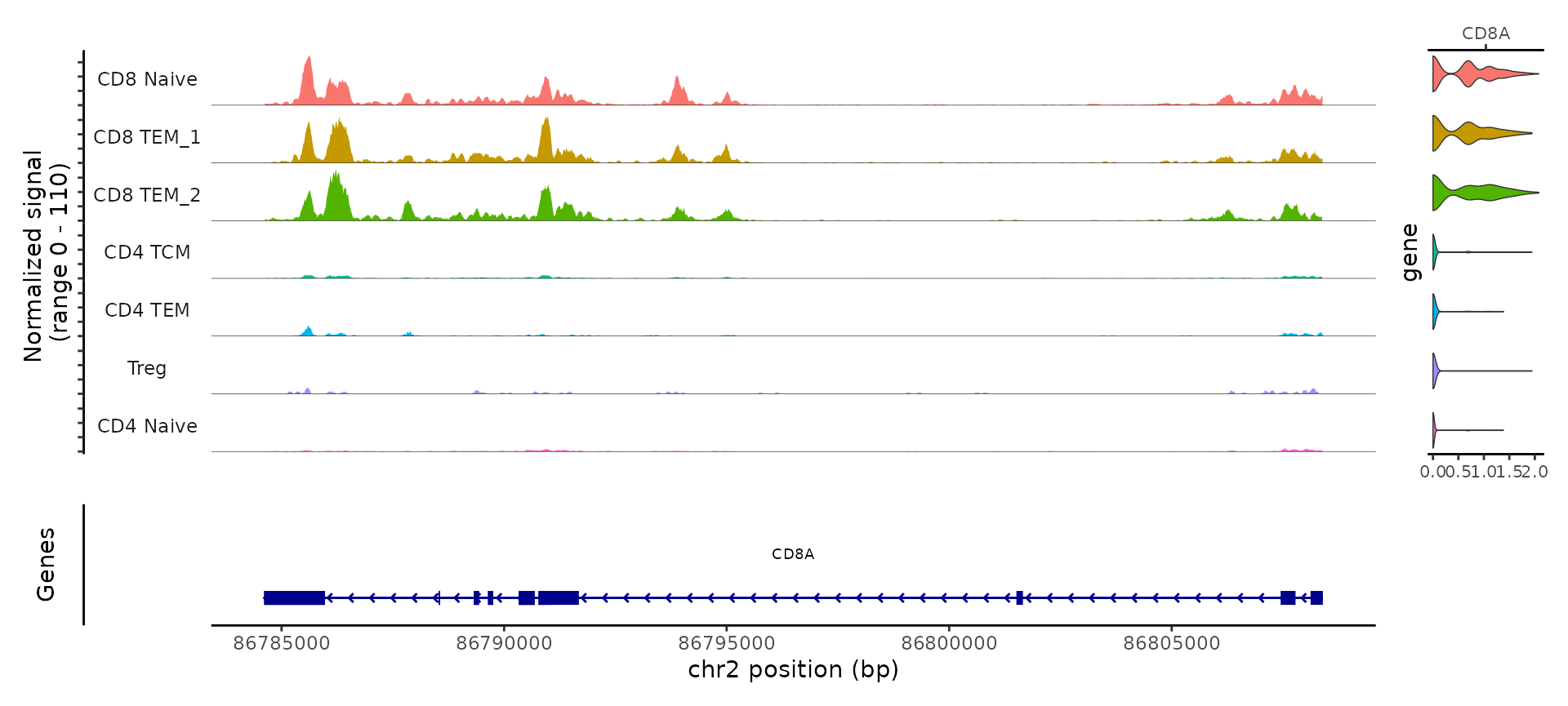
接下来,我们将检查每个细胞的可访问区域以确定富集的 motifs。正如 Signac motifs vignette 中所述,有几种方法可以做到这一点,但我们将使用 Greenleaf 实验室的 chromVAR 包。这会计算已知 motifs 的每个细胞可及性分数,并将这些分数添加为 Seurat 对象中的第三个 assay (chromvar)。
要继续,请确保您已安装以下软件包。
-
chromVAR for the analysis of motif accessibility in scATAC-seq -
presto for fast differential expression analyses. -
TFBSTools for TFBS analysis -
JASPAR2020 for JASPAR motif models -
motifmatchr for motif matching -
BSgenome.Hsapiens.UCSC.hg38 for chromVAR
Install command for all dependencies:
remotes::install_github("immunogenomics/presto")
BiocManager::install(c("chromVAR", "TFBSTools", "JASPAR2020", "motifmatchr", "BSgenome.Hsapiens.UCSC.hg38"))
library(chromVAR)
library(JASPAR2020)
library(TFBSTools)
library(motifmatchr)
library(BSgenome.Hsapiens.UCSC.hg38)
# Scan the DNA sequence of each peak for the presence of each motif, and create a Motif object
DefaultAssay(pbmc) <- "ATAC"
pwm_set <- getMatrixSet(x = JASPAR2020, opts = list(species = 9606, all_versions = FALSE))
motif.matrix <- CreateMotifMatrix(features = granges(pbmc), pwm = pwm_set, genome = 'hg38', use.counts = FALSE)
motif.object <- CreateMotifObject(data = motif.matrix, pwm = pwm_set)
pbmc <- SetAssayData(pbmc, assay = 'ATAC', slot = 'motifs', new.data = motif.object)
# Note that this step can take 30-60 minutes
pbmc <- RunChromVAR(
object = pbmc,
genome = BSgenome.Hsapiens.UCSC.hg38
)
最后,我们探索多模态数据集来识别每个细胞状态的关键调节因子。配对数据提供了一个独特的机会来识别满足多个标准的转录因子(TFs),有助于将假定的调节因子列表缩小到最有可能的候选者。我们的目标是识别在 RNA 测量中表达在多种细胞类型中富集的 TFs,而且在 ATAC 测量中其 motifs 的可及性也富集。
作为示例和阳性对照,CCAAT 增强子结合蛋白 (CEBP) 蛋白家族(包括 TF CEBPB)已被多次证明在骨髓细胞(包括单核细胞和树突状细胞)的分化和功能中发挥重要作用。我们可以看到,CEBPB 的表达和 MA0466.2.4 motif(编码 CEBPB 的结合位点)的可及性都在单核细胞中富集。
#returns MA0466.2
motif.name <- ConvertMotifID(pbmc, name = 'CEBPB')
gene_plot <- FeaturePlot(pbmc, features = "sct_CEBPB", reduction = 'wnn.umap')
motif_plot <- FeaturePlot(pbmc, features = motif.name, min.cutoff = 0, cols = c("lightgrey", "darkred"), reduction = 'wnn.umap')
gene_plot | motif_plot
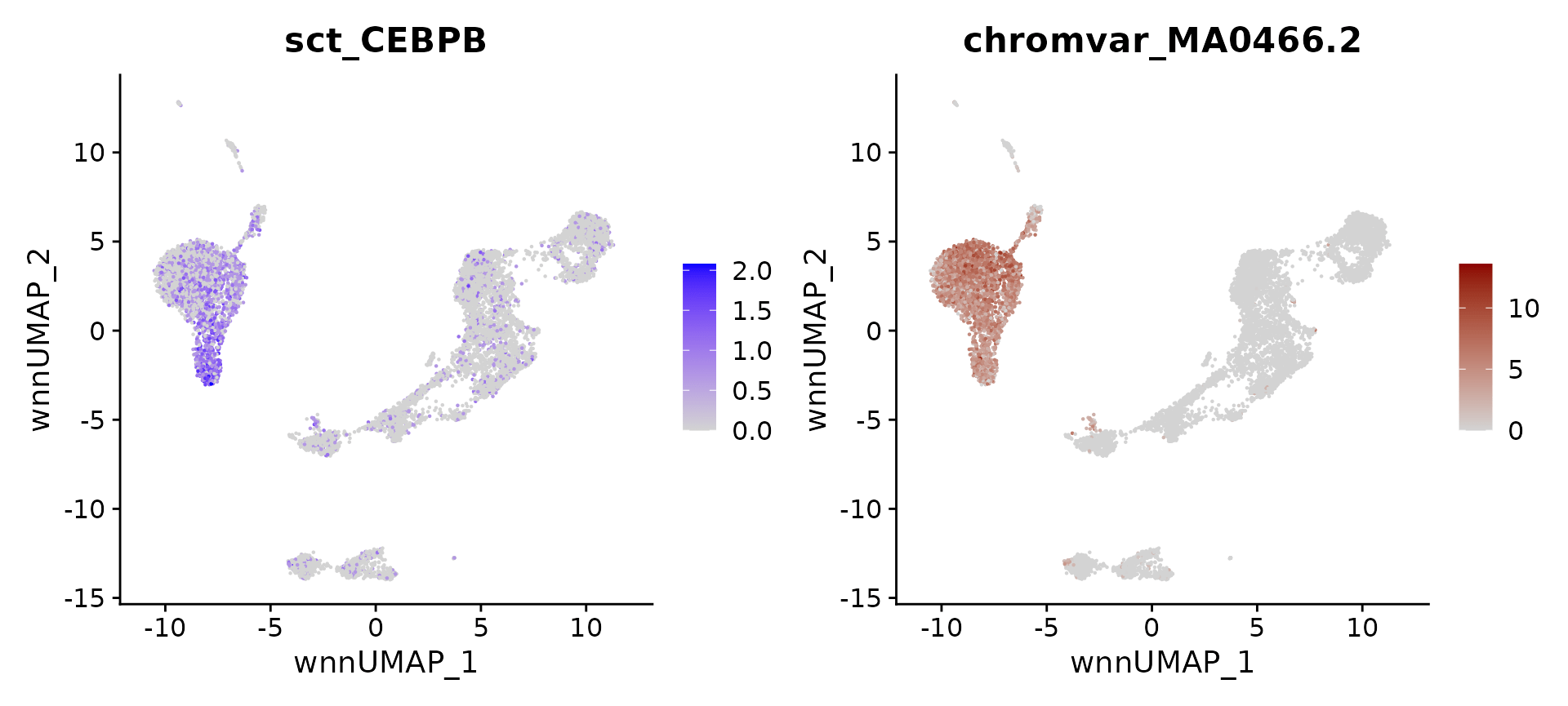
我们想要量化这种关系,并搜索所有细胞类型以找到类似的例子。为此,我们将使用 presto 包来执行快速差异表达。我们进行两项测试:一项使用基因表达数据,另一项使用 chromVAR motif 可及性。presto 基于 Wilcox 秩和检验计算 p 值,这也是 Seurat 中的默认检验,并且我们将搜索限制为在两个检验中都返回显着结果的 TFs。
presto 还计算“AUC”统计数据,反映每个基因(or motif)作为细胞类型标记的能力。最大 AUC 值为 1 表示完美标记。由于基因和 motifs 的 AUC 统计量具有相同的尺度,因此我们取两次测试中 AUC 值的平均值,并使用它对每种细胞类型的 TFs 进行排名:
markers_rna <- presto:::wilcoxauc.Seurat(X = pbmc, group_by = 'celltype', assay = 'data', seurat_assay = 'SCT')
markers_motifs <- presto:::wilcoxauc.Seurat(X = pbmc, group_by = 'celltype', assay = 'data', seurat_assay = 'chromvar')
motif.names <- markers_motifs$feature
colnames(markers_rna) <- paste0("RNA.", colnames(markers_rna))
colnames(markers_motifs) <- paste0("motif.", colnames(markers_motifs))
markers_rna$gene <- markers_rna$RNA.feature
markers_motifs$gene <- ConvertMotifID(pbmc, id = motif.names)
# a simple function to implement the procedure above
topTFs <- function(celltype, padj.cutoff = 1e-2) {
ctmarkers_rna <- dplyr::filter(
markers_rna, RNA.group == celltype, RNA.padj < padj.cutoff, RNA.logFC > 0) %>%
arrange(-RNA.auc)
ctmarkers_motif <- dplyr::filter(
markers_motifs, motif.group == celltype, motif.padj < padj.cutoff, motif.logFC > 0) %>%
arrange(-motif.auc)
top_tfs <- inner_join(
x = ctmarkers_rna[, c(2, 11, 6, 7)],
y = ctmarkers_motif[, c(2, 1, 11, 6, 7)], by = "gene"
)
top_tfs$avg_auc <- (top_tfs$RNA.auc + top_tfs$motif.auc) / 2
top_tfs <- arrange(top_tfs, -avg_auc)
return(top_tfs)
}
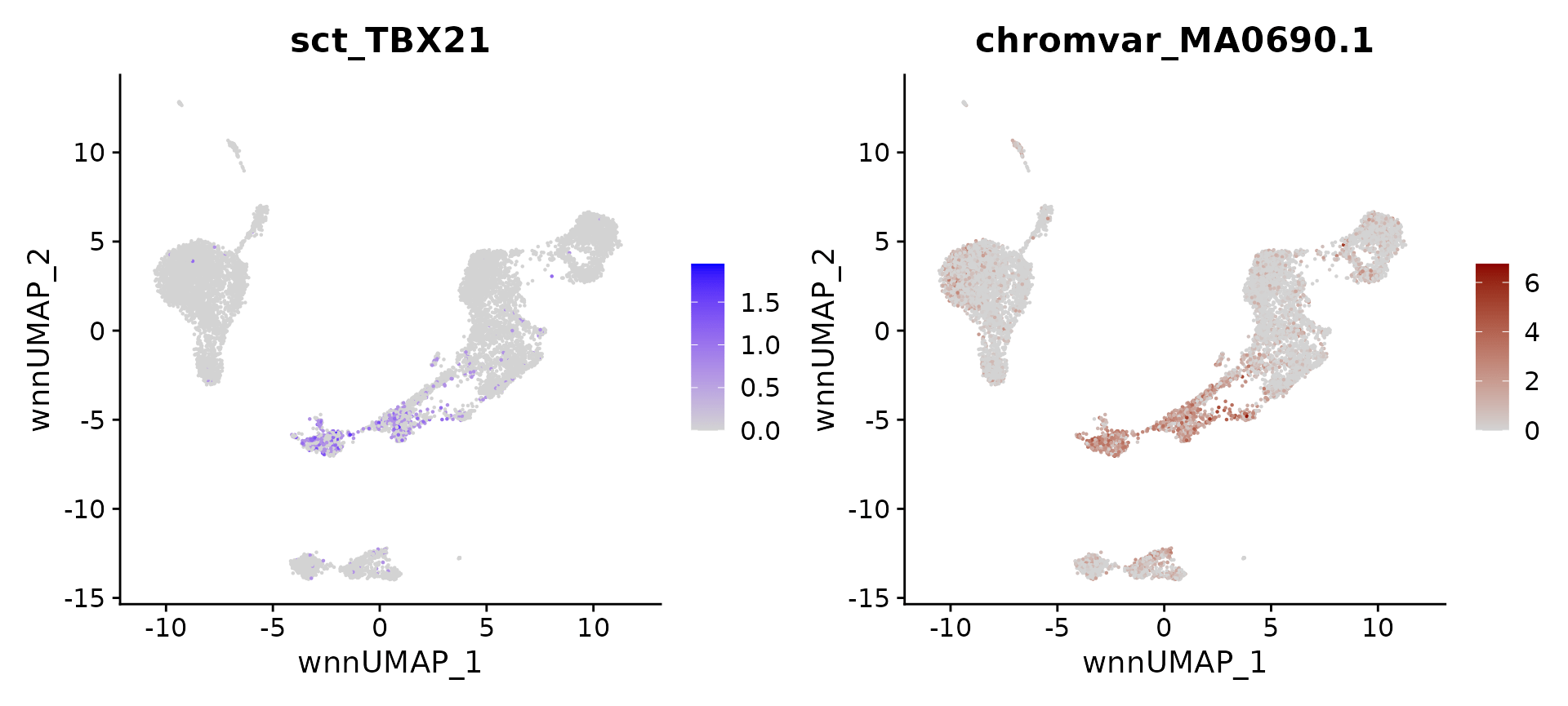
我们现在可以计算,并可视化,任何细胞类型的假定调节因子。我们恢复了成熟的调节因子,包括 TBX21 for NK cells, IRF4 for plasma cells, SOX4 for hematopoietic progenitors, EBF1 and PAX5 for B cells, IRF8 and TCF4 for pDC。我们相信,类似的策略可以用来帮助关注不同系统中的一组假定的调节因子。
# identify top markers in NK and visualize
head(topTFs("NK"), 3)
## RNA.group gene RNA.auc RNA.pval motif.group motif.feature motif.auc
## 1 NK TBX21 0.7270859 0.000000e+00 NK MA0690.1 0.9139614
## 2 NK EOMES 0.5886774 2.411013e-98 NK MA0800.1 0.9214773
## 3 NK RUNX3 0.7705661 5.977716e-120 NK MA0684.2 0.6769022
## motif.pval avg_auc
## 1 1.097940e-202 0.8205237
## 2 4.903866e-210 0.7550774
## 3 1.560061e-38 0.7237341
motif.name <- ConvertMotifID(pbmc, name = 'TBX21')
gene_plot <- FeaturePlot(pbmc, features = "sct_TBX21", reduction = 'wnn.umap')
motif_plot <- FeaturePlot(pbmc, features = motif.name, min.cutoff = 0, cols = c("lightgrey", "darkred"), reduction = 'wnn.umap')
gene_plot | motif_plot
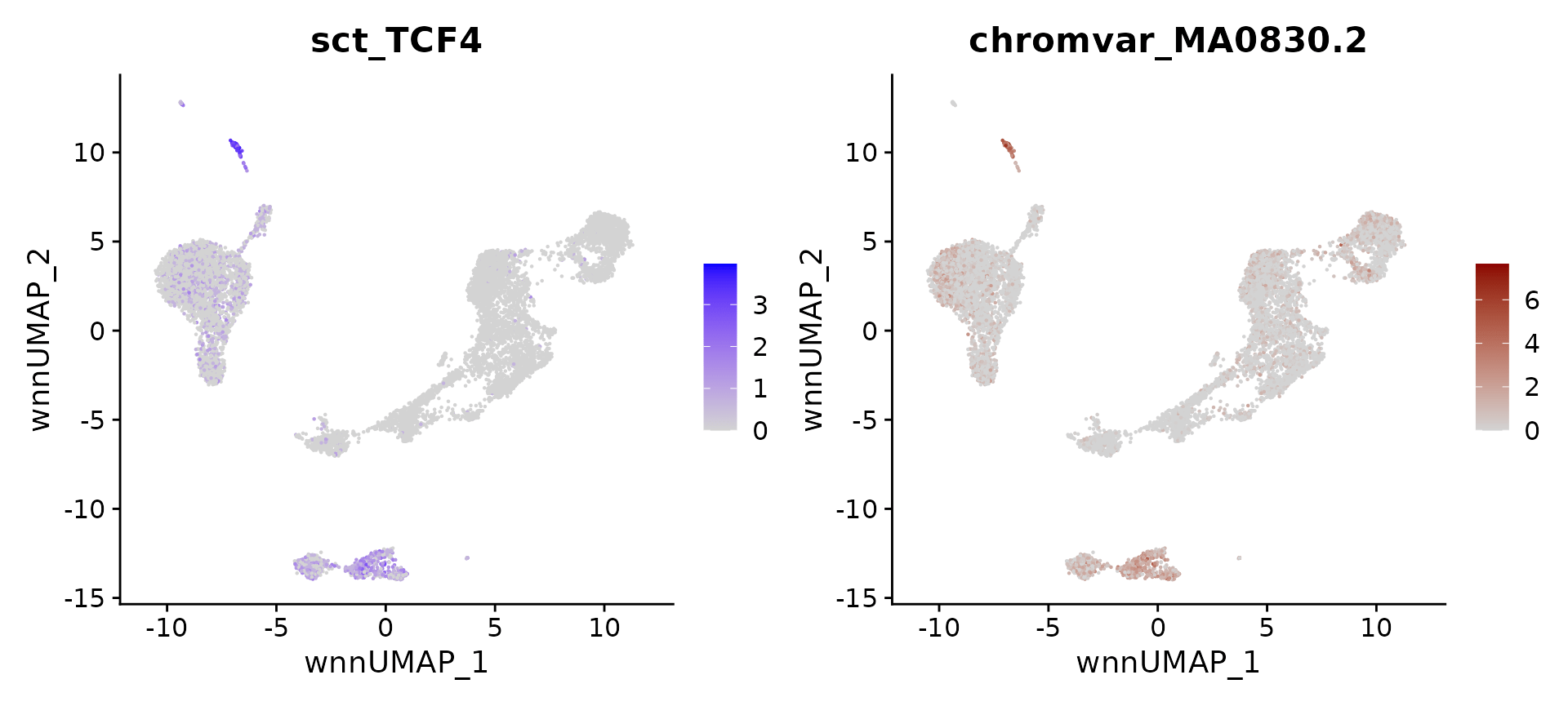
# identify top markers in pDC and visualize
head(topTFs("pDC"), 3)
## RNA.group gene RNA.auc RNA.pval motif.group motif.feature motif.auc
## 1 pDC TCF4 0.9998815 1.120711e-162 pDC MA0830.2 0.9965481
## 2 pDC IRF8 0.9904457 1.308164e-123 pDC MA0652.1 0.8833112
## 3 pDC SPIB 0.9115207 0.000000e+00 pDC MA0081.2 0.9068540
## motif.pval avg_auc
## 1 1.785897e-69 0.9982148
## 2 3.982413e-42 0.9368785
## 3 3.083509e-47 0.9091874
motif.name <- ConvertMotifID(pbmc, name = 'TCF4')
gene_plot <- FeaturePlot(pbmc, features = "sct_TCF4", reduction = 'wnn.umap')
motif_plot <- FeaturePlot(pbmc, features = motif.name, min.cutoff = 0, cols = c("lightgrey", "darkred"), reduction = 'wnn.umap')
gene_plot | motif_plot
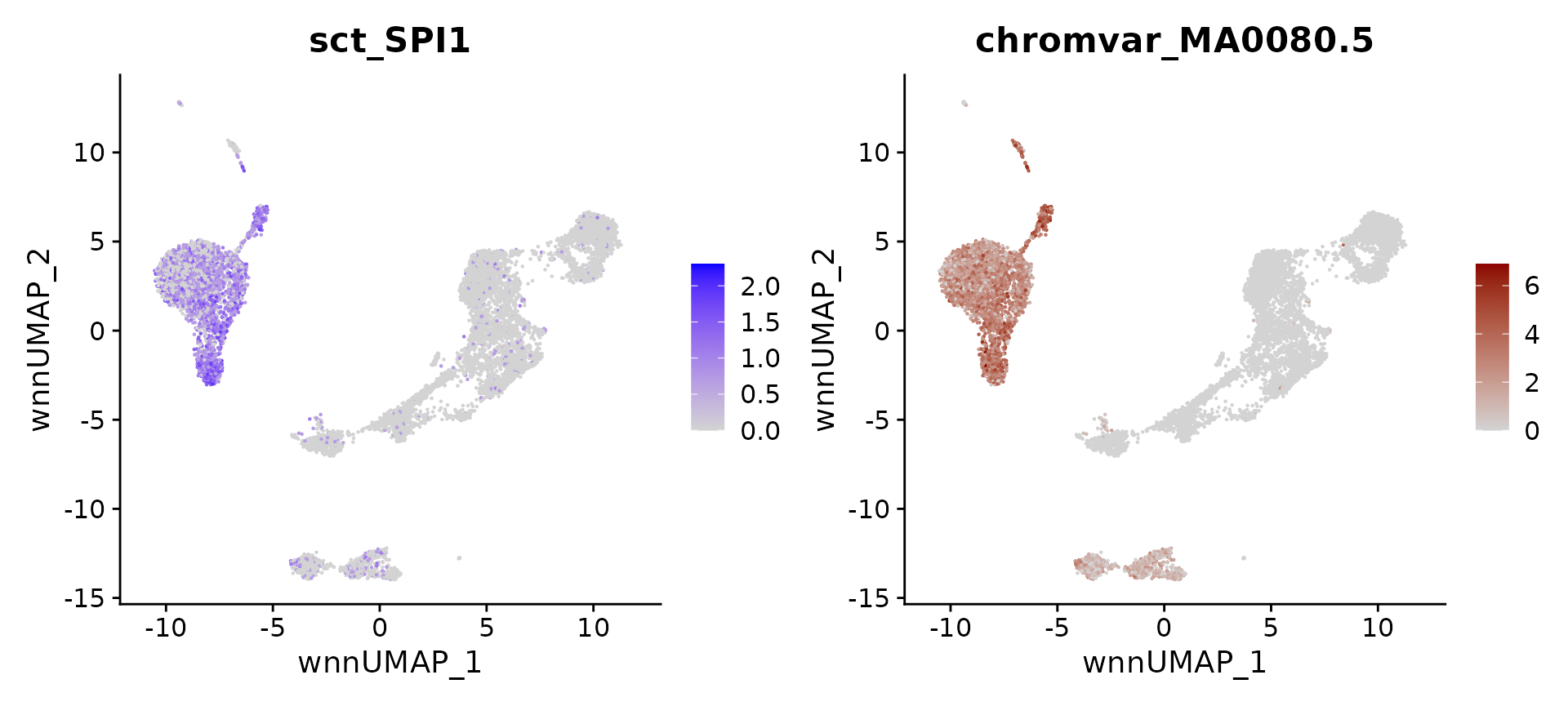
# identify top markers in HSPC and visualize
head(topTFs("CD16 Mono"),3)
motif.name <- ConvertMotifID(pbmc, name = 'SPI1')
gene_plot <- FeaturePlot(pbmc, features = "sct_SPI1", reduction = 'wnn.umap')
motif_plot <- FeaturePlot(pbmc, features = motif.name, min.cutoff = 0, cols = c("lightgrey", "darkred"), reduction = 'wnn.umap')
gene_plot | motif_plot
# identify top markers in other cell types
head(topTFs("Naive B"), 3)
## RNA.group gene RNA.auc RNA.pval motif.group motif.feature motif.auc
## 1 Naive B MEF2C 0.8802747 1.890065e-182 Naive B MA0497.1 0.6789079
## 2 Naive B PAX5 0.9436648 0.000000e+00 Naive B MA0014.3 0.5827061
## 3 Naive B POU2F2 0.6029377 2.892216e-13 Naive B MA0507.1 0.8912268
## motif.pval avg_auc
## 1 9.815118e-32 0.7795913
## 2 5.995185e-08 0.7631854
## 3 6.471677e-145 0.7470822
head(topTFs("HSPC"), 3)
## RNA.group gene RNA.auc RNA.pval motif.group motif.feature motif.auc
## 1 HSPC SOX4 0.9869406 5.524832e-71 HSPC MA0867.2 0.6830497
## 2 HSPC GATA2 0.7884615 0.000000e+00 HSPC MA0036.3 0.8275008
## 3 HSPC MEIS1 0.8638496 0.000000e+00 HSPC MA0498.2 0.6924225
## motif.pval avg_auc
## 1 1.241915e-03 0.8349951
## 2 7.591798e-09 0.8079812
## 3 6.877492e-04 0.7781361
head(topTFs("Plasma"), 3)
## RNA.group gene RNA.auc RNA.pval motif.group motif.feature motif.auc
## 1 Plasma IRF4 0.8189687 5.261955e-35 Plasma MA1419.1 0.9776046
## 2 Plasma MEF2C 0.9111213 2.977155e-12 Plasma MA0497.1 0.7596637
## 3 Plasma TCF4 0.8049409 6.810635e-12 Plasma MA0830.2 0.7840848
## motif.pval avg_auc
## 1 2.334627e-12 0.8982867
## 2 1.374353e-04 0.8353925
## 3 3.028306e-05 0.7945129

本文由 mdnice 多平台发布






















 686
686











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








