生信碱移
组织特异性富集分析
TissueEnrich基于Human Protein Atlas、GTEx和mouse ENCODE的多组织RNA‑seq数据,可以用于判断一组基因里是否富集了某一组织特异的基因
RNA测序技术的发展使得大规模比较不同发育阶段、细胞类型及条件下的基因表达成为可能。通过差异基因表达分析或共表达网络分析,研究者可以鉴定一系列的潜在关键基因用于功能分析。基因本体(GO)富集分析被广泛用于探究给定基因集合的功能,尽管GO分析能识别特定基因涉及的生物过程,但无法确定它们是否特异于某些组织 (组织特异性)。由于组织特异性基因更可能与人类疾病相关,故了解哪些基因群具有组织特异性具有重要价值。
为此,来自美国爱荷华州立大学的研究者提供了TissueEnrich工具,于2019年发表于Bioinformatic [IF:5.4]。TissueEnrich工具专门用于进行组织特异性基因富集分析,并被证实可为单细胞亚群和分化胚胎干细胞分配组织特征。

DOI: 10.1093/bioinformatics/bty890
简单来讲,TissueEnrich可以判断一组基因里是否富集了某一组织特异的基因,它基于Human Protein Atlas、GTEx和mouse ENCODE的多组织RNA‑seq数据,并在此基础上把基因分成三类:
-
Tissue‑Enriched(只在单一组织高表达)
-
Group‑Enriched(在 2 – 7 个组织一起高表达)
-
Tissue‑Enhanced(在单一组织高于其余组织平均值)
上述分好类的基因就是“参考库”。当用户把自己的基因列表丢进去时,TissueEnrich会对比参考库,用超几何检验算出每个组织的P值和富集倍数,并默认用Benjamini–Hochberg方法做多重检验校正。在此基础上,如果用户还提供了“背景基因集”,算法就只在这组背景里做统计。
本文简要介绍该工具使用,感兴趣的铁子可以进一步阅读以下内容:
-
https://www.bioconductor.org/packages/release/bioc/vignettes/TissueEnrich/inst/doc/TissueEnrich.html#teenrichment-tissue-specific-gene-enrichment-using-human-or-mouse-genes
执行组织特异性富集
①R包安装,TissueEnrich可以通过以下代码进行安装:
if (!require("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("TissueEnrich")
随后可以引用该R包:
library(TissueEnrich)
②准备数据,示例输入基因集inputGenes是一个R语言向量,根据基因名称进行准备:
inputGenes
# [1] "ADM" "ANXA1" "ATP10D" "BHLHE40" "C10orf54" "C2orf72" "C5orf30" "CAPN6" "CAV1" "CCR7" "CGA"
#[12] "CGB" "CGB5" "CGB8" "CLEC1A" "CPM" "CREB3L2" "CSF3R" "CYP19A1" "CYP2C18" "DLX5" "DLX6"
#[23] "ERBB3" "ERVFRD-1" "ERVV-1" "ERVV-2" "ESRRG" "FAM131A" "FAM49A" "FHDC1" "FST" "GAST" "GCM1"
#[34] "GNE" "GNG12" "GPR32" "GRAMD3" "GREM2" "HEXIM1" "HOPX" "HSPB8" "IFRD1" "IL1R1" "KATNBL1"
#[45] "KRT23" "KRT80" "LGALS16" "LURAP1L" "LYPD3" "MLLT11" "MRGPRX1" "MUC15" "MXD1" "MYO10" "MYO1E"
#[56] "NATD1" "NRP1" "NUCB2" "OVOL1" "PDLIM2" "PGC" "PGF" "PHKG2" "PHLDB2" "PHLDB3" "PID1"
#[67] "PKIA" "PLK2" "PRKCH" "PRR9" "PTPRE" "PVRL4" "QSOX1" "RHOBTB1" "S1PR2" "SDC1" "SERPINB9"
#[78] "SGK1" "SLC13A4" "SLC22A11" "SLC38A3" "SLC40A1" "SLCO4A1" "SMPDL3B" "SP6" "SPATA13" "ST8SIA4" "TGFBR2"
#[89] "TNFAIP3" "TREM1" "TREML2" "TRPV2" "WIPI1" "ZC2HC1C" "ZFAT" "ZNF277" "ZNF750"
③执行组织特异性富集分析,GeneSet函数中的物种除了人类Homo Sapiens以外还可以选择鼠Mus Musculus:
gs<-GeneSet(geneIds=inputGenes,organism="Homo Sapiens",geneIdType=SymbolIdentifier())
# 执行分析,小编实测小鼠时发现它的源码有点抽象,这里不多说,问题不大
output<-teEnrichment(inputGenes = gs, rnaSeqDataset = 1)
上述代码需要注意teEnrichment函数的rnaSeqDataset参数,其用于选择富集使用的参考数据集,可以设置为如下三种(默认为1):
-
1: Human Protein Atlas数据集(人) -
2: GTEx数据集(人) -
3: Mouse ENCODE数据集(鼠)
根据输出的output的第一个数据槽可以看看富集的结果:
seEnrichmentOutput<-output[[1]]
enrichmentOutput<-setNames(data.frame(assay(seEnrichmentOutput),row.names = rowData(seEnrichmentOutput)[,1]), colData(seEnrichmentOutput)[,1])
enrichmentOutput$Tissue<-row.names(enrichmentOutput)
head(enrichmentOutput)
# Log10PValue Tissue.Specific.Genes fold.change samples Tissue
#Adipose Tissue 0.0000000 1 1.2394054 5 Adipose Tissue
#Adrenal Gland 0.0000000 0 0.0000000 3 Adrenal Gland
#Appendix 1.0631035 4 4.4829558 3 Appendix
#Bone Marrow 0.5552268 4 2.7907142 4 Bone Marrow
#Breast 0.0000000 0 0.0000000 4 Breast
#Cerebral Cortex 0.0000000 3 0.4191623 3 Cerebral Cortex
结果解释:
-
Log10PValue:富集P值 -
Tissue.Specific.Genes:富集到的组织特异基因输入 -
fold.change:参考中的表达编号 -
Tissue:对应的组织
④针对这个结果表,首先可以可视化富集p值:
ggplot(enrichmentOutput,aes(x=reorder(Tissue,-Log10PValue),y=Log10PValue,label = Tissue.Specific.Genes,fill = Tissue))+
geom_bar(stat = 'identity')+
labs(x='', y = '-LOG10(P-Adjusted)')+
theme_bw()+
theme(legend.position="none")+
theme(plot.title = element_text(hjust = 0.5,size = 20),axis.title = element_text(size=15))+
theme(axis.text.x = element_text(angle = 45, vjust = 1, hjust = 1),panel.grid.major= element_blank(),panel.grid.minor = element_blank())
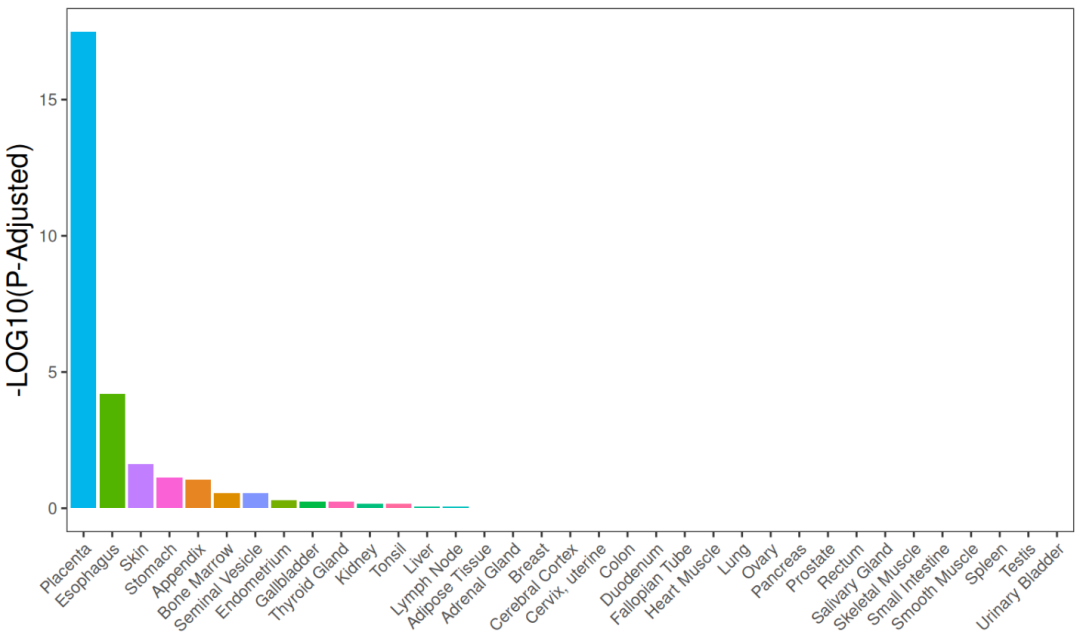
然后是富集数量(其实可以画在同一张表中):
ggplot(enrichmentOutput,aes(x=reorder(Tissue,-fold.change),y=fold.change,label = Tissue.Specific.Genes,fill = Tissue))+
geom_bar(stat = 'identity')+
labs(x='', y = 'Fold change')+
theme_bw()+
theme(legend.position="none")+
theme(plot.title = element_text(hjust = 0.5,size = 20),axis.title = element_text(size=15))+
theme(axis.text.x = element_text(angle = 45, vjust = 1, hjust = 1),panel.grid.major= element_blank(),panel.grid.minor = element_blank())
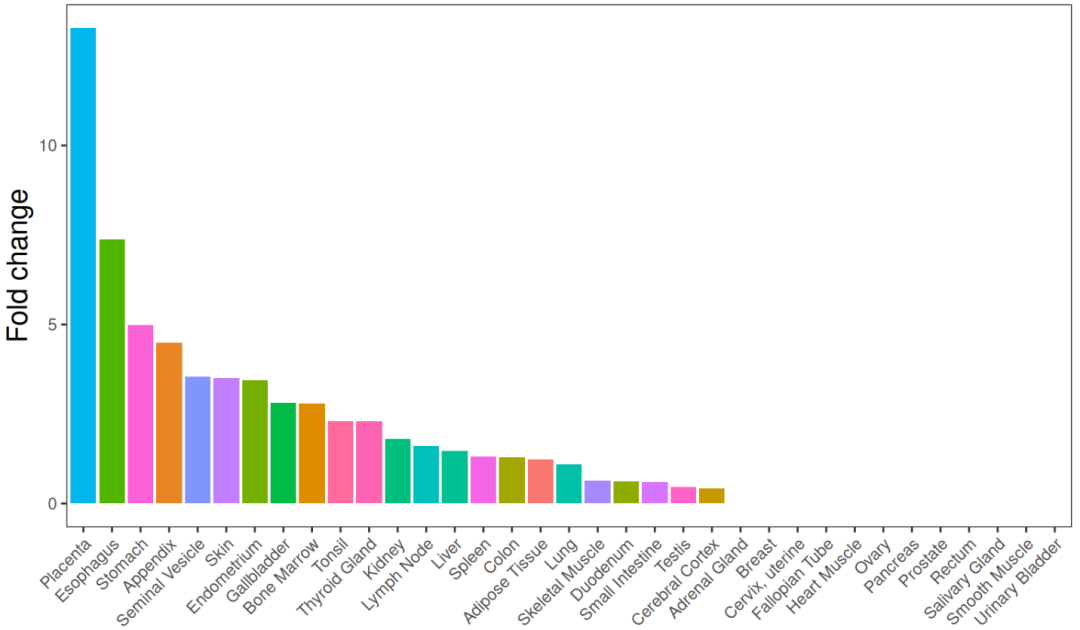
⑤提取指定组织的富集结果,并可视化RNA数据集中的表达:
library(tidyr)
seExp<-output[[2]][["Placenta"]] # 选择Placenta组织
exp<-setNames(data.frame(assay(seExp), row.names = rowData(seExp)[,1]), colData(seExp)[,1])
exp$Gene<-row.names(exp)
exp<-exp %>% gather(key = "Tissue", value = "expression",1:(ncol(exp)-1))
ggplot(exp, aes(Tissue, Gene)) + geom_tile(aes(fill = expression),
colour = "white") + scale_fill_gradient(low = "white",
high = "steelblue")+
labs(x='', y = '')+
theme_bw()+
guides(fill = guide_legend(title = "Log2(TPM)"))+
#theme(legend.position="none")+
theme(plot.title = element_text(hjust = 0.5,size = 20),axis.title = element_text(size=15))+
theme(axis.text.x = element_text(angle = 45, vjust = 1, hjust = 1),panel.grid.major= element_blank(),panel.grid.minor = element_blank())
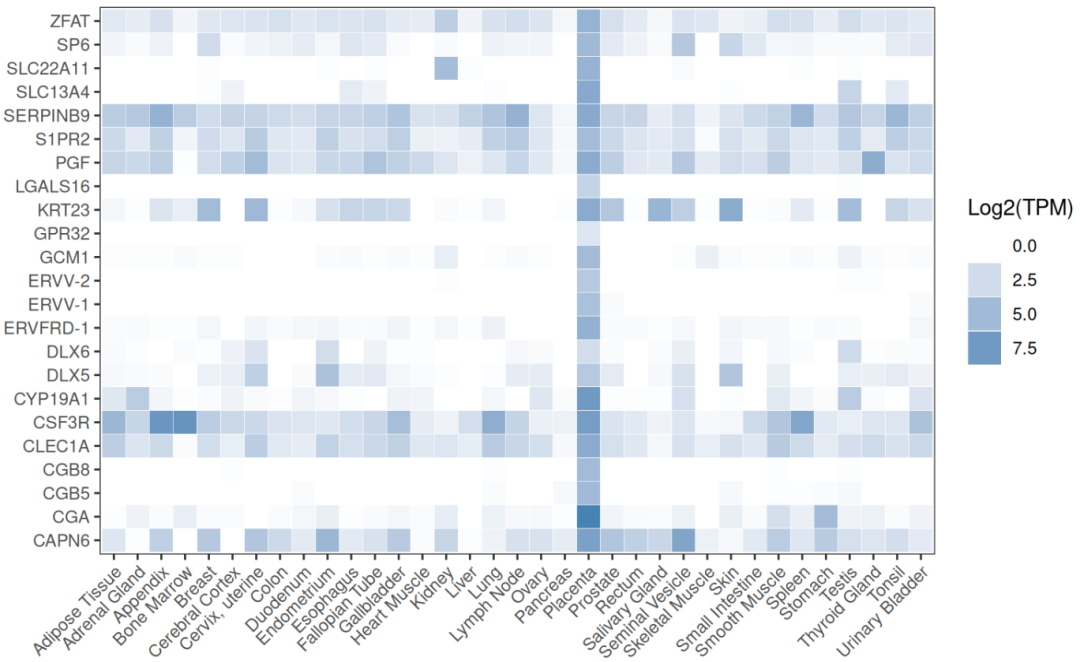
后续可以对接gganatogram可视化
近期在忙一些事情
影响因子更新期刊表也得整理
忙完这段时间增加更新频率




















 1780
1780

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








