hello,大家好,今天我们来分享一个新的空间注释的方法,当然主要思想还是解卷积,关于空间转录组的注释方法,我已经分享了很多了,在这里列举出来,供大家参考。
MIA用于单细胞和空间的联合分析
10X单细胞和空间联合分析的方法---cell2location
10X空间转录组和10X单细胞数据联合分析方法汇总
10X单细胞空间联合分析之四----DSTG
10X单细胞空间联合分析之三----Spotlight
10X单细胞空间联合分析之五----spatialDWLS
10X单细胞空间联合分析之六(依据每个spot的细胞数量进行单细胞空间联合分析)----Tangram
10X单细胞-10X空间转录组联合分析之七----CellDART
当然还有依据marker注释空间转录组的方法,10X空间转录组数据分析之思路总结(针对肿瘤样本)
10X单细胞-10X空间转录组联合分析之八----STRIDE(三维重构)
当然了,方法很多,不见得都是适合自己的样本分析,每种方法都有其优势和不足,我们要根据自己的实际情况进行选择。好了,开始我们今天的分享,文章在Reference-free cell-type deconvolution of pixel-resolution spatially resolved transcriptomics data,用到的方法是STdeconvolve,下面放几张分析效果图。



当然了,空间转录组可分析的内容是在太多了,如果大家有兴趣,可以在文章的下面评论一下,如果人员比较多的话,我就给大家讲一节公开课,大家一起交流交流,好了,开始我们今天的内容分享。
Abstract
最近的技术进步使空间解析转录组分析成为可能,但在多细胞像素分辨率下,从而阻碍了细胞类型空间共定位模式的识别。 作者开发了 STdeconvolve 作为一种无监督方法来对包含这种多细胞像素分辨率空间分辨转录组学数据集的底层细胞类型进行去卷积。 实际运用表明 STdeconvolve 有效地恢复了细胞类型的假定转录组学特征及其在空间分辨像素内的比例表示,而无需依赖外部单细胞转录组学参考(如果真的不以来单细胞数据,那真的是很完美,我们往下看看)。
Main
描绘组织内转录不同细胞类型的空间组织对于理解组织功能的细胞基础至关重要。最近的技术已经能够以多细胞像素分辨率在组织内进行空间分辨转录组 (ST) 分析。因此,这些 ST 测量代表可能包含多种细胞类型的细胞混合物。这种缺乏单细胞分辨率阻碍了细胞类型特定空间组织的表征。为了应对这一挑战,最近开发了 SPOTlight 和 RCTD 等监督解卷积方法来预测 ST 像素内细胞类型的比例。然而,这些有监督的反卷积方法依赖于合适的单细胞参考的可用性,如果由于预算、技术或生物学限制而没有这样的参考,则可能会出现限制。在这里,作者开发了 STdeconvolve作为一种用于解卷积多细胞像素分辨率 ST 数据的无监督、无参考的分析方法(下图)。


给定 ST 数据的计数矩阵,STdeconvolve 推断不同细胞类型的推定转录组特征及其在每个多细胞空间分辨 ST 像素内的比例表示。 简而言之,STdeconvolve 的第一个特征选择可能提供转录不同细胞类型信息的基因。 STdeconvolve 然后基于Latent Dirichlet Allocation (LDA) 来估计转录不同细胞类型 K 的数量,并在 ST 像素中对这些 K 细胞类型进行解卷积。 STdeconvolve 利用 ST 数据的几个独特功能,使 LDA 的此应用程序特别适合。
首先评估了 STdeconvolve 在使用模拟 ST 数据恢复细胞类型及其转录组学特征的比例表示方面的性能。 这是通过将小鼠内侧视前区 (MPOA) 的单细胞分辨率多重错误鲁棒荧光原位杂交 (MERFISH) 数据聚合到 100 μm2 像素中来完成的。

- 注:Ground truth single-cell resolution MERFISH data of one section of the MPOA partitioned into 100 μm2 pixels (black dashed squares). Each dot is a single cell colored by its ground truth cell-type label.
应用 STdeconvolve,确定了 K=9 转录不同的细胞类型,并在每个模拟像素中对它们的转录组谱和比例表示进行了反卷积(下图)。
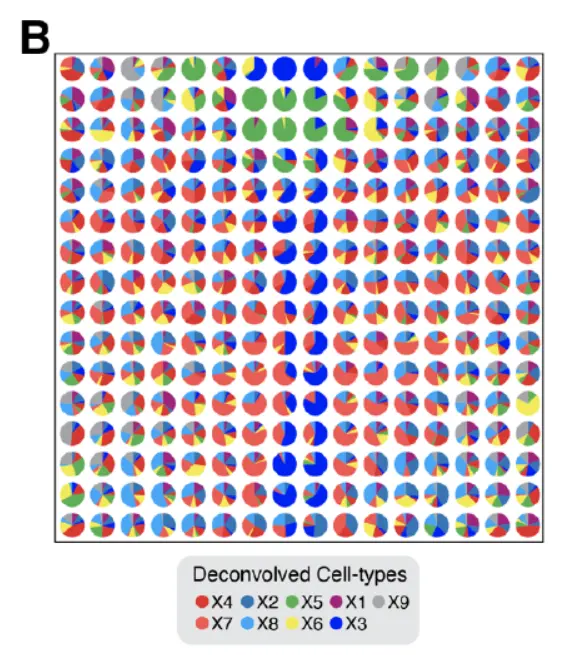
- 注:B. Proportions of deconvolved cell-types from STdeconvolve, represented as pie charts for each simulated pixel。
为了推断去卷积细胞类型的身份以进行基准测试,将它们的去卷积转录谱与真实细胞类型的转录谱进行了匹配(下图)。

- 注:(B)Pearson’s correlation between the transcriptional profiles of the 9 ground truth cell-types in the MERFISH MPOA data and the 9 deconvolved cell-types by STdeconvolve.(C). Distributions of Pearson’s correlations between the transcriptional profiles or pixel proportions of matched deconvolved and ground truth cell-types. Matched cell-types are indicated as highlighted boxes in B.
观察到每个去卷积细胞类型的转录组谱与跨基因匹配的真实细胞类型之间的强相关性,同样地,在模拟像素中每个去卷积细胞类型和匹配的真实细胞类型的比例之间存在强相关性。

- 注:(C)The ranking of each gene based on its expression level in the transcriptional profiles of the deconvolved cell-types, compared to its gene rank in the transcriptional profile of the matched ground truth cell-type. (D). Heatmap of Pearson’s correlations between the proportions of the deconvolved cell-types and ground truth cell-types across simulated pixels. Ground truth cell-types are ordered by their frequencies in the ground truth dataset. Matched deconvolved and ground truth cell-types are boxed.
一些去卷积的细胞类型,如细胞类型 X2 和 X8 都与兴奋性神经元匹配,而细胞类型 X4 和 X7 都与抑制性神经元匹配。 根据先前的注释,将真实的兴奋性和抑制性细胞类型进一步划分为其他亚型(共 76 种),发现这些去卷积的细胞类型与神经元亚型的特定组合相关

当进一步将去卷积细胞类型的数量扩大到 K=76 时,能够识别在转录谱和像素比例与更精细的神经元亚型以及稀有细胞类型方面高度相关的去卷积细胞类型such as pericytes and microglia。此外,由于当前的 ST 技术允许以不同的分辨率进行空间转录组分析,因此进一步模拟了另一个 20 μm2 分辨率的 ST 数据集,并观察到解卷积的细胞类型转录组谱与 STdeconvolve 的基本事实的比例之间类似的强相关性(下图).

- 注:Comparison of STdeconvolve cell-types to ground truth cell-types of a 20 µm2 197 pixel MERFISH MPOA ST dataset。








 本文介绍了无监督的STdeconvolve方法,用于空间转录组数据中细胞类型的解卷积,无需依赖单细胞转录组学参考。该方法基于Latent Dirichlet Allocation (LDA),在模拟和实际数据中表现出与监督方法相当的性能,适用于多细胞像素分辨率的数据分析,有助于理解组织中细胞类型的空间组织。
本文介绍了无监督的STdeconvolve方法,用于空间转录组数据中细胞类型的解卷积,无需依赖单细胞转录组学参考。该方法基于Latent Dirichlet Allocation (LDA),在模拟和实际数据中表现出与监督方法相当的性能,适用于多细胞像素分辨率的数据分析,有助于理解组织中细胞类型的空间组织。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 5149
5149

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








