参考我的上一篇博客<https://blog.csdn.net/weixin_62528784/article/details/142092974?spm=1001.2014.3001.5501>,
安装了hic-pro并进行了初步的数据处理,接下来是对处理得到的数据进行一些基本的上游分析,并不限制于文章中所提到的模块,待之后阅读CNS文献进行拓展;
hic-pro输出结果解读:
1,首先要理解hic-pro的处理流程:
那就要理解下面这张图:
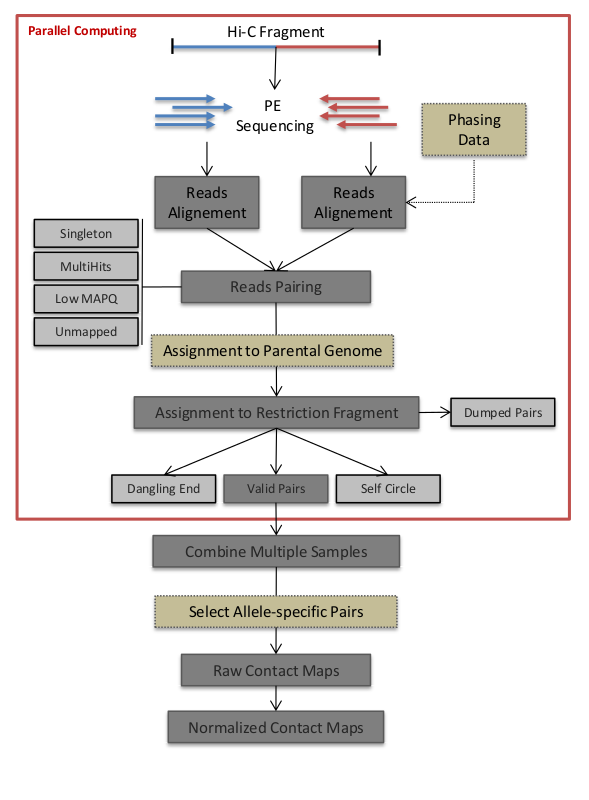
主要参考官方<https://github.com/nservant/HiC-Pro/blob/master/doc/MANUAL.md#how-does-hic-pro-work->
2,解读输出的文件结果:

在hic-pro -o 输出的文件夹里一共5个子文件夹,


其中比较重要的文件是hic_results目录中结果文件:

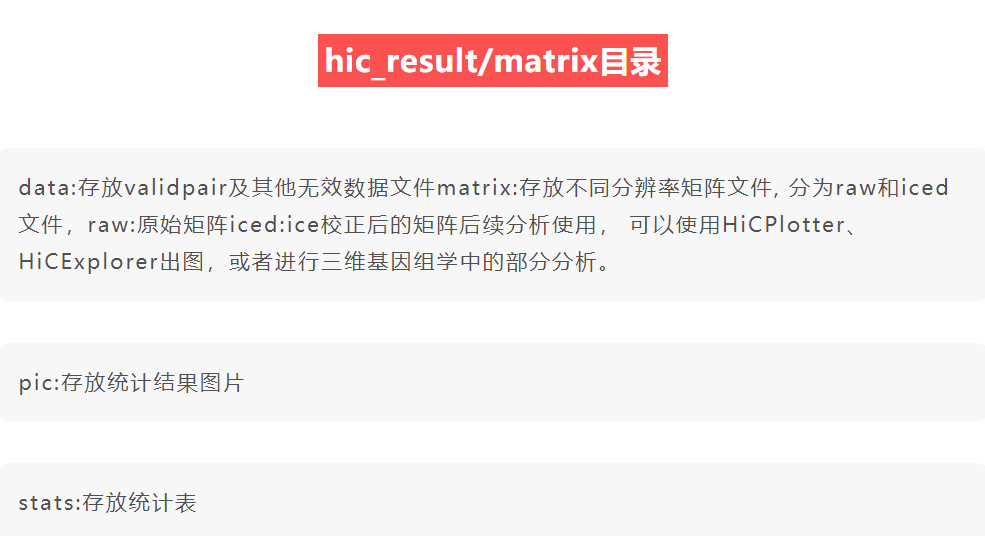

因为hic_results的文件比较重要,下面进行解读:
(1)整个输出中最重要的文件就是data文件夹中的 ** *.allValidPairs文件**,以及matrix文件夹中(对应不同分辨率的iced/raw不同处理)的bed文件+matrix文件,
①.allValidPairs文件:
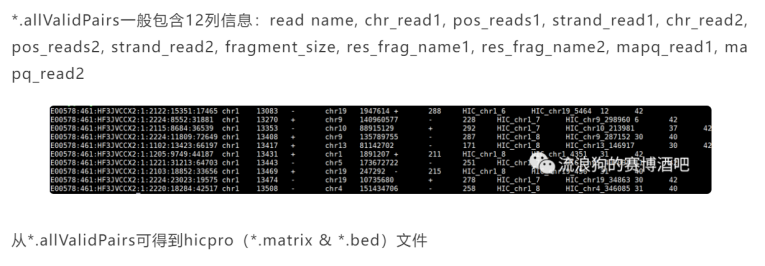

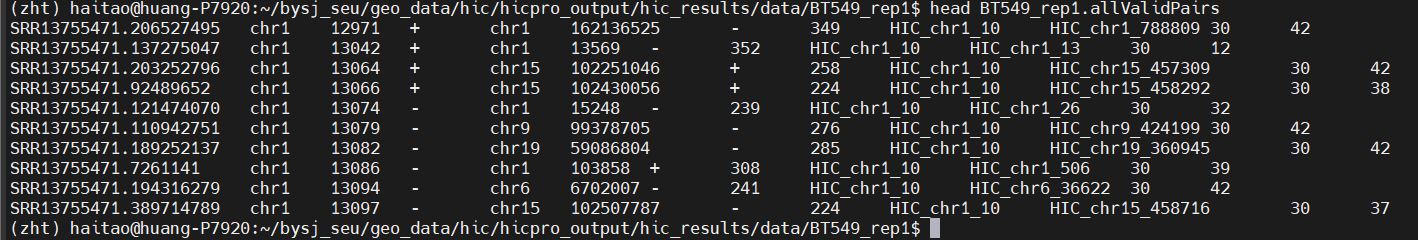
可以很直观地看出每一行基本上都是互作pair的信息:
后面在计算区室compartment如果使用homer的时候只用该文件中的前6列;
allVaildPairs:合并后的Valid pairs数据,数据上validpairs比allvalidpairs大,但是还是使用合并之后的数据进行后续处理
②bed文件:bed文件就只在raw文件夹中有,与指定分辨率相关的基因组间隔列表
储存着bin的位置信息
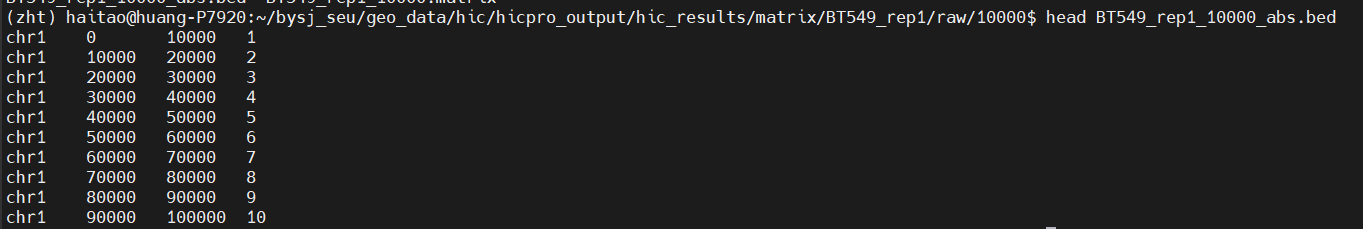
以上图为例子就是10kb的bin,应该就是对分区出来的bin进行一个编号(第三列),然后这个编号在对应的matrix文件中(对称的,无论是上下三角还是稀疏矩阵),记录对应的contact的时候使用的编号就是此处文件中标记的编号;
③matrix文件:.matrix储存互作count数量
Raw contact maps are in the raw folder and normalized contact maps in the iced folder. ICE是校正之后的,matrix文件在raw以及iced文件夹中都有,

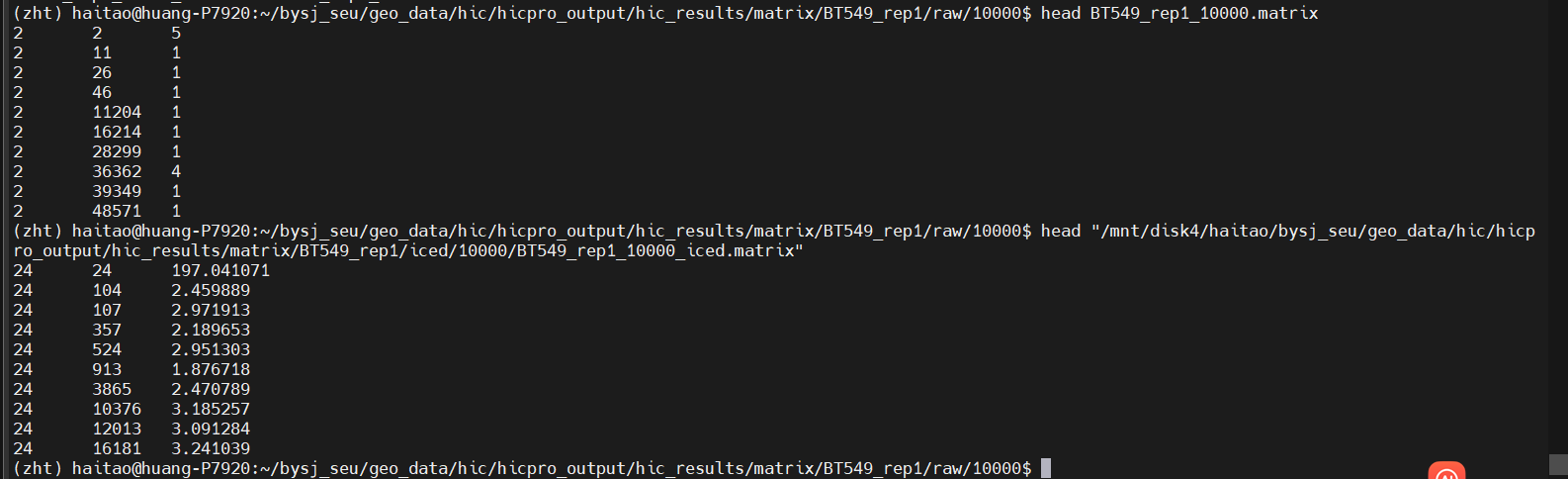 是一个3列的数据,前两列使用的就是bed文件中分区的编号,
是一个3列的数据,前两列使用的就是bed文件中分区的编号,
第三列就是互作contact的count,以及对应的ice标准化后的
(2)hic-pro质控结果解读:
参考<https://nservant.github.io/HiC-Pro/RESULTS.html >, 主要解读hic_results/pic/文件夹中的质控pdf图,
主要是查看pic文件夹中的这5图pdf:
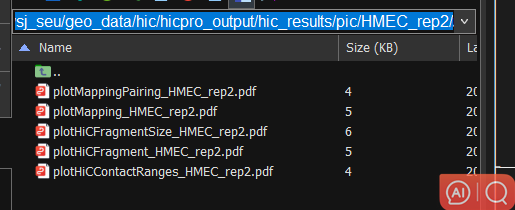
对照该文档的解读,输出结果中的pic中的5幅pdf图基本上没有问题,
①plotMapping_图:mapping步骤,单端比对过滤结果图
read回帖统计信息表示为回帖上的R1/R2 reads比例的条形图,
每一幅图中有两个条形图,左条形图表示整体的回帖比例,右条形图区分两个回帖步骤;右条形图就是拆分了左条形图。
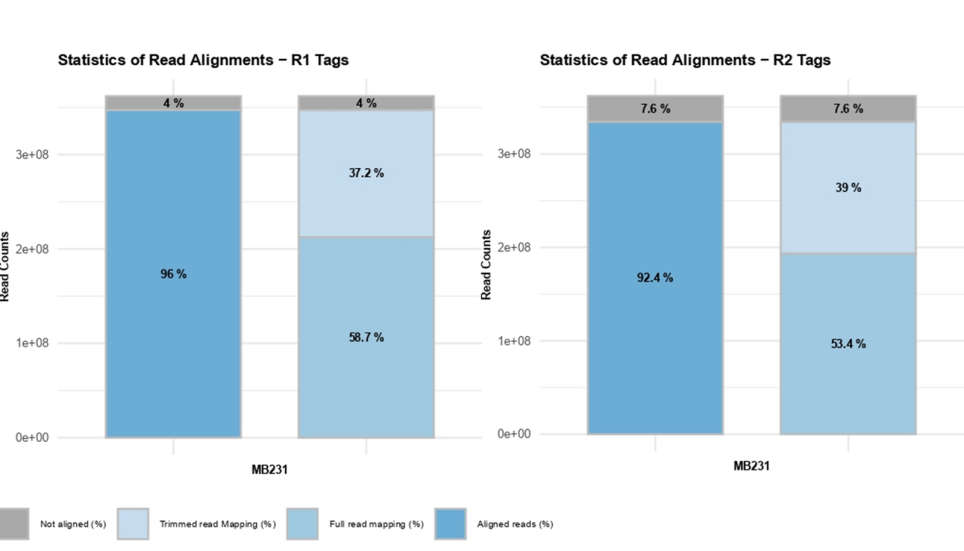

所谓的两步回帖:

官方文档中将左条形图中aligned reads拆分成trimmed reads mapping(step2)+full reads mapping(step1);
一般是aligned reads比例在80~90%左右(即左条形图中),step2即trimmed reads mappping比例在10%左右,然后R1/R2都这么看;
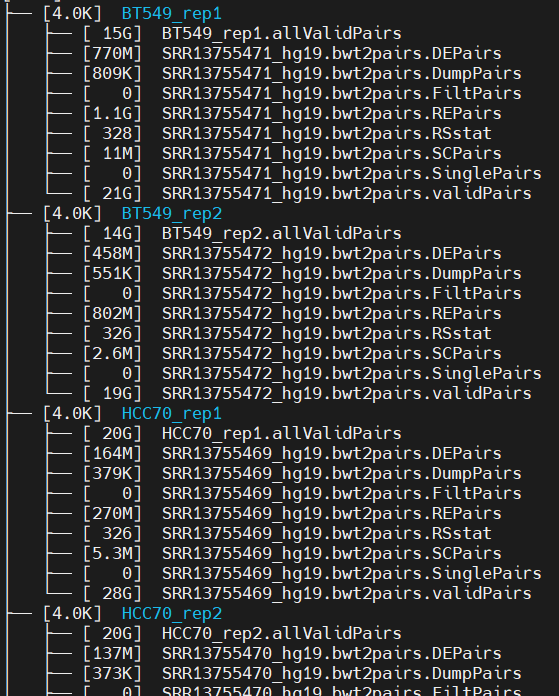
以下是我本人曾经做过的一个项目的质控图,图片见本人项目,暂列出结果对照:
按照颜色由深入浅看,由左向右看
MB231:全体mapping 95左右合格,但是trim在40
BT549rep1:全体95左右合格,trim在50左右
rep2:全体95左右合格,trim在50左右
HCC70rep1:全体95左右合格,trim在60左右
rep2:全体95左右合格,trim在55左右
HMECrep1:全体95左右合格,trim在55左右
rep2:全体95左右合格,trim在50左右
问题1:查看7个rep之后发现step2基本上都是在50%左右,异常,这个数据要不要用?
查看github中的其他issue报告来看,也有将近30的,以及秉持着公共数据库的存在即合理-已经发表。
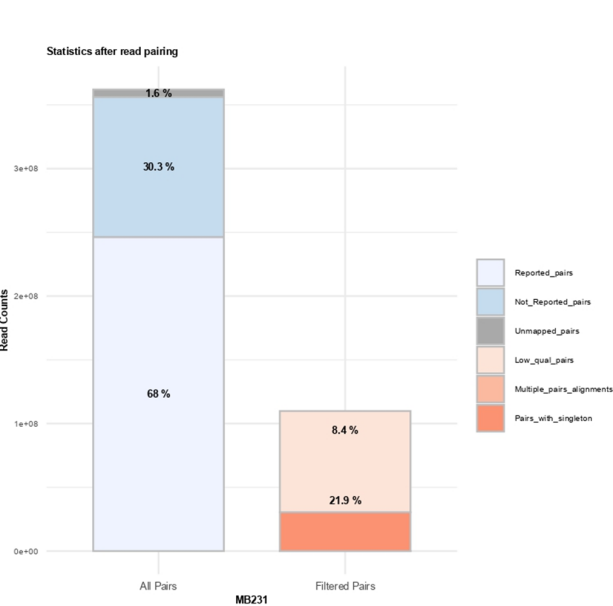
②plotMappingPairing:pairing步骤,合并后双端比对过滤结果图
主要是关注singleton or multi-hits的比例,singleton的比例一般是接近于未回帖的R1+R2的比例,
但是从对应的pdf图片中看不出什么信息,
The fraction of singleton is usually close to the sum of unmapped R1 and R2 reads,
7个rep都差不多(本人是项目),
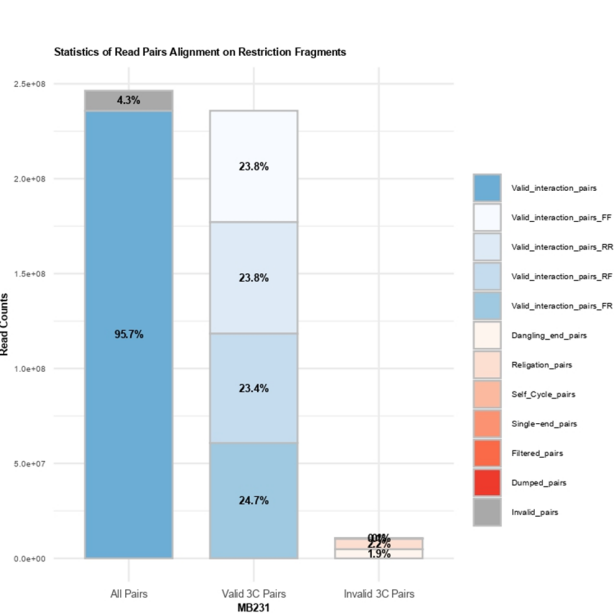
③ plotHiCFragment_限制酶片段回帖信息统计:有效数据过滤结果图
主要是第二个条形图:在有效的Valid 3C Pairs中4类预期是各个25%左右(至少是4个差不多比例),
在第三个条形图中dangling-end or self-circle read pairs的比例要很低,
7个rep都正常(依然是本人项目),
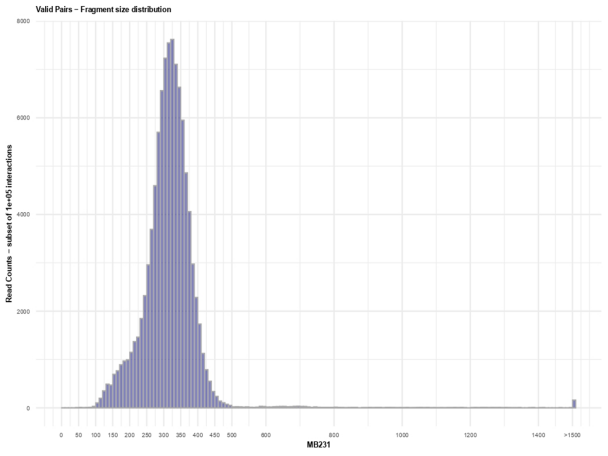
④plotHicFragmentSize_:从valid interaction中提取的片段大小分布,有效互作的片段大小分布图
预期以300pb为中心的分布,7个rep都正常
⑤plotHiCContactRanges_:有效互作中各类型比例图
第一个条形图中duplicates的比例要低,
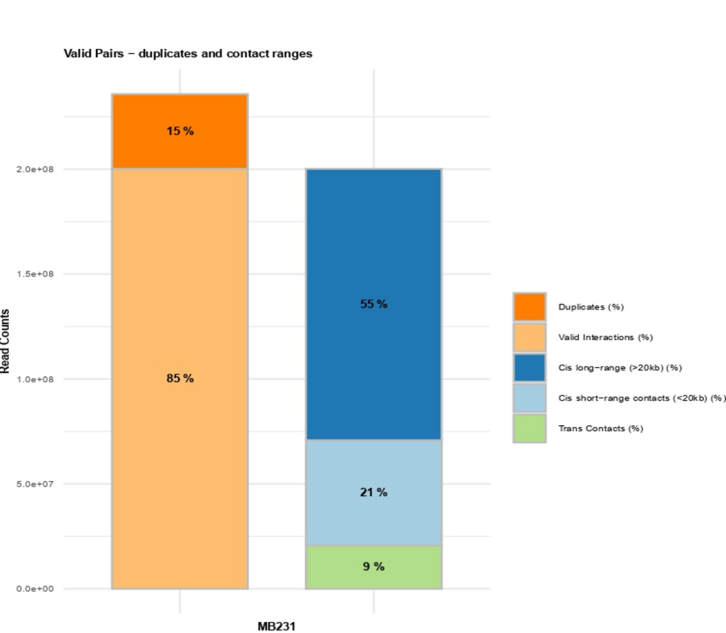
这对应的5个图表的信息就可以作为hic数据异质性的展示(可以将对应的数据进行重绘制),
hic数据格式转换:
hic-pro输出数据如何转化为其他数据,主流的hic软件处理格式有.hic、.allvalidpairs、.cool、.mcool、.h5等数据格式,而下游因为数据分析需求,hic-pro本身的输出数据格式不足以完成大部分分析,所以需要将hic-pro的输出数据格式转换为其他的数据格式;
可以参考<https://mp.weixin.qq.com/s/GNAvhT2rIkePqzTFPVHr6g>,
1,使用hic-pro官方维护以及开发的对接脚本进行数据格式转换:
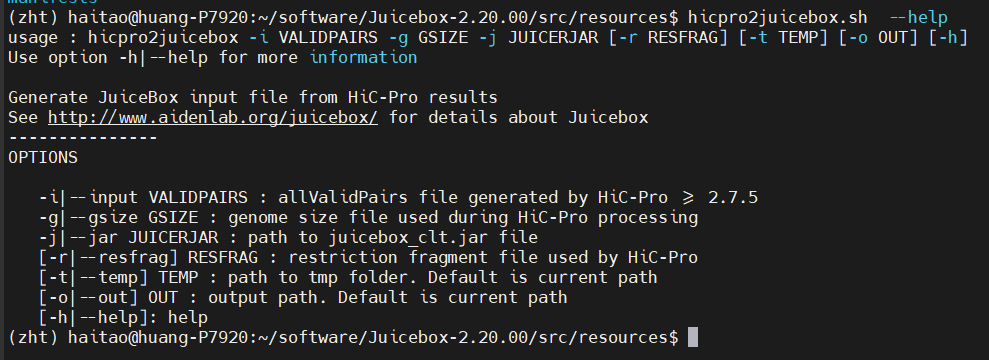
(1)转化为.hic文件:使用juicebox/juicer+hic-pro的脚本hicpro2juicebox的shell脚本,
参考<https://nservant.github.io/HiC-Pro/UTILS.html#hicpro2juicebox-sh> ,
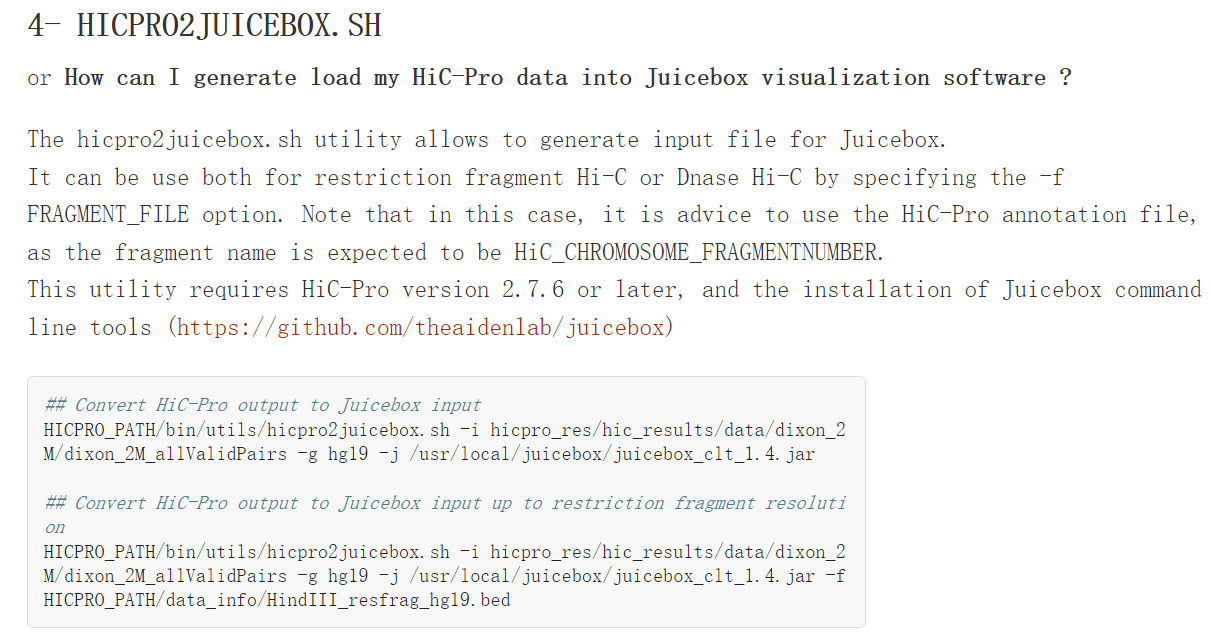
需要两个工具,一个是hic-pro的内置函数程序,一个是juicebox的java程序;
首先是hic-pro的内置转换函数:
至于juicebox的这个java程序,进入 下面网站:

实际上对应的release却是juicer_tools文件,最新的juicer_tools文件是2.2.0,
参考<https://github.com/aidenlab/Juicebox/releases>,
后来参考issue以及询问开发者:<https://github.com/nservant/HiC-Pro/issues/610>以及<https://github.com/nservant/HiC-Pro/issues/446>,应该下载juicer_tools,
当然在juicer系工具开发者网站中,有其他的juicer工具,曾经也下载了不少:
理论上讲,至于具体用哪一个jar文件,应该不会太影响,
之前使用juicer_tools最新版2.2.0,但是生成的.hic文件在后续cool文件处理上有问题,所以还是用到v1版本,即使用juicer_1.22版本,所以目前所有生成的hic文件基本上都是1.22版本生成的;
总之,v2目前处于开发中,如果有需求自然下载最新版2.2.0,如果后面分析报错排除了之后需要换v1,那么可以参考juicer中的hic-pro issue,或者是hic-pro中的juicer issue,基本上是推荐使用v1的某个版本,而我本人就选择了juicer_1.22版本。
正式开始操作:
注意以linux中的help的参数为准,github中hic-pro的指南参数不准确,
注意<https://github.com/nservant/HiC-Pro/blob/master/doc/UTILS.md>和<https://nservant.github.io/HiC-Pro/UTILS.html#hicpro2juicebox-sh>中的参数命令设置有点差异,相互比较之后,-g部分使用size文件的全路径,下面的命令中修改:
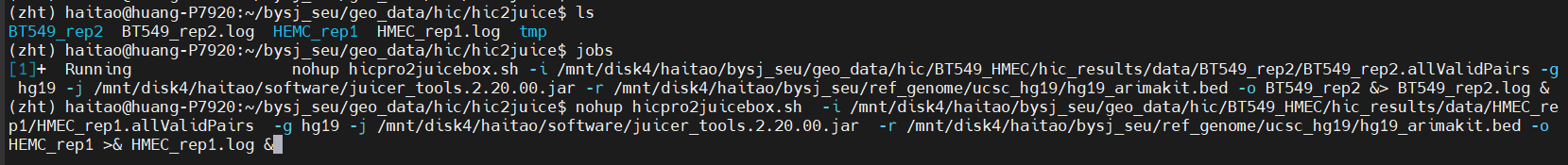

-i 就是allvalidpairs文件
-g 这个参数的写法有点问题,指南里既有hg19这样简单的指示,也有提供size文件路径的指示,但是我很担心这样表示是否能够保证识别到前期特意处理过的size文件,所以我本人建议使用size文件的全路径!
-j hic-pro配置时候获取的3个注释文件之一,就是限制酶的酶切bed文件
-o 注意先建立-o的目标文件夹,不然会报错!!!
(2)hic-pro官方和开发者们也开发出了其他下游软件分析格式所需的对接脚本,用于将hic-pro的输出转换为其他数据格式,具体参考<https://nservant.github.io/HiC-Pro/UTILS.html>,
2,除了使用hic-pro官方开发的脚本来善后hic-pro输出数据格式,也可以使用其他数据转换工具:
比较有名的是hicexplorer(<https://hicexplorer.readthedocs.io/en/latest/content/tools/hicConvertFormat.html>),
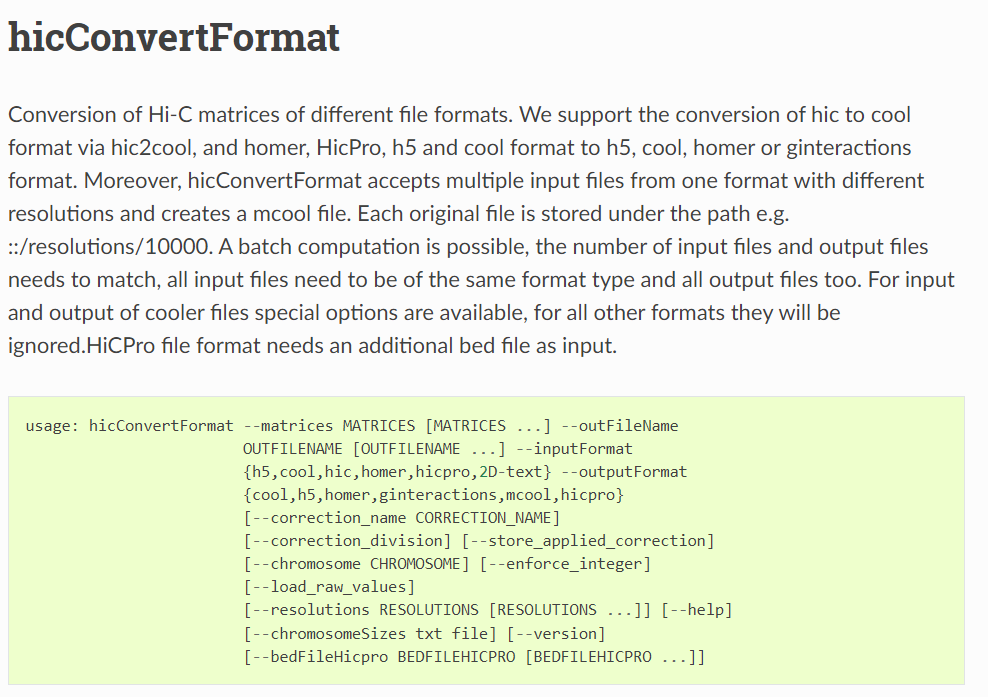
这里假设我们的上游数据格式依然是hic-pro的原始输出,而非.hic等一次转换数据格式,
下游常用的数据格式是cool格式(mcool实际上是包含多分辨率的cool文件集合),
(1)hicpro格式到cool格式:
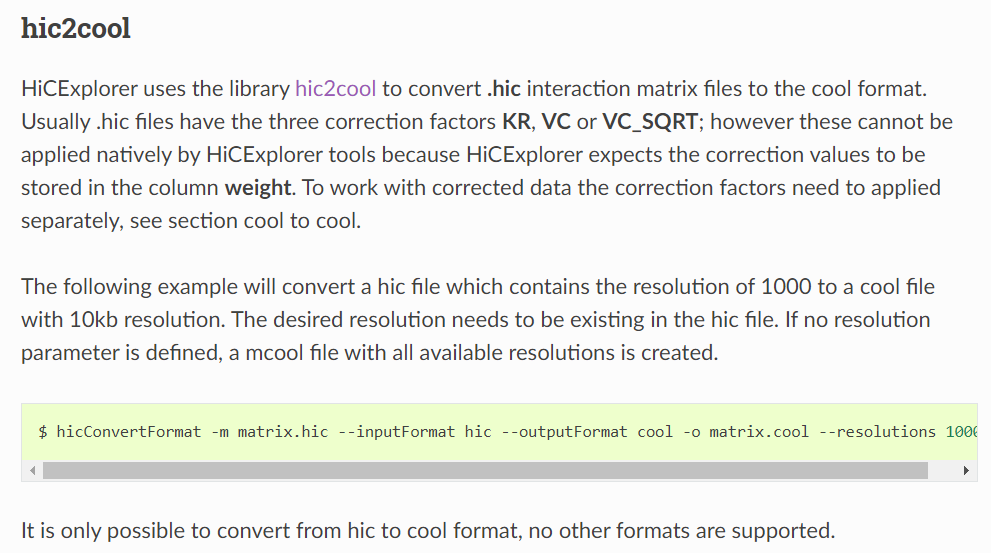
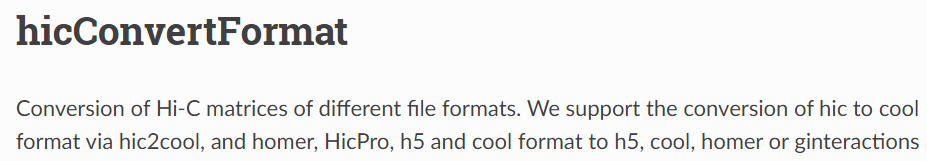
可以发现hicexplorer将hic转换到cool格式,实际使用的也是hic2cool工具,所以不用考虑hic2cool工具和这个工具哪个好的问题,就这一个工具一条龙使用,
下面贴一点我本人使用hic2cool时候遇到过的问题记录:
参考<https://github.com/4dn-dcic/hic2cool>以及<https://github.com/4dn-dcic/hic2cool/issues/57>,
以及<https://github.com/4dn-dcic/hic2cool/issues/56>(就是这个问题里报错,然后我换了上游的hicpro2juicebox函数中所需的juicer tools,改成了v1,所以注意:可能v2版本也没有问题,用v2版本juicer处理的hic-pro到hic格式也许能够正常进行下游分析只不过我在hic2cool的黑匣子中搞错了什么。总之保险起见,我本人避免使用上游v2 juicer处理hic-pro到hic+下游使用hic2cool,因为hicexplorer中用的也是hic2cool模块,所以上游统一改成了v1!!!)
正式开始hicpro到cool格式,
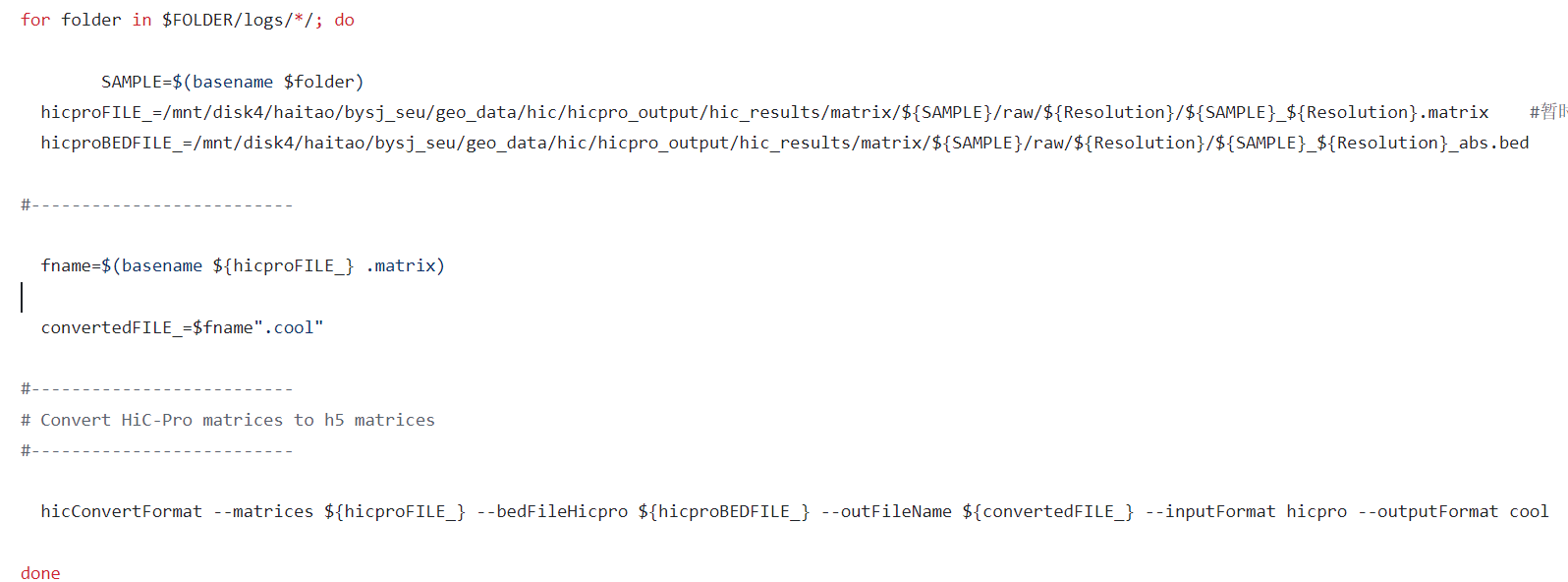
实际上如果使用hicpro的输出数据的话,只能使用matrix+bed数据,暂时没有看到allvalidpairs数据如何使用的例子(也许可以去issue看看?),
那问题来了, bed文件好说,毕竟在hic-pro的输出文件夹中只有raw文件夹有,那matrix呢?是使用iced还是raw?
这个问题需要到hicpro issue中搜hicexplorer的转换函数,或者到hicexplorer中搜hicpro转换问题;
所以以下仅仅是我个人的看法:
参考<https://github.com/deeptools/HiCExplorer/issues/786>

matrix和bed文件实际上前缀一致的话都是raw,只有iced中的matrix才会显示_iced.matrix,但是这能说明什么呢,一个人选用了raw,那也可能是他用错了。
参考<https://github.com/deeptools/HiCExplorer/issues/753> ,

好,现在是2个人使用raw了,
<https://github.com/deeptools/HiCExplorer/issues/752>
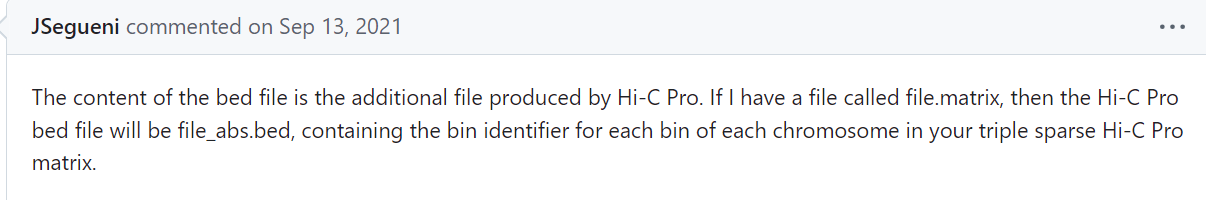
<https://github.com/deeptools/HiCExplorer/issues/707>,总之我可以从issue中找到很多用法,看用的最多的人是哪种用法(如果有权威文献告诉我更好),总之本人matrix+bed都使用raw文件夹中的!
再加上我看到某人用iced报错了:
<https://github.com/deeptools/HiCExplorer/issues/809>
经过上面的操作,hicpro格式终于转换到了cool格式,但是我本人下游分析的时候用的mcool格式更多,所以1个1个零散分辨率的cool格式也许并不是终点,需要进一步合并!
经过本人尝试,hicexplorer不能直接将hicpro格式转换为mcool,所以需要中间先转换为cool格式,再合并为mcool(下图2)。
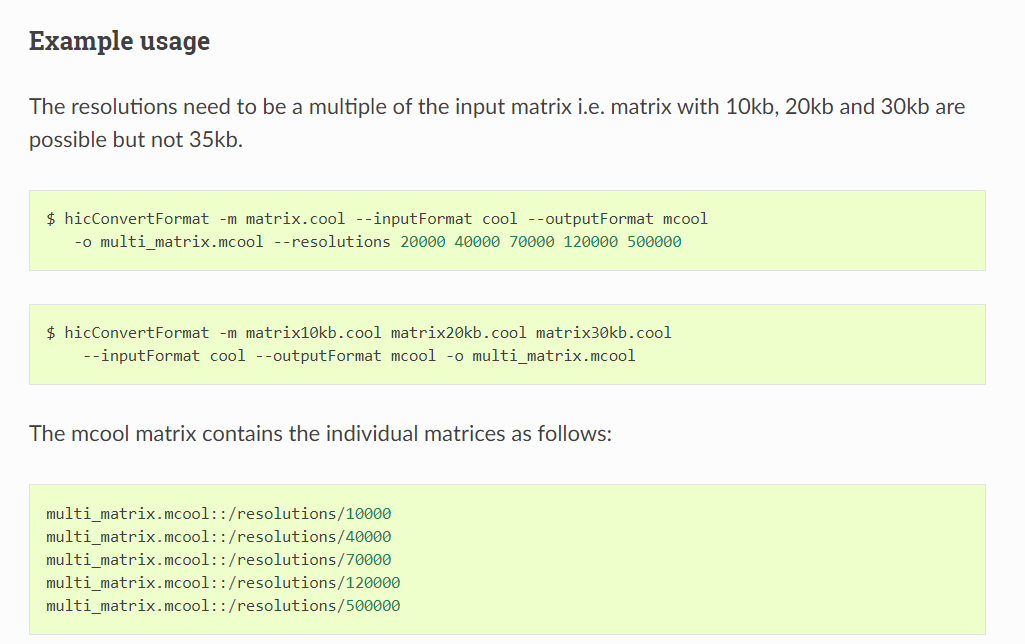
但是这里又有另外一个问题,如果仅仅是1个文件处理的话,那当然可以使用转换函数换来换去,因为1个数据单个数据没有比较的标准,但是我们做项目数据分析的时候经常会遇到多个样本比较多个样本处理的情况。所以有一个问题我们一直没有解决,对于这些hic数据的矫正?问题也涉及到hic-pro中的ice问题。
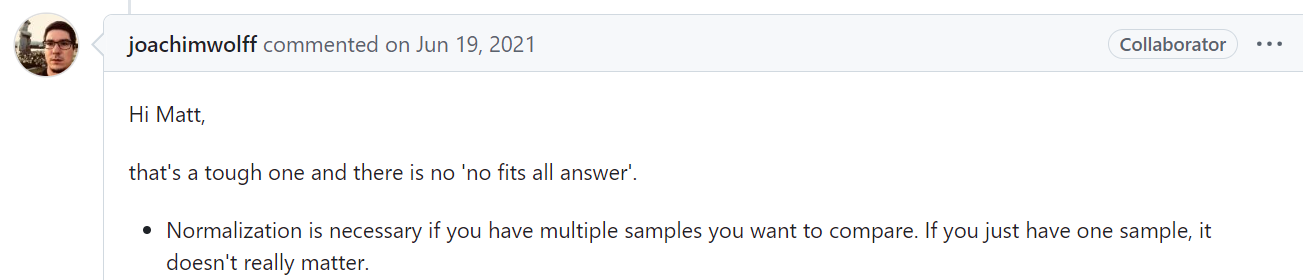
对此,我挑选了一些在hicexplorer中有代表性的issue,就是为了打通hicpro到hicexplorer的中间环,
<https://github.com/deeptools/HiCExplorer/issues/753>
另外参考这里的:
<https://github.com/deeptools/HiCExplorer/issues/681>
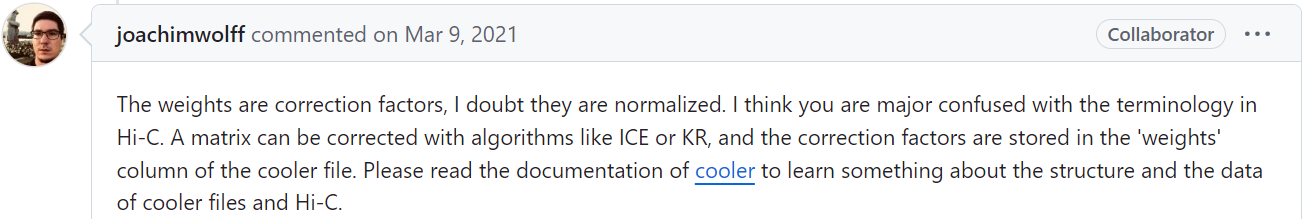
所以至少有一点需要确认的是:如果上游hic-pro用的数据不是raw而是iced来转换(即经过ICE矫正),那么下游应该不能再进行矫正了(因为矫正本质也是ICE或KR之类算法)
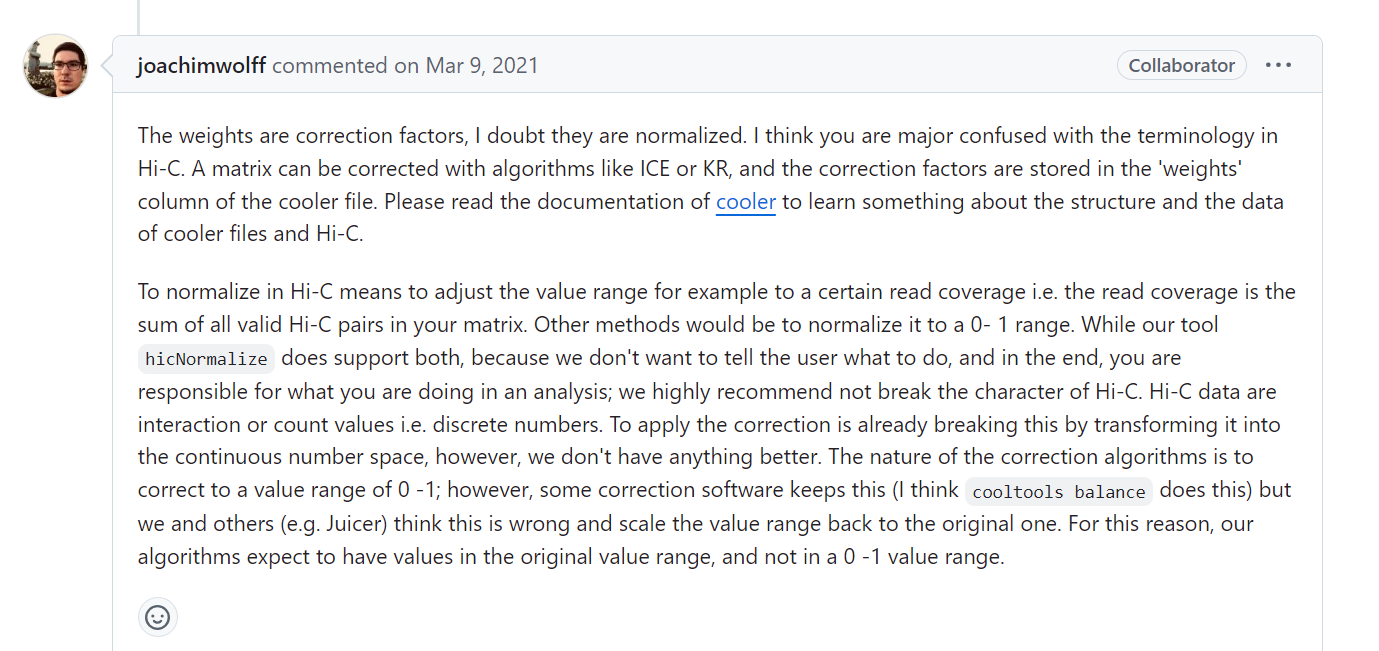
这里其实也提到了diss cooler balance的矫正算法,所以如果要矫正(就考虑清楚并理解清楚hic-pro的iced即ICE、juicer也有hic的矫正算法、以及hicexplorer的hicNormalize);
<https://github.com/deeptools/HiCExplorer/issues/567>
最重磅的还是下面这个issue:724
<https://github.com/deeptools/HiCExplorer/issues/724>
首先里面又提到了两个issue: 661和570
<https://github.com/deeptools/HiCExplorer/issues/661>,
这里有个线索:
 <https://github.com/deeptools/HiCExplorer/issues/570>
<https://github.com/deeptools/HiCExplorer/issues/570>
570里的描述其实就很直白了:
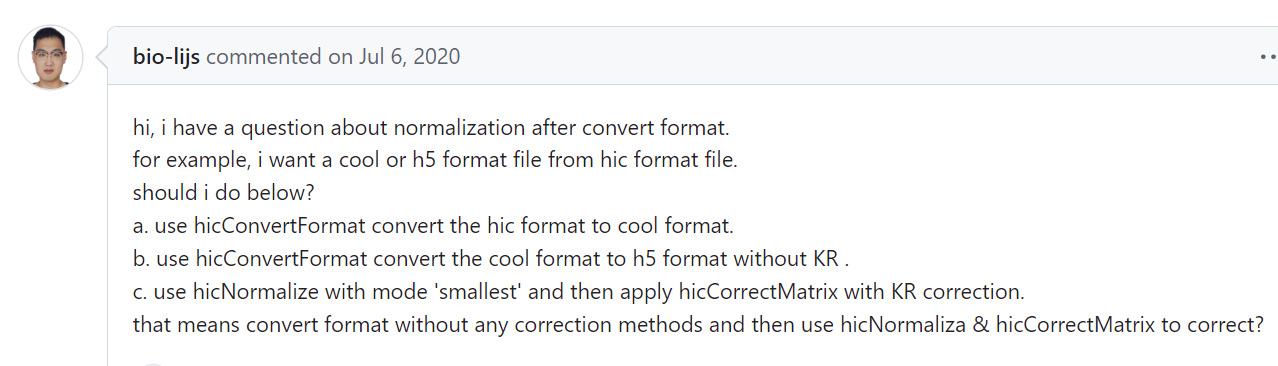
数据转换过程中不涉及到矫正,数据转换之后再使用hicNormalize&hicCorrectMatrix来矫正!
再回到<https://hicexplorer.readthedocs.io/en/latest/content/tools/hicConvertFormat.html#usage-example>
只有在cool到cool格式转换中才会出现–correction_name参数,(注意使用hicInfo工具!!!)

大概意思就是hicexplorer预期矫正因子在weight列,但是hic文件中矫正因子列通常不是这个名称,而是 KR, VC or VC_SQRT,
所以在hic2cool处理之后需要进一步cool 2 cool,将矫正因子列矫正到hicexplorer能够使用的weight列中?
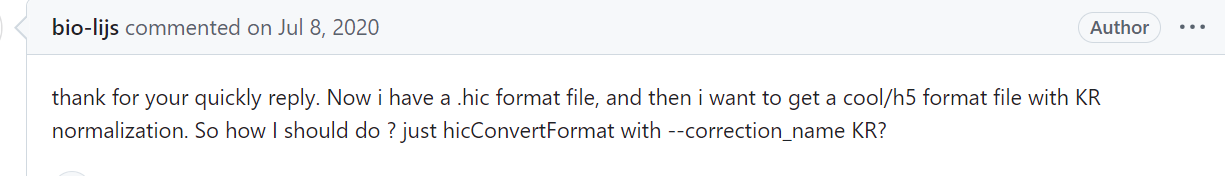
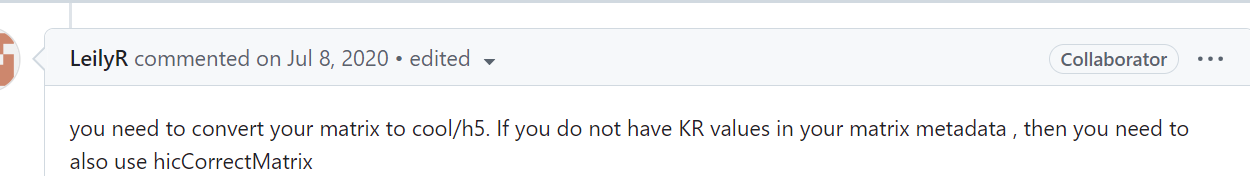
所以道理就很简单了:如果是转换之后没有KR值的(比如说hic-pro,没有走上面hic那一套),那么这样转换出来的cool格式,需要hicNormalize&hicCorrectMatrix 两套流程都上;
如果KR处理了,只要hicNormalize即可。
现在回到issue 724,

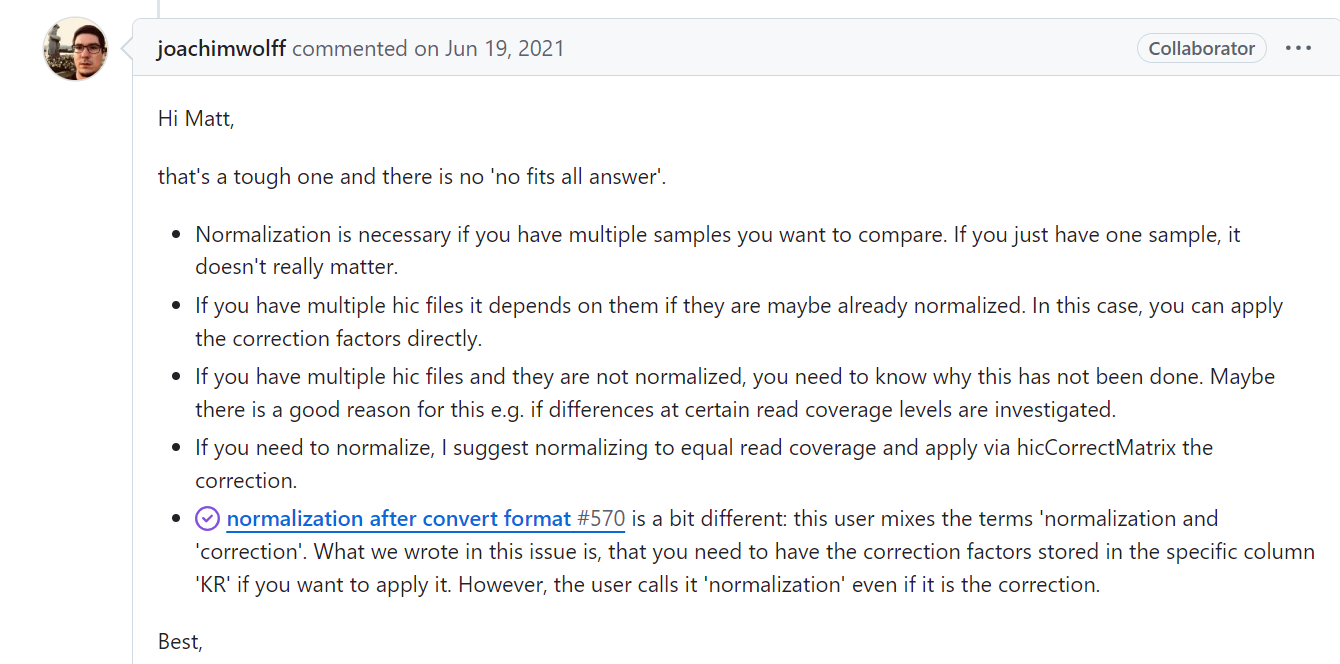
确实提问中说了:
使用hicInfo查看你的上游数据(转换之后的cool),如果矩阵中有correction因子(比如hic文件的KR列,那么在转换成cool过程中就可以使用correction_name KR了),那就后续只要用hicNormalize来处理,否则hicCorrectMatrix也要用;
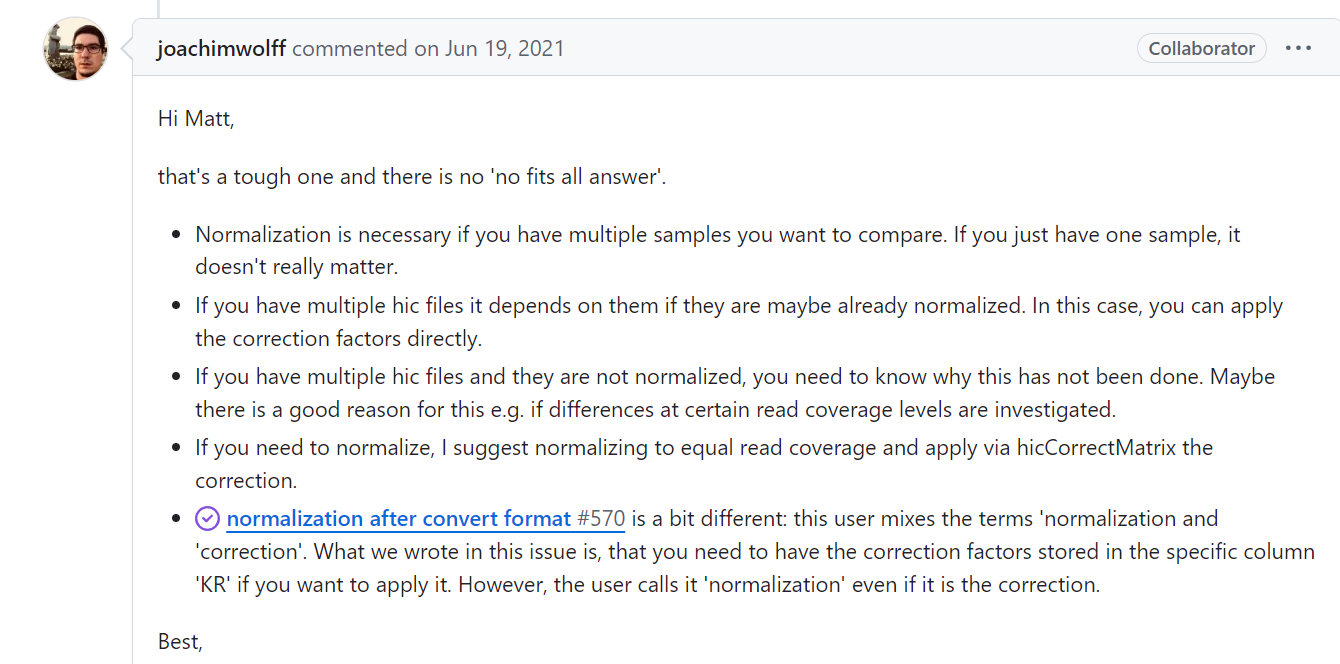
再来看一下回答,那道理就很简单了:
首先,我是比较多样本,所以我肯定需要矫正过程;
其次,我确定使用hic-pro的raw文件是没有normalized的,
所以normalizing first and then using hicCorrectMatrix,

看看该issue是怎么做的:

不同样本的同1分辨率下进行hicNormalize,并且是 --normalize smallest参数,
再然后对每个分辨率样本hicCorrectMatrix --correctionMethod KR
所以回过头来看脚本<https://github.com/MaybeBio/TNBC-project/blob/main/1%2CHiC-pro%20upstream%20processing/hicpro_cool_mcool.sh>,
每个分辨率下统一hicNormalize --normalize smallest,
然后单独每个样本接着hicCorrectMatrix correct --correctionMethod KR,
这样矫正的问题解决了,然后就是cool合并为mcool了!
这两个函数参考<https://hicexplorer.readthedocs.io/en/latest/content/tools/hicNormalize.html>以及<https://hicexplorer.readthedocs.io/en/latest/content/tools/hicCorrectMatrix.html>

我们可以知道,官方矫正其实有两种方式:

其中一种就是hic-pro中iced文件夹中的ICE方法,但是我们选用raw文件夹就没有这个问题了,所以下游可以勇敢地使用KR了!(相当于上游数据没有校正因子,所以需要重新矫正),
至于为什么这里矫正不使用ICE而是使用KR,因为KR算法在hicexplorer中运行优化了,其次使用ICE按照官方指示需要评估参数之类比较麻烦,还不如直接使用KR默认处理!
所以,使用hic-pro raw转换为cool,再normalize-correct,最后cool合并为mcool,参照我的脚本<https://github.com/MaybeBio/TNBC-project/blob/main/1%2CHiC-pro%20upstream%20processing/hicpro_cool_mcool.sh>就对了!
如果没有特殊情况,就使用这种方法获取cool文件!
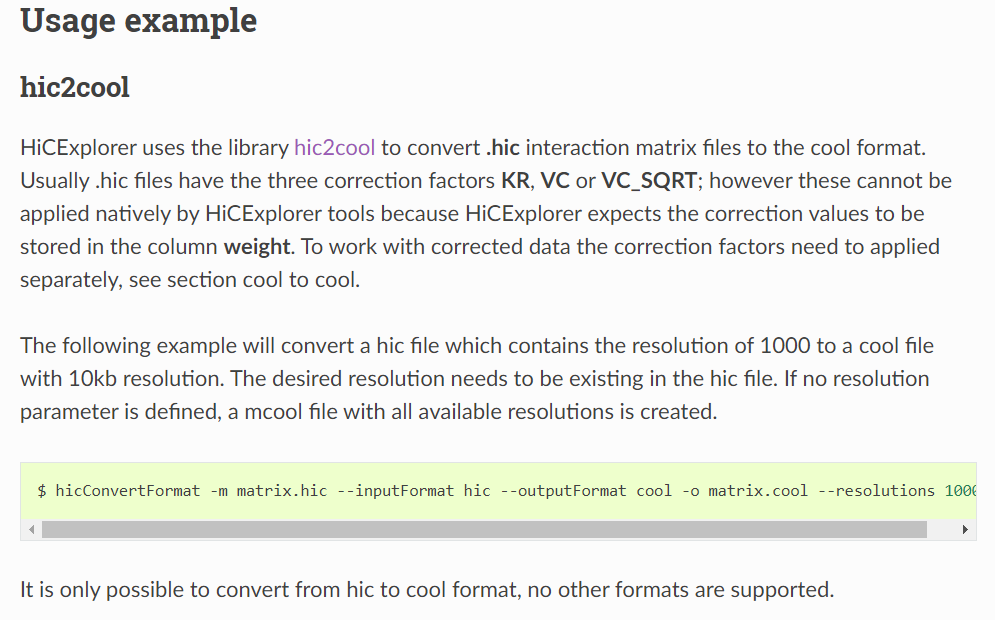
(2)hic格式到cool格式:
如果上游数据是.hic数据,我们发现hicexploer实际上不支持hicpro到hic数据格式,所以这种情况要么是我们上游使用hic-pro的脚本将hicpro数据格式转换为了hic,要么是我们上游的数据一开始就是hic(比如使用juicer的pipeline),
(3)另外还有一些曲折的数据格式转换,我也都或多或少地看过了,只觉得非常麻烦以及混乱:
都是从hic-pro、hicexplorer中的issue中找到的,当然也可以逛一下hic2cool、genova、juicer,事实上如果这些主流工具的issue都收集逛过了,仔细比较分析过了,那么总会找到最可信的一种数据处理方法!
<https://github.com/nservant/HiC-Pro/issues/614>
<https://github.com/nservant/HiC-Pro/issues/621>
写这篇博客花了我不少时间整理,毕竟做的项目太过久远了,要回忆起来细节真不容易!
























 5247
5247

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








