水稻种子内生核心微生物组垂直传播机制
Dynamics of rice microbiomes reveal core vertically transmitted seed endophytes

Research article,2022年12月9日,Microbiome,[IF 16.837]
原文链接:https://doi.org/10.1186/s40168-022-01422-9
第一作者:Xiaoxia Zhang(张晓霞);Yi-Nan Ma(马毅楠)
通讯作者:Hai-Lei Wei(魏海雷)
合作作者:Xing Wang(王星); Kaiji Liao(廖开吉); Shanwen He(何山文); Xia Zhao(赵霞); Hebao Guo(郭鹤宝); Dongfang Zhao(赵东芳)
主要单位:
中国农科院农业资源与农业区划研究所(Key Laboratory of Microbial Resources Collection and Preservation, Ministry of Agriculture and Rural Afairs, Institute of Agricultural Resources and Regional Planning, Chinese Academy of Agricultural Sciences, Beijing 100081, China)
- 导读 -
植物相关微生物组被称为植物的“第二基因组”,而内生微生物组是植物相关微生物组的核心,与植物进化、抗病抗逆、营养转化具有密切联系。但是,有关内生微生物的传播途径仍然存在很多争论,其功能机制也有待进一步挖掘。本研究以四个种植区的六个水稻品种,历经两个完整生长季的五个不同组织为样本,通过高通量微生物组深度解析,阐明了水稻微生物组群落组装的偏好性及在水稻不同部位的动态变化,发现了14个水稻内生核心分类单元(ASV),并成功分离到可垂直传播的3个泛菌种类和1个糖黄单胞种类。基因组分析表明,这两类细菌基因组简单、无致病特征、产特殊次生代谢物质和酶类等,且具有溶磷、促生等益生功能。该研究创新性地提出并分析验证了水稻种子内生微生物的垂直传播机制,同时建立了水稻内生微生物资源库。
- 实验设计 -
本研究基于两代水稻、6个品种(RBQ, L31, M63, P64, Dular和Kasalath)、4个种植区(三亚,廊坊,南昌和西双版纳)、5个取样部位(散土,根际土,根内,茎内,种子内)的481份样品进行了高通量微生物组深度解析。

- 结果 -
1. 水稻微生物群落多样性及其驱动因素
Diversity and driving factors of rice microbiomes
作者利用Chao1和Shannon指数描述样品的α-多样性后发现,同一水稻品种的根际、根、茎和种子内生微生物在不同地区的α-多样性没有显著差异。此外,6个水稻品种的根际、根内、茎内和种子内样品的α-多样性指数在4个种植地点没有显著差异,说明水稻基因型对微生物多样性没有影响。此外,根际和散土微生物群落的α-多样性在4个种植地点具有明显的差异,然而,内生微生物(茎、根和种子)多样性在4个地点具有不同的分布,表明外界环境对水稻内部的微生物多样性影响不大。重要的是,作者还发现水稻微生物的α-多样性不受品种和种植地区的影响,始终呈现从根际、根、茎到种子内降低的规律。
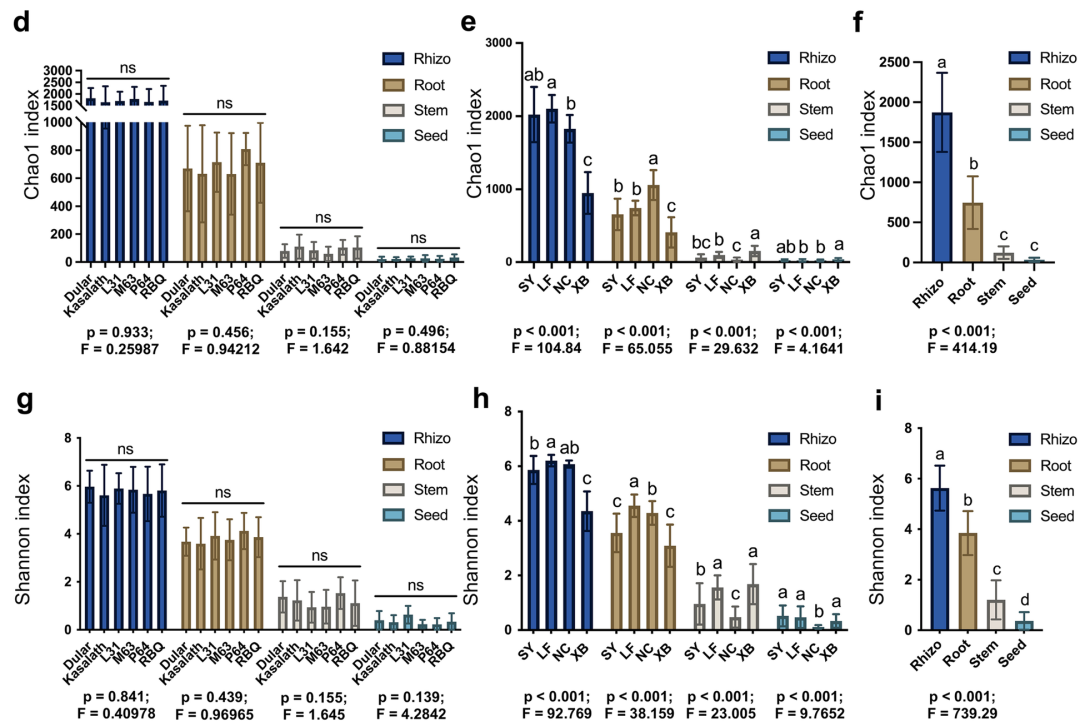
图1:水稻相关微生物群落的α-多样性。
d和g, 6个水稻品种不同取样部位的4个种植地区合集的Chao1和Shannon指数。e和h, 4个种植地区6个水稻品种不同微生境微生物区系的Chao1和Shannon指数。f和I, 不同取样部位的所有水稻品种和种植地区合集的Chao1和Shannon指数。
通过基于Bray-Curtis相异性的PCoA分析,结果显示取样部位是微生物组变异的最主要影响因素(R2 = 0.314, p < 0.001),而受种植地区(R2 = 0.0967, p < 0.001)和水稻品种(R2 = 0.0106, p = 0.478)的影响较小。可见,取样部位是水稻微生物组组成的主要驱动力。这些数据表明,水稻微生物群落的多样性从根部远处到近处、从外部到内部、从地下到地上呈稳步下降趋势。

图2:基于Bray-Curtis相异性的水稻取样部位、种植地区和品种的PCoA分析。
2. 水稻微生物的群落组成及动态变化
Taxonomic composition and dynamics of rice microbiota
为了调查水稻微生物的群落组成及动态变化,作者分析了在不同条件下的水稻微生物群落富集和组成情况。对于散土样品,三亚种植区的β-变形菌纲(Betaproteobacteria)的占比最高(57.9%),而其他细菌菌纲占比均小于10%。相比之下,放线菌纲(Actinobacteria)广泛分布在廊坊和西双版纳种植区,而疣微菌门(Verrucomicrobia subdivision 3)在南昌种植区显著富集。根际土样品细菌群落组成与散土样品十分相似,但少数类群在这两种取样部位间变化明显,例如β-变形菌纲(Betaproteobacteria)。γ-变形菌纲(Gammaproteobacteria)是唯一一个在散土、根际、根、茎和种子中逐渐富集的菌纲,而放线菌纲、α-变形菌纲和β-变形菌纲逐渐减少,说明水稻内部生态位更加有利于γ-变形菌纲的生存。因此,来自于γ-变形菌纲的泛菌属(Pantoea)和黄单胞菌属(Xanthomonas)比来自α-变形菌纲的鞘氨醇单胞菌属(Sphingomonas)和β-变形菌纲的食酸菌属(Acidovorax)在研究水稻内生方面更加具有优势。此外,在不同取样部位中丰度最高的前五个菌纲和菌属呈现动态变化,在子代种子样品中,γ-变形菌纲和泛菌属最为优势。

图3:水稻取样部位微生物群落组成。
a, 纲水平散土、根际土、根内、茎内、子代种子和亲代种子内生微生物组成柱状图。b, 纲水平各取样部位前五汇总图。c, 属水平散土、根际土、根内、茎内、子代种子和亲代种子内生微生物组成柱状图。d, 属水平各取样部位前五汇总图。
3. 细菌共现性网络和关键类群
Bacterial co-occurrence networks and keystone taxa
通过构建不同取样部位的细菌共现性网络,作者进一步解析了细菌类群和取样部位间复杂相互作用对水稻微生物群落组成的影响。总体而言,网络的复杂性从根际、根内、茎内到种子内逐步降低。根据模块化指数可以观察到地下部分(散土、根际土和根内)比地上部分(茎内和种子内)具有更明显的模块化趋势。子代种子内样品的网络节点数、边数和平均聚类系数均为最低,网络组成最为简单。由此可见,取样部位对微生物网络的构建具有显著影响。作者根据网络节点解析微生物群落中的关键微生物。网络节点中变形菌门数量最多,且在茎内和种子内生样品中所有的节点微生物都来自变形菌门,说明该菌门在水稻内生微生物组中的重要地位。

图4:基于SparCC构建的微生物网络及网络参数。
a, 水稻微生物组不同取样部位间的共现性网络。每个节点代表一个细菌ASV,青色标记代表网络节点,边的颜色代表作用类型,红色代表正相关,蓝色代表负相关。b, 各取样部位间微生物网络的主要拓扑结构特征。
4. 水稻核心内生菌群
Core rice endophytic microbiota
为了挖掘能够在水稻中垂直传播的核心内生微生物类群,作者首先以大于70%的阈值在不同地点、水稻品种和微生境中提取核心ASV,最终在根、茎和子代种子内生样品中分别发现了438个、94个和27个ASV,其中三者共有的ASV为14个,分布在两个菌门,6个菌目。因此,作者推测水稻核心内生菌群的组成并不完全随机,而是受到了细菌特征、宿主环境和代谢特点的影响。
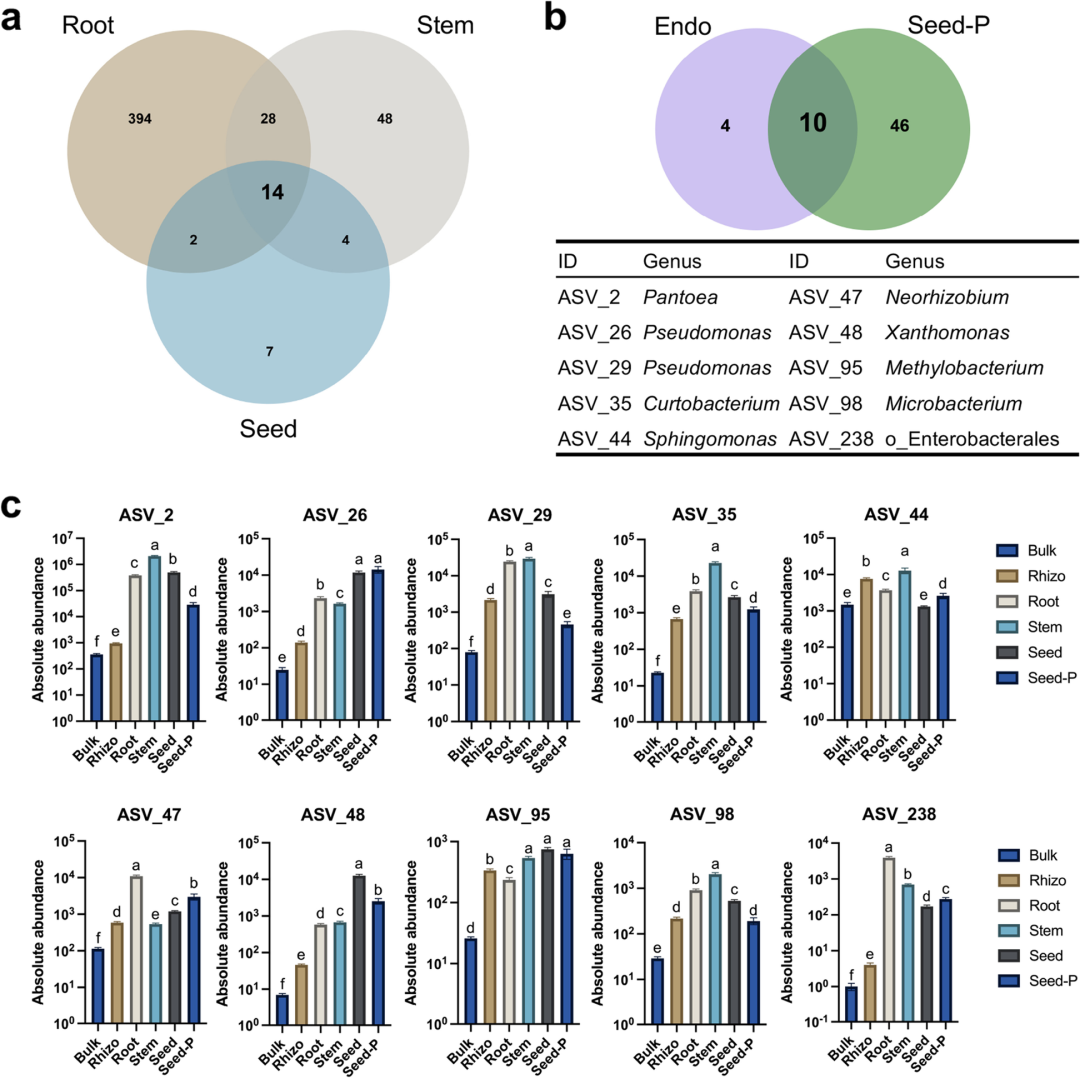
图5:水稻核心内生微生物和垂直传播类群。
a, 韦恩图显示高频率出现在水稻内生取样部位(根内、茎内和种子内)的ASV。b, 韦恩图展示10个潜在的从亲本种子垂直传播到子代种子的ASV。c, 在不同取样部位的潜在垂直传播ASV的绝对丰度。
5. 种子内生菌垂直传播的证据
Evidence for the vertical transmission of seed endophytes
为鉴定潜在的垂直传播细菌类群,作者分析了核心内生菌和亲代种子内生样品之间的重叠ASV,发现在14个共有内生ASV中,10个均来自亲代种子内生样品。同时ASV_2(Pantoea)、ASV_26 (Pseudomonas)、ASV_48 (Xanthomonas)和ASV_238 (o_Enterobacterales) 在根内、茎内、亲代种子和子代种子样品中的绝对丰度和出现频率均显著高于散土和根际土样品,表明这4种ASV最有可能是垂直传播的类群。
为进一步获得垂直传播的细菌类群,作者对种子内生细菌进行了高通量地分离、培养、纯化和鉴定,从4个种植地区和2个水稻品种(P64和Dular)的亲子代种子中分离获得了957株细菌。其中泛菌和黄单胞菌是数量最多的两个细菌菌属,分别占分离菌株的39.39%和27.69%。21株泛菌和27株黄单胞菌的部分16S rRNA基因序列分别与ASV_2和ASV_48序列完全相同。其中,9株泛菌和17株黄单胞菌来源于子代种子样品,其余均来自亲代种子样品。作者对这些菌株进行了基因组草图测序并进行了系统发育分析,发现分离获得的泛菌菌株分别属于P. ananatis,P. dispersa,和 P. stewartia三个种,黄单胞菌均为X. sacchari种。通过比对亲代和子代的ANI值和核心基因组的相似性,发现来源于子代种子的8株泛菌与来源于亲代种子的9株泛菌具有极高的相似性;来自子代种子样品的4株黄单胞菌与来自亲代的4株菌株黄单胞菌具有极高相似性。以上结果表明种子内生菌在株系水平上存在垂直传播。
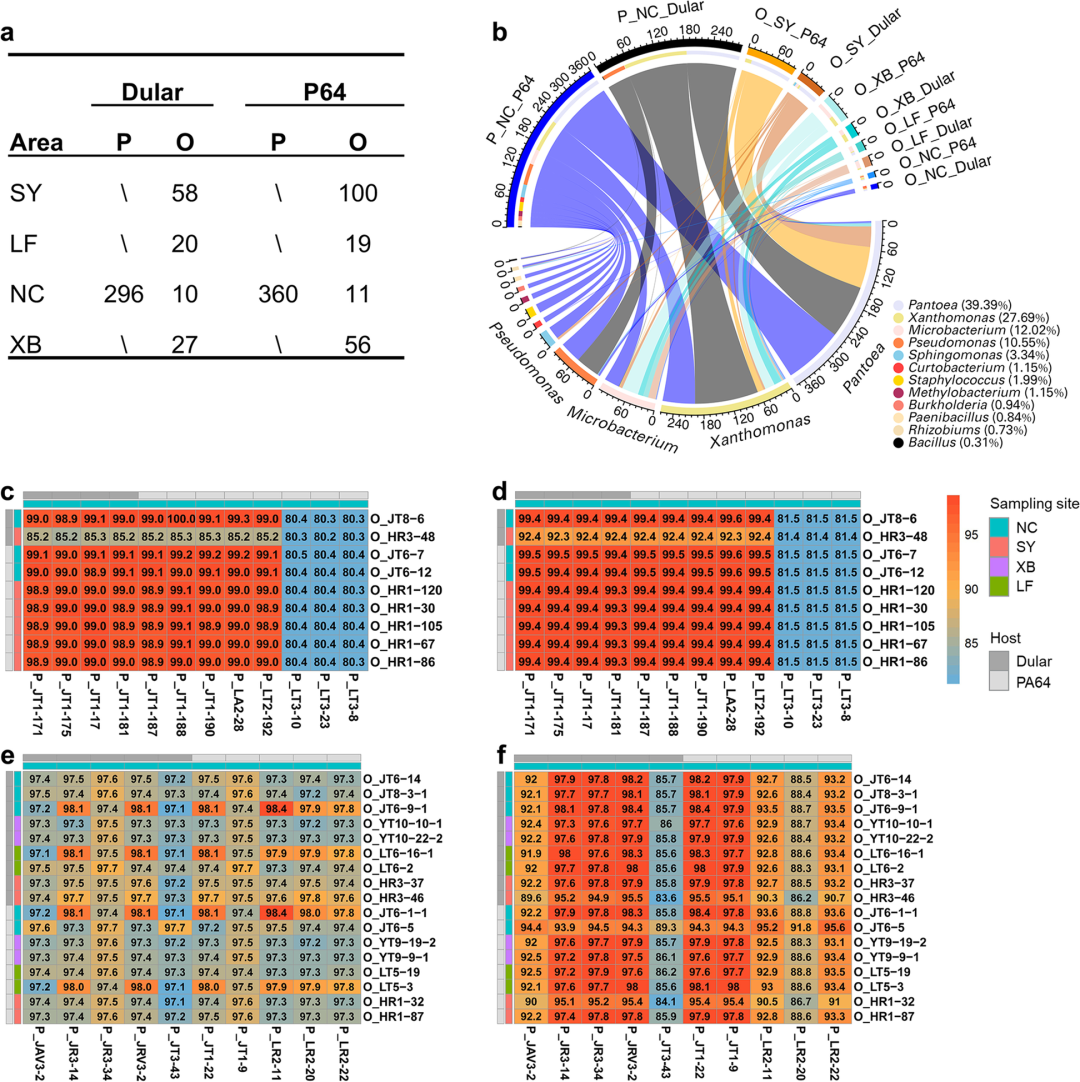
图6:可培养的垂直传播水稻种子内生菌鉴定。
a, 四个水稻种植区、两个水稻品种的亲代种子和子代种子中分离获得的可培养细菌菌株数量。b, 圈图展示a中菌株属水平相关关系。c, 亲代种子和子代种子样品中分离的泛菌菌株ANI热图。d, 亲代种子和子代种子样品中分离的泛菌菌株串联核心基因组一致性热图。e, 亲代种子和子代种子样品中分离的黄单胞菌菌株ANI热图。f, 亲代种子和子代种子样品中分离的黄单胞菌菌株串联核心基因组一致性热图。
6. 可垂直传播的种子内生菌群
基因组挖掘和功能特征
Genome mining and phenotypic characterization of vertically transmitted seed endophytes
为了进一步阐明水稻中可垂直传播类群的潜在功能,作者对所有已测序的内生泛菌和黄单胞菌菌株进行基因组挖掘分析。作者计算了泛菌和黄单胞菌的泛基因和核心基因数量,并利用COG和KEGG数据库对核心基因进行了功能注释。在COG注释中,核心基因组显著富集在E(氨基酸转运和代谢)和G(碳水化合物转运和代谢)两个功能类别中,且KEGG注释中同样存在高度相关的 “碳水化合物代谢”和“氨基酸代谢” 通路。随后,作者重点关注了级代谢产物、蛋白质分泌系统和酶三大功能类别,分析中发现所有泛菌菌株都含有促进植物生长相关的1-氨基环丙烷-1-羧酸(ACC)脱氨酶和吲哚乙酰胺水解酶(iaaH)。此外,所有泛菌基因组中都具有编码多种消化酶的基因,如右旋糖酶、β-半乳糖苷酶、果胶酶、纤维素酶和淀粉酶基因。并且均具有T1SS、T5aSS和T6SS,而只有少数菌株具有T2SS、T3SS、T4SS、T5bSS和T5cSS。黄单胞菌基因组中也具有iaaH基因和丰富的消化酶编码基因如β-半乳糖苷酶、淀粉酶和果胶酶基因。然而,分离出的黄单胞菌菌株基因组中仅含有T1SS、T4SS和T5SS,而不含致病性T3SS和T6SS。随后作者对部分泛菌和黄单胞菌进行了一些促生功能特征的检测并发现所有菌株都具有纤维素酶活性,并能够产生吲哚-3-乙酸(IAA),这与基因组挖掘的结果相对应。

水稻种子垂直传播内生菌的系统发育分析。
a, 基于1258个单拷贝同源基因的串联多序列比对建立的21株泛菌菌株与20株模式菌株的最大似然发系统发育关系。b, 基于892个单拷贝同源基因的串联多序列比对建立的27株泛菌菌株与22株模式菌株的最大似然发系统发育关系。SM,次生代谢产物;PSS,蛋白质分泌系统;ENZ,消化酶。
- 总结 -
本研究建立了水稻内生微生物资源库,并通过多尺度微生物组学分析,阐明了种子内生微生物组中核心类群的垂直传播与功能特征,对未来开发种子内生菌并提高植物适应性奠定了理论基础。对进一步理解微生物-植物共进化理论提供了新的证据。水稻内生微生物资源在营养转化与吸收、抗病抗逆、耐胁迫等方面表现出的巨大潜力,也为微生物肥料、微生物农药、微生物种衣剂、微生物防腐剂的研发开辟了新的思路。
参考文献
Zhang, X., Ma, YN., Wang, X. et al. Dynamics of rice microbiomes reveal core vertically transmitted seed endophytes. Microbiome 10, 216 (2022). https://doi.org/10.1186/s40168-022-01422-9
- 作者简介 -
第一作者

中国农业科学院
农业资源与农业区划研究所
张晓霞
研究员
主要从事农业微生物的保藏、评价以及植物与微生物的互作研究。承担多项国家及省部级课题并参与国家自然科技资源平台“微生物菌种资源”的建设与运行、中国农业微生物种质资源保护与利用体系建设。已获得国家发明专利10项;发表学术论文50余篇,主编及参编著书5本,获得农业部中华农业科技奖三等奖。

中国农业科学院
农业资源与农业区划研究所
马毅楠
博士后
主要关注土壤、植物微生物组学以及植物与微生物的互作研究。目前以第一作者发表1篇Microbiome、1篇Frontiers in Plant Science。
通讯作者

中国农业科学院
农业资源与农业区划研究所
魏海雷
研究员
魏海雷,博士,研究员。中国农业科学院农业资源与农业区划研究所农业微生物资源创新团队首席科学家,国家菌种资源库常务副主任,中国农业微生物菌种保藏管理中心主任,农业农村部农业微生物资源收集与保藏重点实验室主任,中国微生物学会微生物资源专业委员会,副主任。长期从事微生物资源利用与功能基因组学研究。先后主持国家重点研发计划、科技部科技基础条件平台项目、国际科学基金、国家自然科学基金、北京市科技计划等项目和课题,在Cell Host & Microbe,Cell Reports,Microbiome,EMBO Journal,Molecular Plant Pathology,Molecular Microbiology,Cellular Microbiology,Frontiers in Plant Science等发表30多篇研究论文。目前兼任学术期刊Frontiers in Microbiology,BMC Plant Biology副主编。
猜你喜欢
iMeta简介 高引文章 高颜值绘图imageGP 网络分析iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文,跳转最新文章目录阅读























 7964
7964

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








