中文文档先于英文文档发布,TTfriends公众号首发。
最新完整文档请访问在线版:tongzhou2017.github.io/itol.toolkit/
前言
为了系统展示itol.toolkit的多个功能协同使用,我们选择了一些用iTOL制作并已发表的图来复现。复现必须的数据是Newick格式的树文件以及用于注释的元数据,如果没有元数据,也可以使用模版文件逆向生成元数据。
本节文档使用的数据来自厉舒祯等2022年发表在Journal of Hazardous Materials的论文:🔗 DARHD: a sequence database for aromatic ring-hydroxylating dioxygenase analysis and primer evaluation。作者用几年时间构建了一个芳环羟化双加氧酶数据库,芳环羟化双加氧酶催化的反应为多种芳香化合物微生物好氧降解的第一步及限速步骤之一,收集了功能基因序列并构建了相应的数据库,对现存的100多对功能基因的引物进行了评价。
在作者的协助下,我们获得了数据,数据存放在:https://github.com/TongZhou2017/itol.toolkit/discussions/9
考虑到网络访问问题以及为了方便用户学习,将本文所需的全部代码及对应版本的安装包打包放在公众号后台,留言“itol.toolkit”即可获得。
初步评估
根据论文中的结果图,以及作者提供的树及模版文件,我们大致评估该图复现需要用到以下功能:
DATASET_DOMAINS
LABELS
TREECOLORS
同时我们使用作者提供的模版文件进行了实测,对比发现有一些可以优化的工作流,可以进一步提升该图的可复现性以及操作简便性。我们将最终优化完成的工作流展示给大家。优化后实际使用的功能有:
DATASET_TEXT
LABELS
TREECOLORS
优化复现
我们按从内到外的顺序进行复现。
键(key)信息的书写使用了rep表示复现系列文档,Li2022jhm表示论文信息,第一个数字表示推荐拖入顺序,之后的字符串表示所使用的模版类型,如果同文档中使用了多个同类模版,则会在最后追加序号。
加载所需工具。
library(itol.toolkit)
library(data.table) # 用于元数据文件读取
library(ape) # 用于树文件读写
library(stringr) # 用于字符串处理加载数据。树文件的分枝名包含了物种名等冗余信息,需要使用正则表达式匹配去除。元数据中如果包含多余或重复id也需要进行处理。
tree <- "tree.nwk" # 树分枝名存在多余信息会导致注释文件匹配失败
# 修改树分枝名
phylo <- ape::read.tree(tree)
phylo$tip.label <- stringr::str_remove(phylo$tip.label, "_.*$") # 去除多余信息
tree <- "tree_short_label.nwk"
ape::write.tree(phylo,tree) # 导出树,实际上传该树进行可视化
# 创建本次分析的数据及样式仓库
hub <- create_hub(tree)
# 加载元数据
data_file <- "iTOL_annotation.txt"
data <- data.table::fread(data_file)
data <- data %>% filter(ID != "Seq30_") # 去除多余信息首先要使用PRUNE模版功能去除不属于双加氧酶大亚基的分枝。
select_tip = phylo$tip.label[phylo$tip.label != "Seq372"]
unit_1 <- create_unit(data = select_tip,
key = "rep_Li2022jhm_1_prune",
type = "PRUNE",
tree = tree)第二层是内环文本信息,作者使用DATASET_DOMAINS实现,但该方法会增加后续图像处理的复杂度,比如文本对齐、形状去除。如果使用DATASET_COLORSTRIP其实是最合适的,因为直接就有对label位置靠左、居中、靠右的设置,但很可惜,在环形图中该功能失效。所以我们最终选择DATASET_TEXT模版功能优化复现。这里建议使用1.0.1及以上版本。
其中rotation参数将字符串旋转180度,实现了内环文字的靠左对齐效果。
margin样式参数设置为-500,可以实现将外环文字缩进到内环。
同时需要对全局的label margin在操作面板上进行设置,这一参数无法使用模版文件修改,该参数本示例设置了150。
刚刚拖入的注释文件会有重叠现象,这是正常的,在iTOL控制面板稍微增减DATASET的margin参数就会刷新效果。
df_gene <- data %>% select(ID, Gene_name)
unit_2 <- create_unit(data = df_gene,
key = "rep_Li2022jhm_4_text_1",
type = "DATASET_TEXT",
size_factor = 1,
rotation= 180,
position = -1,
color = "#000000",
tree = tree)
unit_2@common_themes$basic_theme$margin <- -500第三层使用LABELS模版功能实现分枝重命名。
df_relabel <- data %>% select(ID, Substrate)
unit_3 <- create_unit(data = df_relabel,
key = "rep_Li2022jhm_2_labels",
type = "LABELS",
tree = tree)第四层使用TREE_COLORS模版功能的range类型,实现分枝label的区域配色,使用wesanderson色盘。
值得注意的是,官方模版需要用户输入4列数据:编号、类型、颜色、标签。而使用itol.toolkit可以只输入2列必填信息,其他信息自动根据参数填充,如颜色可以根据数据列(此时数据列与标签列一致)分配。如果输入完整的4列信息,程序也会进行自动的识别,默认第一列为编号,以#或rgb开头的列识别为颜色。
最大程度简化用户的数据整理过程,更加符合R语言用户的使用习惯,让工作流更加丝滑。
df_substrate <- data %>% select(ID, Substrate)
set.seed(666)
unit_4 <- create_unit(data = df_substrate,
key = "rep_Li2022jhm_3_range",
type = "TREE_COLORS",
subtype = "range",
color = "wesanderson",
tree = tree)最后再次使用DATASET_TEXT模版功能,生成外圈文本,这里rotation设置为0度,position为-1,实现靠右对齐效果。
df_species <- data %>% select(ID, Species)
unit_5 <- create_unit(data = df_species,
key = "rep_Li2022jhm_5_text_2",
type = "DATASET_TEXT",
size_factor = 1,
rotation= 0,
position = -1,
color ="#000000",
tree = tree)
unit_5@common_themes$basic_theme$margin <- 50最终将所有单元加入仓库,批量输出。
hub <- hub + unit_1 + unit_2 + unit_3 + unit_4 + unit_5
write_hub(hub, getwd())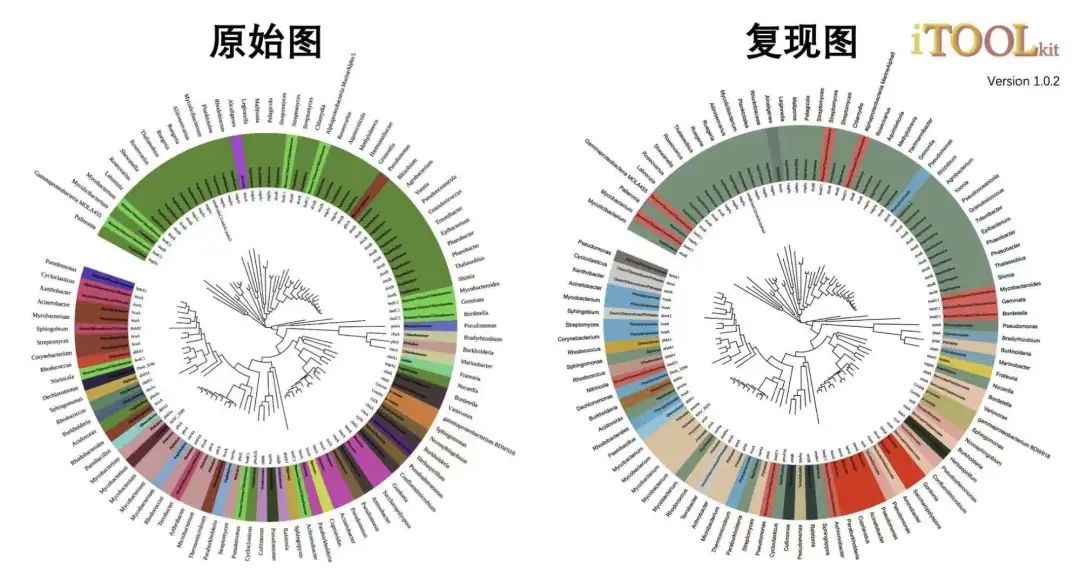
更新
GitHub版更新了2个补丁版本 1.0.1、1.0.2
1.0.1修复了以下问题:
使用DATASET_TEXT模版时,create_unit设置全局颜色报错
使用TREE_COLORS模版时,且节点数据为空时,合并至hub时,子函数hub_to_unit报错
1.0.2添加了一个新功能:
支持wesanderson配色盘,只在TREE_COLORS的range情境下生效,如果反馈好(比如本篇文档文末点赞超过前面3篇之和),考虑全面支持该配色
社群
加作者微信(id:zitangumu),邀请进入itol.toolkit用户群。
用户群功能:
解决用户使用itol.toolkit过程中遇到的报错
将用户提出的功能需求纳入开发列表
协助用户使用itol.toolkit完成数据分析
文档类型:用户-工作流-rep_Li2022jhm
链接状态:1
文档版本:1.0.2
文本:周通
编辑:凌浩
审阅:厉舒祯,凌浩,李明,赵启祥,张思威

END
TTfriends,是一个面向需求定制化开发生物信息分析工具的开发者团队,致力于推动科学前沿进步贡献新工具。欲了解更多详情,欢迎访问TTfirends官方网站:
tongzhou2017.github.io/ttfriends
Copyright © 2022 TTfirends























 461
461

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








