Methanosarcina的醋酸分解和甲基营养型甲烷生成的时间序列表达研究揭示菲律宾稻田土壤甲烷生成动力学
2024年德国马普陆地微生物所李馨博士以第一作者在国际权威学术期刊Microbiome上发表最新研究成果,题为Time‑shifted expression of acetoclastic and methylotrophic methanogenesis by a single Methanosarcina genomospecies predominates the methanogen dynamics in Philippine rice field soil,该研究深入探讨了稻田土壤中被低估的稻田微生物产甲烷途径。

Article,2024-02-26,Microbiome,[IF 15.5]
DOI:https://doi.org/10.1186/s40168-023-01739-z
原文链接:https://microbiomejournal.biomedcentral.com/articles/10.1186/s40168-023-01739-z
第一作者:Xin Li (李馨)
通讯作者:Jing Jing Peng (彭静静);Werner Liesack
主要单位:德国马普陆地微生物所
- 摘要 -
有机质厌氧分解的最后一步是产甲烷。稻田土壤是甲烷的主要人为源,而水稻秸秆是水稻种植的重要碳源。本研究破译了菲律宾稻田土壤在缺氧条件下长期培养(120天)期间产甲烷微生物组对水稻秸秆添加的结构和功能响应机制。该研究综合代谢物测定、特定生物标记基因(16S rRNA、mcrA)定量、宏基因组学和宏转录组学分析,揭示了两个重要的产甲烷古菌活跃阶段,即前期(第7至21天)和后期(第28至60天),这两个阶段之间以微生物的基因和转录丰度及CH4产生率的显著下降为分界线。这两个产甲烷的活跃阶段与甲烷八叠球菌科的物种丰度和基因表达趋势一致,但甲烷八叠球菌科所利用的产甲烷途径却不同。前期,三个不同的甲烷八叠球菌种群参与乙酸型产甲烷过程,而后期,则由单一的特定甲烷八叠球菌参与甲基型产甲烷途径。进一步通过Methanosarcina sp. MSH10X1 物种的转录和基因组组装(MAGs)分析结果表明,该菌株是乙酸型和甲基型产甲烷途径的关键物种。此外,厌氧产甲烷微生物食物网由一个复杂的细菌和古菌群落驱动,其中Geobacteraceae和Peptococcaceae可能与产甲烷古菌互作促进甲烷的产生。Methanocellaceae是氢营养型产甲烷途径的关键物种,而Methanotrichaceae的噬乙酸活性只有在群落活跃的后期才能被检测到。本研究除了乙酸型和氢营养型产甲烷作用的发生之外,甲基型产甲烷途径的高度表达是一项重要的新发现,它扩展了我们迄今为止对水稻土中产甲烷途径的认识。
- 前言 -
甲烷是仅次于二氧化碳(CO2)的第二大温室气体,在100年内全球增温潜势(GWP)为CO2的25倍,它贡献了每年全球人为温室气体排放的约15% (IPCC, 2007)。实际上,大气中CH4的排放量继续增加,最近3年(2020-2022年)是1983年以来全球年增幅最高的。厌氧产甲烷菌的微生物产甲烷是大气甲烷(CH4)的最大生物源。淹水稻田是人为向地球大气排放CH4的一个重要来源。目前对淹水稻田年排放率的估计在20至100 Tg/年之间。秸秆是作物生产中最丰富的可再生生物质资源之一,也是水稻土作为肥料添加的主要有机碳源之一。秸秆是一种木质纤维素生物质,由纤维素(32-37%)、半纤维素(29-37%)、木质素(5-15%)和果胶(2-3%)组成。秸秆组分作为一个复杂微生物群落的底物,最终将生物聚合物降解为CO2和CH4。因此,秸秆的厌氧降解最终促进了CH4的产生和排放。秸秆添加后稻田产甲烷降解涉及一个复杂的微生物食物网的活动,该微生物食物网是由各种功能微生物组成,包括水解菌、发酵菌、互营菌,及产甲烷古菌。这个微生物联合体互作完成了一系列厌氧降解步骤,包括聚合物水解、发酵、脂肪酸的互营养化、同型产乙酸和甲烷生成。目前已知的三种产甲烷途径:乙酸型(乙酸)、氢营养型(H2和CO2)和甲基营养型(甲醇和其他甲基化化合物)产甲烷。在稻田土壤中,乙酸型和氢营养型产甲烷已被证明以2:1的比例主导CH4的产生。而对意大利稻田土壤的分析表明,甲基营养化产甲烷作用在稻田土壤中仅起次要作用。
为了探究稻田土壤有机质分解和CH4产生的微生物机理,前人开展了大量研究。特别是,在过去的20年中,水稻秸秆添加稻田土壤的微宇宙培养体系已经被频繁用于研究有机质厌氧降解的代谢过程。例如,针对意大利水稻土的研究已经涉及双RNA水平上对产甲烷微生物组的动态分析,但仅考虑了水稻秸秆降解的早期阶段。相比之下,对菲律宾稻田土壤中产甲烷微生物组的研究仅限于利用基于PCR的特定生物标志物扩增子测序分析。然而,这种方法只能处理群落潜力的特定方面,并且在采样时既不能提供有关活性微生物群的信息,也不能提供其功能基因表达的信息。这促使我们采用多组学方法来解析菲律宾稻田土壤中产甲烷微生物组的结构和功能动态变化。
本研究以水稻秸秆添加水稻土的厌氧微宇宙培养为模型体系。鉴于之前对不同地理区域的水稻土微生物群落的研究结果,我们希望在一个复杂的产甲烷菌微生物组中检测到乙酸型和氢型产甲烷菌的优势表达。我们利用代谢物测定、宏基因组分析和双RNA的宏转录组学分析,揭示了产甲烷微生物的结构和功能的动态变化。微宇宙培养阶段分为三个连续阶段,分别为早期阶段(3至21天)、中期阶段(21至28天)和后期阶段(28至120天)。
- 结果 -
1. 代谢产物的变化
Metabolite turnover
乙酸、丙酸和丁酸是产甲烷过程中最重要的中间产物,其浓度在无氧培养初期短暂积累,然后迅速消耗并达到生成与消耗的平衡状态。在120天的培养期间,甲烷生成呈现出两个活跃阶段。甲烷浓度从第11到21天迅速增加,第21天至28天期间其生成速率下降,然而从第28至35天,甲烷生成再次变得活跃。

图1:稻田土壤在30°C培养120天期间中间产物和甲烷浓度
乙酸(a)、丁酸(b)和丙酸(c)的浓度变化,以及甲烷产量(d)。插入图显示了高分辨率下14至120天的乙酸浓度,11至120天的丁酸浓度,35至120天的丙酸浓度c。数据为均数±标准差(n = 3)。
2. 定量PCR和实时定量PCR
qPCR and RT‑qPCR
与甲烷生成结果一致,细菌和产甲烷菌标记基因及转录活性的绝对定量结果同样显示出两个活性阶段。这可能与底物的消耗与补充以及不同主导微生物和代谢途径的转变有关。

图2:稻田土壤中细菌16S rRNA和产甲烷古菌mcrA基因的定量
细菌16S rRNA基因(a)和转录本(c)的拷贝数及mcrA基因(b)和转录本(d)的拷贝数。数据为均数±标准差(n = 3)。两个培养时间点之间的差异采用单因素方差分析(PFDR < 0.05)。
3. 细菌宏转录组(16S rRNA,mRNA)物种特异性动力学
The bacterial metatranscriptome (16S rRNA, mRNA)Taxon‑specifc dynamics
随着无氧培养的进行,优势细菌群落也随之发生改变-从前期的Geobacteraceae到中期的Peptococcaceae再到后期的Heliobacteriaceae等。优势群落的转变与产甲烷过程的转变和土壤不同有机物质的降解有关。

图3:优势细菌群落在rRNA和mRNA水平上的相对丰度变化
丰度百分比分别与细菌总16S rRNA和mRNA有关。数据均为平均值±标准差(n = 3),* (PFDR ≤ 0.05)、** (PFDR ≤ 0.01)、*** (PFDR ≤ 0.001)表示显著性差异。
4. 产甲烷宏转录组(16S rRNA,mRNA)
The methanogen metatranscriptome (16S rRNA, mRNA)
类似地,产甲烷菌群落的相对丰度随着培养时间发生变化。但与细菌群落活性不同的是Methanosarcinaceae始终是实验土壤的主导产甲烷菌群落,并呈现出两个活性阶段。

图4:优势产甲烷菌群落在rRNA和mRNA水平上的相对丰度变化
它们的丰度百分比分别与总古菌16S rRNA和mRNA有关。数据均为平均值±标准差(n = 3),* (PFDR ≤ 0.05)、** (PFDR ≤ 0.01)、*** (PFDR ≤ 0.001)表示显著性差异。
5. 确定Methanosarcina的优势种群
Defning the dominant Methanosarcina populations
进一步对实验土壤的Methanosarcina 16S RNA序列进行了系统发育分析结果显示四个不同的Methanosarcina成员主导了Methanosarcinaceae的两个活性阶段。组装的近全长16S rRNA序列分为四个不同的Methanosarcina群体(组I至组IV),其丰度动态变化不同。II群与Methanosarcina sp. MSH10X1亲缘关系最密切,且在培养过程中处于优势地位。Methanosarcina Group IV是第二丰富的种群,与M. horanabensis密切相关。II组和IV组种群表现出相反的16S rRNA丰度动态,在第11天,II组的最低丰度和IV组的最高丰度 (51% [II组] vs. 34% [IV组])。在整个培养过程中,Methanosarcina Group I和Group III种群的转录物丰度较低,但相对稳定。

图5:稻田土壤泥浆中检测到的4个Methanosarcina种群的系统发育进化树及其在培养期间的相对丰度动态变化
a:基于Emirge从宏转录组数据集中(415个序列)组装的近全长16S rRNA序列(> 1200bp)和GTDB中Methanosarcina基因组提取的参考序列构建系统发育树。
b:四个不同的Methanosarcina群体(类群I ~类群IV)的相对丰度动态。数据为平均值±标准差(n = 3)。
6. 产甲烷途径的映射独立表达分析
Mapping‑independent expression analysis of methanogenic pathways
此外,三个主要产甲烷途径及其关键功能基因在不同培养阶段显示出不同的活性。有趣的是,乙酸型产甲烷途径在前期最为活跃,而此前常常被忽略的甲基型产甲烷途径与Methanosarcinaceae呈现出一致的“双峰”活性,并在培养后期成为主导产甲烷途径。

图6:不同甲烷生成途径的相对转录丰度随培养时间的动态变化
a:单个产甲烷途径在培养时间内的相对mRNA丰度动态
b:关键途径基因的转录动态
c:转录本的微生物分类分配。
相对丰度值是根据KEGG类别“甲烷代谢”的总mRNA计算的,根据TPM值计算相对表达水平。
7. 竞争性转录子映射到四个参考基因组
Competitive transcript mapping onto the four reference genomes
进一步对宏基因组的MAGs及其功能分析发现,在培养前期,三个不同的Methanosarcina种群参与乙酸型产甲烷途径,而甲基型产甲烷途径仅由GroupⅡ种群执行。这表明稻田土壤中由Methanosarcina参与的甲基型产甲烷途径可能被低估了。
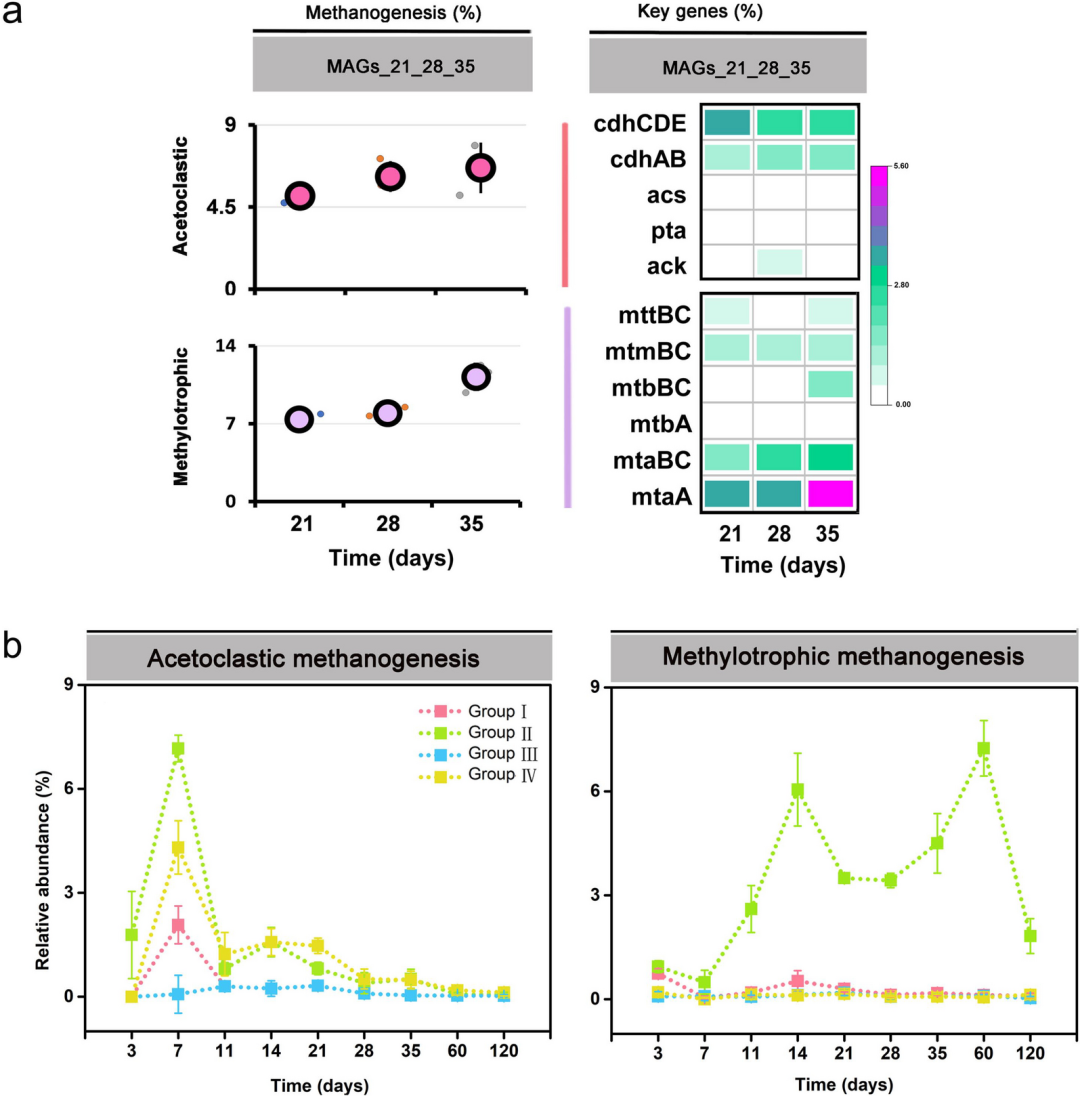
图7:Methanosarcina MAGs的产甲烷途径和关键途径基因的相对转录丰度
a:与总mRNA相关的途径特异性,mRNA和关键代谢途径基因转录本的相对丰度可以映射到产甲烷菌的基因组上。
b:通过Methanosarcina Group I至IV参考基因组的转录图谱推断出乙酸和甲基营养型产甲烷途径的相对转录动力学。
- 结论 -
探究稻田土壤甲烷产生的来源和产甲烷微生物组的响应机制对于精准预测全球甲烷排放至关重要。宏转录组学与宏基因组学方法为我们研究稻田土壤有机质厌氧分解的关键功能微生物的作用和动态变化提供了详细见解。特别是,Methanosarcina sp. MSH10X1物种在整个培养期内占主导地位。该物种在培养期内的产甲烷途径发生了显著的演替变化,其中乙酸型产甲烷途径在早期(约第7天)非常活跃,而甲基型产甲烷途径在后期非常活跃。厌氧培养的14天和60天后出现了两个独立的基因表达高峰。总的来说,我们的研究结果扩展了我们迄今为止对水稻土中活跃的生物产甲烷途径的认识,并表明甲基型产甲烷途径是某些稻田土壤中主要的甲烷生成途径。
参考文献
Li, X., Bei, Q., Rabiei Nematabad, M. et al. Time-shifted expression of acetoclastic and methylotrophic methanogenesis by a single Methanosarcina genomospecies predominates the methanogen dynamics in Philippine rice field soil. Microbiome 12, 39 (2024). https://doi.org/10.1186/s40168-023-01739-z
- 作者简介 -
第一作者

德国马普陆地微生物研究所
李馨
博士
主要研究方向为土壤厌氧微生物组、植物根际微生物组及植物与微生物互作。
通讯作者

中国农业大学
资源与环境学院
国家农业绿色发展研究院
彭静静
副教授、博士生导师
2011-2012年于中国科学院华南植物园基因工程中心助理研究员,2013-2018年于德国马普陆地微生物研究所环境基因组/转录组实验室进行博士后研究。主要研究方向为土壤微生物组功能特征与关键生物地球化学过程研究。在Microbiome、mSystems等期刊发表论文40余篇;担任mSystems、ISME Communications编委;iMeta、Soil Ecology Letters、生态环境学报青年编委。

德国马普陆地微生物研究所
Werner Liesack
博士生导师
主要研究方向为环境微生物基因组学、转录组学、甲烷产生及甲烷氧化过程。在Science、Nature、PNAS、ISME、Microbiome等期刊发表论文140余篇,H指数60。
宏基因组推荐
本公众号现全面开放投稿,希望文章作者讲出自己的科研故事,分享论文的精华与亮点。投稿请联系小编(微信号:yongxinliu 或 meta-genomics)
猜你喜欢
iMeta高引文章 fastp 复杂热图 ggtree 绘图imageGP 网络iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文,跳转最新文章目录阅读























 130
130

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








