iMeta 近2年最高引论文30篇
iMeta 近2年最高引论文TOP30覆盖前沿热点与技术突破,包括在线生信分析平台、微生物网络互作、肠道菌群研究等。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.238
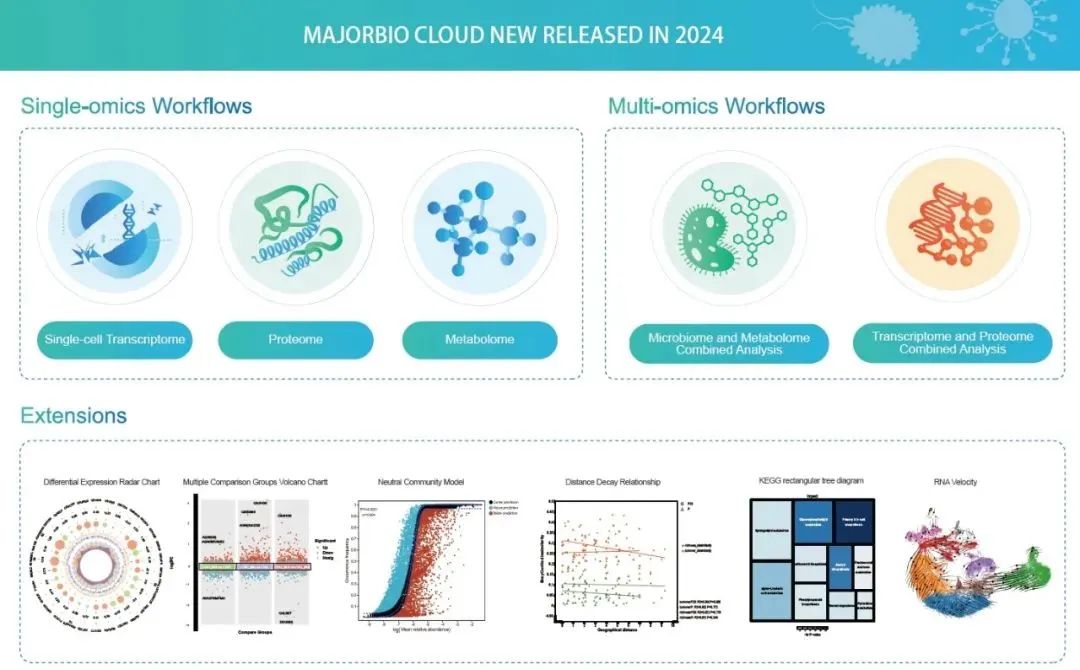
Majorbio Cloud 2024: Update single-cell and multiomics workflows
本研究在已开发的美吉生物云(https://cloud.majorbio.com/) 在线数据平台上发布了3款单组学云流程,2款多组学流程以及流程拓展工具助力多组学数据的挖掘和意义阐释。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.217
03 使用fastp进行超快的FASTQ数据预处理、质量控制和去重
Ultrafast one-pass FASTQ data preprocessing, quality control, and deduplication using fastp
本研究表明fastp已经得到了许多生物信息学用户的认可,并且一直在持续维护和更新,根据Google Scholar的数据,fastp论文目前已经被引15000多次。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.107
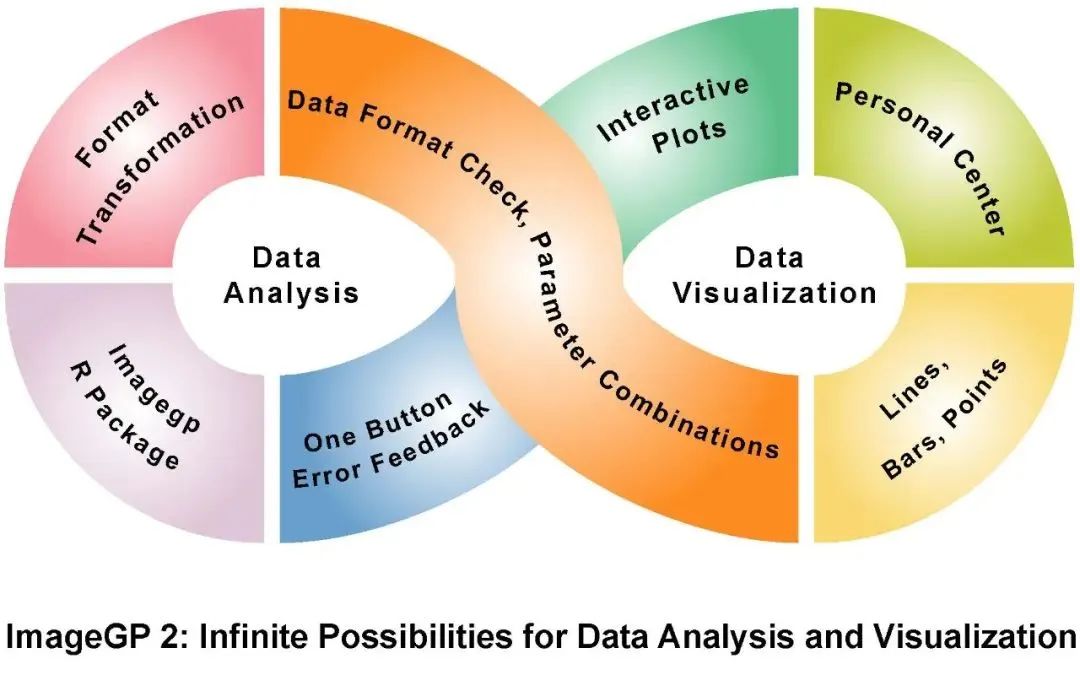
04 ImageGP 2.0:用于生物医学研究的强大数据可视化与可重复性分析工具
ImageGP 2 for enhanced data visualization and reproducible analysis in biomedical research
本文在升级版ImageGP 2.0(https://www.bic.ac.cn/BIC/#/)中,引入了最新网络开发技术,重新设计了用户使用界面,大大丰富和增强了原有功能,并显著改善了用户使用体验。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.239
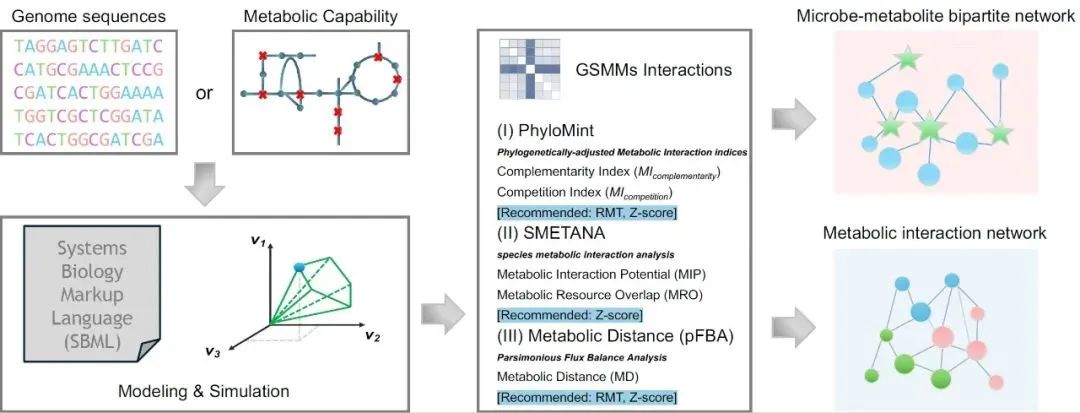
iNAP 2.0: Harnessing metabolic complementarity in microbial network analysis
本研究中提出了更新的iNAP2.0,其最重要的进步是整合了代谢模型的构建与分析模块,用于根据宏基因组测序数据进行微生物代谢互作的研究。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.235
06 使用PhyloSuite进行分子系统发育及系统发育树的统计分析
Using PhyloSuite for molecular phylogeny and tree-based analyses
https://onlinelibrary.wiley.com/doi/10.1002/imt2.87
07 OmicStudio: 可组合的高品质生物信息学云平台
https://onlinelibrary.wiley.com/doi/10.1002/imt2.85
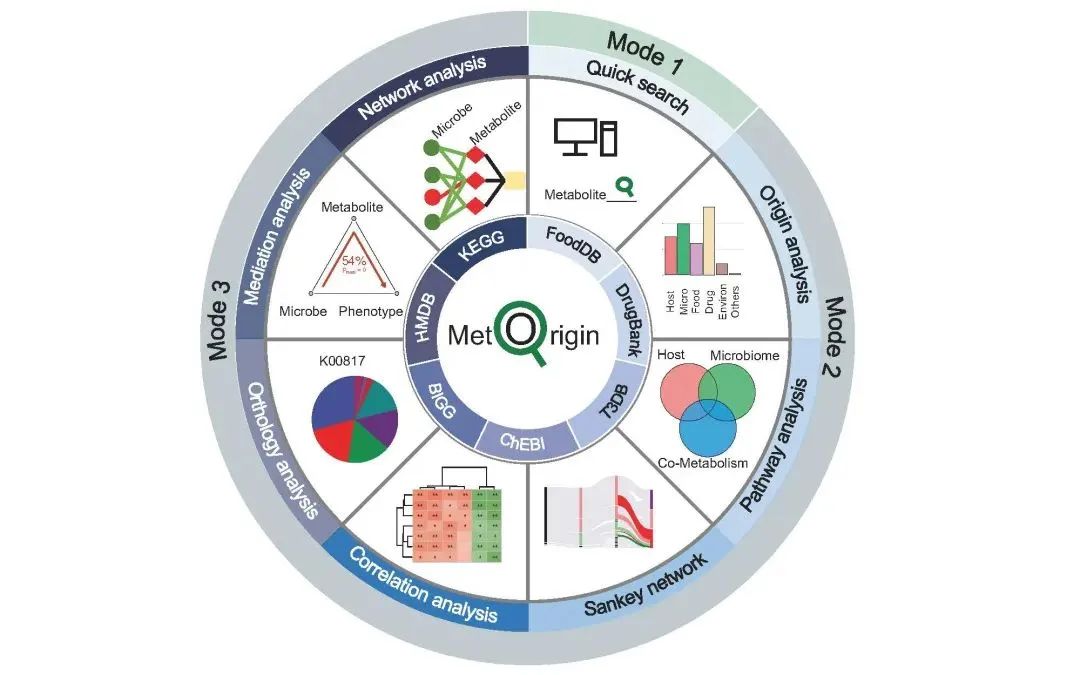
08 MetOrigin 2.0:推动微生物代谢物的发现和溯源
MetOrigin 2.0: advancing the discovery of microbial metabolites and their origins
https://onlinelibrary.wiley.com/doi/10.1002/imt2.246
09 易扩增子(EasyAmplicon):微生物组研究中易用的扩增子分析流程
https://onlinelibrary.wiley.com/doi/10.1002/imt2.83
Metabolite profiling of human-originated Lachnospiraceae at the strain level
本文构建了人源毛螺菌株资源库,包含148株来自33个属77个物种,对代表性毛螺菌科菌株进行体外挥发代谢谱表征,共检测到17类、242种在传统肠道代谢组学研究中鲜少关注的挥发性代谢产物。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.58
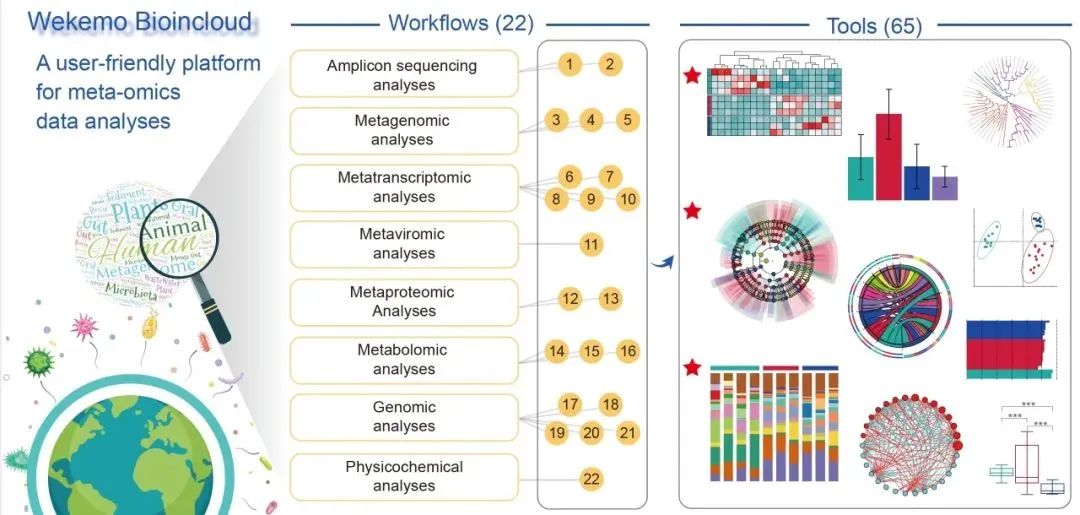
11 微科盟生科云(Wekemo Bioincloud):一个专为宏组学数据设计的用户友好型在线分析平台
Wekemo Bioincloud: A user-friendly platform for meta-omics data analyses”
https://onlinelibrary.wiley.com/doi/10.1002/imt2.175
12 盐度胁迫下失稳的微生物生态网络:丰富与稀有物种在维持生态网络中的差异化表现
本研究通过深入挖掘环境胁迫下的微生物生态模式,强调了在环境压力不断增加的人类世下平衡对丰富物种和稀有物种的保护策略以维持生态系统功能和服务的重要性。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.79
13 土壤微生物网络复杂性介导了土壤多功能性在降水改变下的变化
本研究结果提供了关于降水改变控制土壤功能的潜在机制的新见解。这些发现表明,地下微生物之间的相互作用应该被纳入未来气候变化情景下半干旱草地的土壤功能预测中。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.106
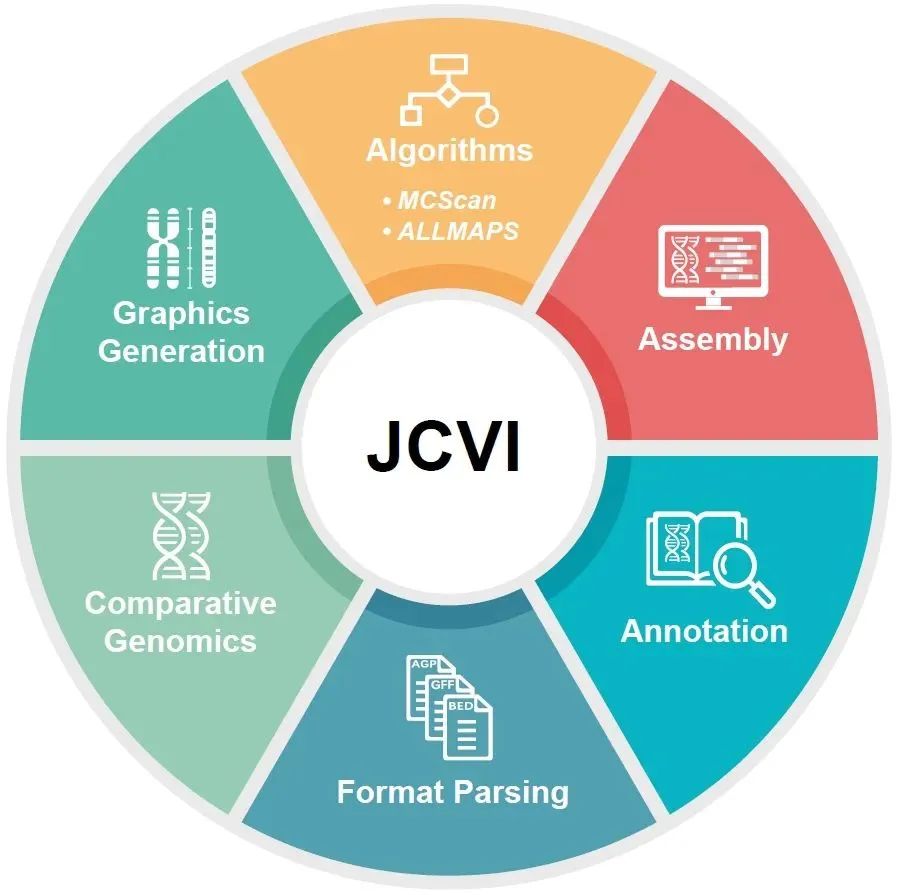
JCVI: A versatile toolkit for comparative genomics analysis
本研究开发了基于 Python 的多功能JCVI库,该库采用模块化设计,为格式解析、图形生成和基因组组装和注释操作等任务提供了非常实用的程序,使研究人员能够以更简洁的方式进行全面的基因组学分析。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.211
IPGA: A handy integrated prokaryotes genome and pan-genome analysis web service
https://onlinelibrary.wiley.com/doi/10.1002/imt2.55
SeqKit2: A Swiss army knife for sequence and alignment processing
本文介绍已被广泛使用的序列分析工具SeqKit新的版本SeqKit2,它具有更多的功能、更高的性能并增加对更多压缩格式的支持。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.191
17 新视角:高脂饮食介导的肠菌紊乱与慢性疾病的互作机制研究
New Insights into the Mechanisms of High Fat Diet Mediated Gut Microbiota in Chronic Diseases
https://onlinelibrary.wiley.com/doi/10.1002/imt2.69
MBPD: A multiple bacterial pathogen detection pipeline for One Health practices
https://onlinelibrary.wiley.com/doi/10.1002/imt2.82
19 shinyCircos‐V2.0: 易用性提高和功能增强的Circos图绘制工具
本研究开发的shinyCircos-V2.0是shinyCircos的升级版本,包括了一个新的用户界面,增强了其易用性,并提供许多用于创建高级Circos图形的新功能
https://onlinelibrary.wiley.com/doi/10.1002/imt2.109
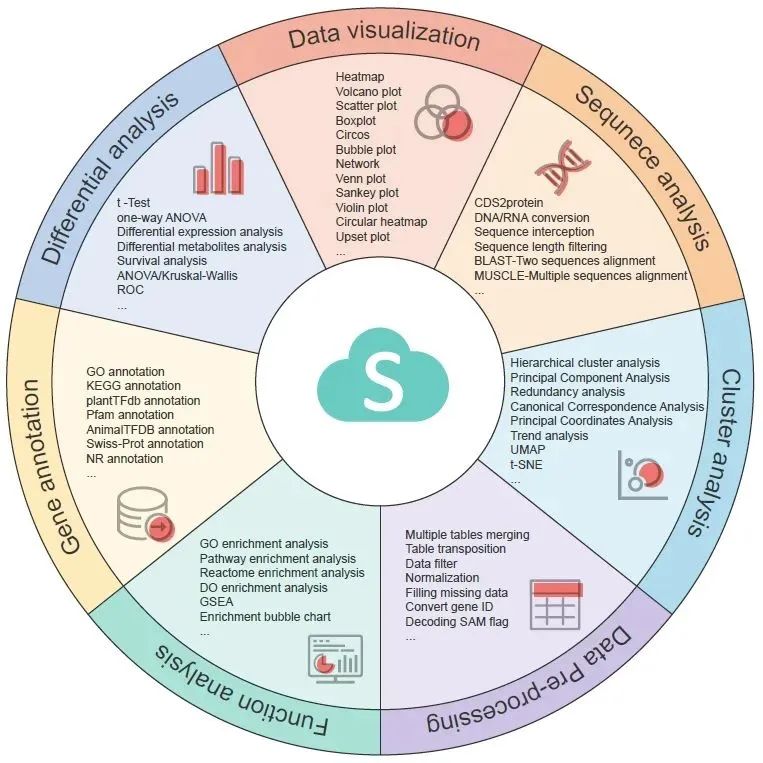
20 OmicShare tools: 零代码交互式的在线生物数据分析与可视化工具
本研究介绍的OmicShare tools是一个用户友好且高效的数据分析与可视化在线工具,包含161款生物信息分析小工具,主要用于生物数据特别是高通量测序数据的分析与可视化。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.228
Plastisphere microbiome: Methodology, diversity, and functionality
https://onlinelibrary.wiley.com/doi/10.1002/imt2.101
Research advances in probiotic fermentation of Chinese herbal medicines
https://onlinelibrary.wiley.com/doi/10.1002/imt2.93
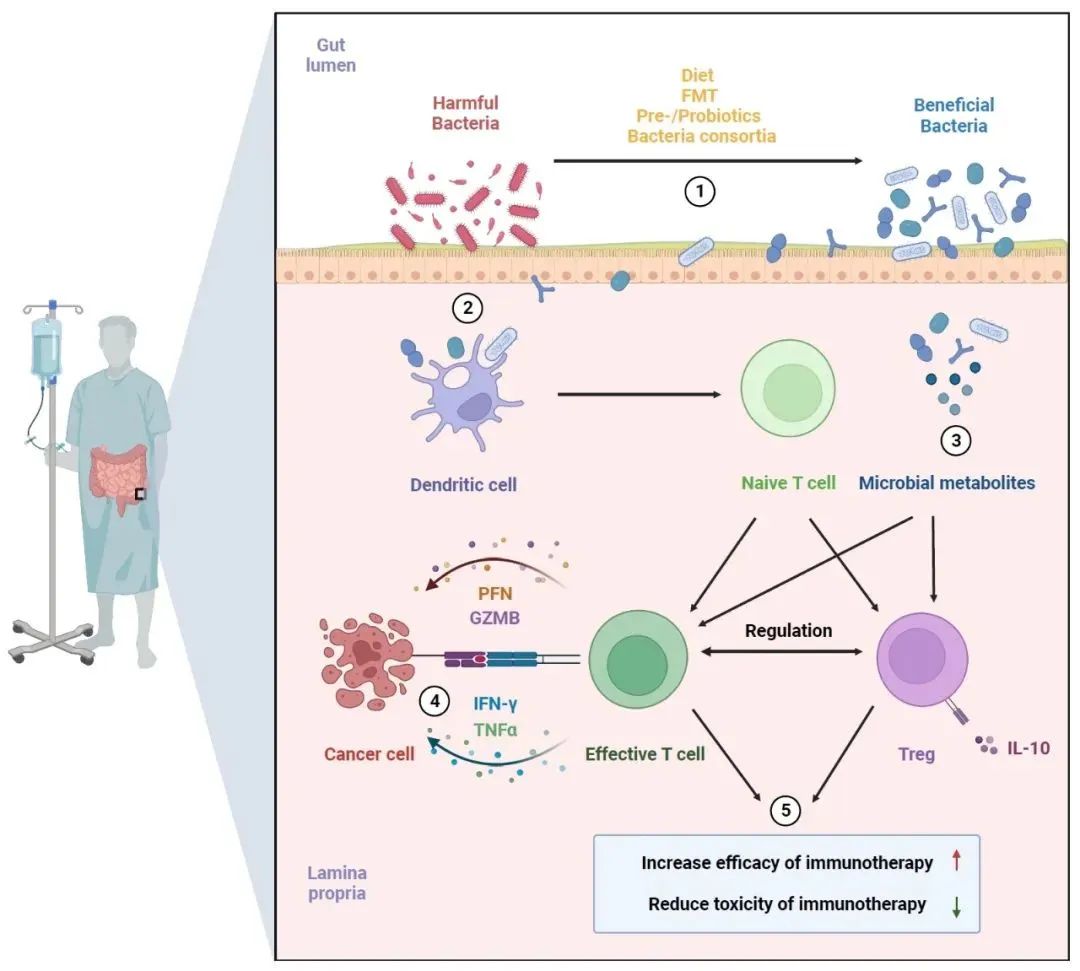
Gut microbiota reshapes cancer immunotherapy efficacy: Mechanisms and therapeutic strategies
本文回顾了微生物菌群组成及微生物菌群衍生的代谢物是否与免疫治疗反应和irAEs有关。此外,本文还讨论了通过调节微生物菌群组成来提高免疫治疗疗效或减轻毒性的各种方法。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.156
24 ViWrap:用于从宏基因组中识别、归类和预测病毒的宿主关系的模块化工具
https://onlinelibrary.wiley.com/doi/10.1002/imt2.118
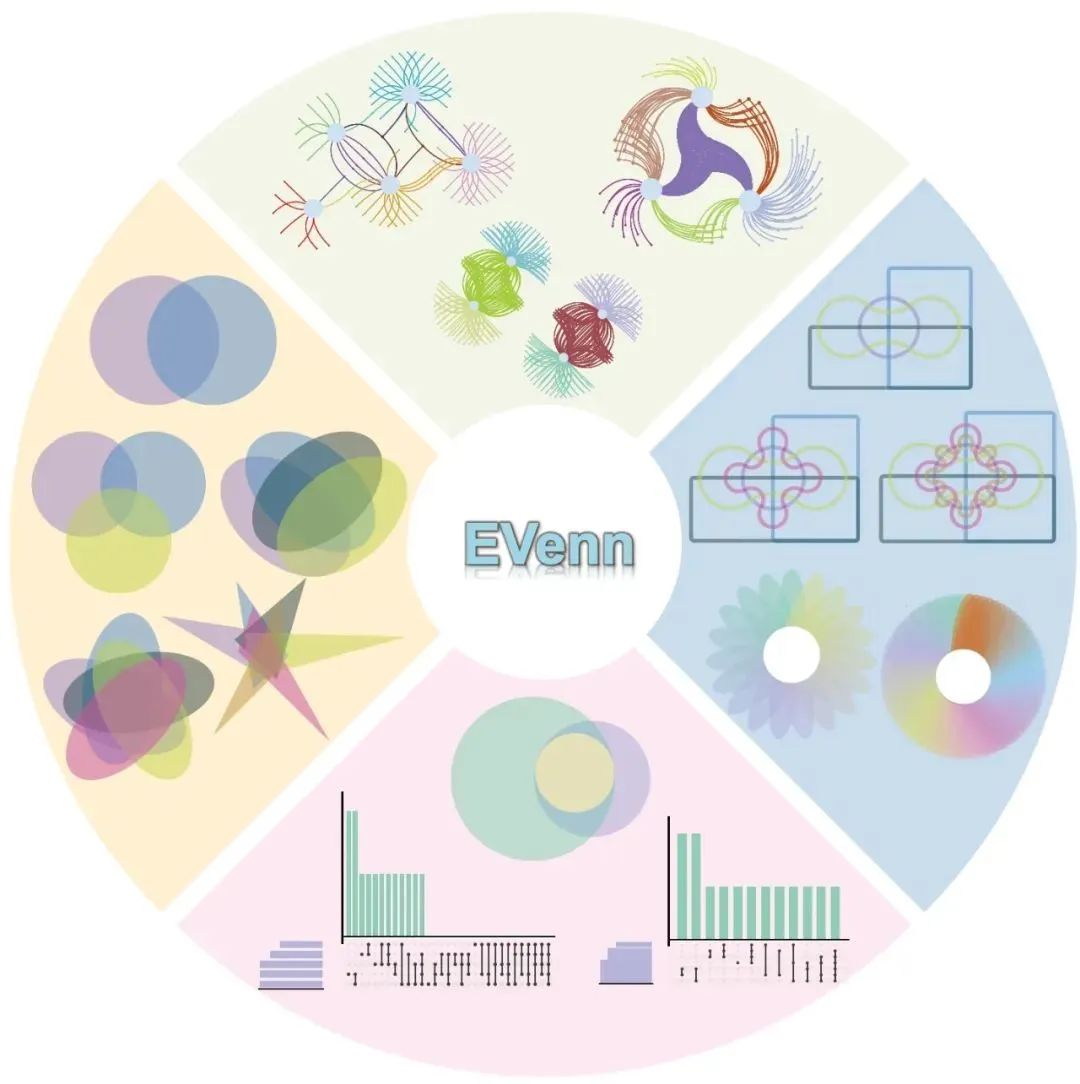
Visualizing set relationships: EVenn's comprehensive approach to Venn diagrams
本文概述了EVenn的各种应用,包括韦恩图的代表性结果解读、数据中心管理数据,多个案例演示。通过这些功能,使EVenn成为一个用户友好的集合关系探索工具,为深入探索多组学数据提供强大的支持。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.184
An overview of host-derived molecules that interacted with gut microbiota
https://onlinelibrary.wiley.com/doi/10.1002/imt2.88
27 火山极端环境下不同岩石表层土壤微生物组的模式和驱动因素
本研究以乌兰哈达火山群全新世玄武岩、全新世花岗岩和晚更新世玄武岩表层土壤为研究对象,采用Illumina MiSeq高通量测序分析细菌和真菌群落的组成和结构。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.122
28 定量微生物组分析揭示鸡肠道微生物发育轨迹及其与宿主代谢的关系
本研究以绝对定量的方式描绘鸡肠道微生物群发育过程,肠道微生物与血清代谢物之间的关联分析进一步凸显了肠道微生物对鸡生长发育的影响。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.105
Cultivation strategies for prokaryotes from extreme environments
本研究首先介绍了嗜极微生物的通用分离步骤,然后总结了多种从极端环境中获取纯培养原核微生物的策略,提出了一系列在微生物分离工作中容易被忽视但重要的技巧,并指出多组学指导的培养方式将在未来大有可为。
https://onlinelibrary.wiley.com/doi/10.1002/imt2.123
https://onlinelibrary.wiley.com/doi/10.1002/imt2.160
更多推荐
(▼ 点击跳转)
iMeta | 引用16000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 兰大张东组:使用PhyloSuite进行分子系统发育及系统发育树的统计分析
iMeta | 唐海宝/张兴坦-用于比较基因组学分析的多功能分析套件JCVI
iMeta封面
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
3卷1期
3卷2期
3卷3期
3卷4期
3卷5期
3卷6期

4卷1期
iMetaOmics封面
1卷1期
1卷2期
2卷1期
期刊简介
“iMeta” 是由威立、宏科学和本领域数千名华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表所有领域高影响力的研究、方法和综述,重点关注微生物组、生物信息、大数据和多组学等前沿交叉学科。目标是发表前10%(IF > 20)的高影响力论文。期刊特色包括中英双语图文、双语视频、可重复分析、图片打磨、60万用户的社交媒体宣传等。2022年2月正式创刊!相继被Google Scholar、PubMed、SCIE、ESI、DOAJ、Scopus等数据库收录!2024年6月获得首个影响因子23.8,位列全球SCI期刊前千分之五(107/21848),微生物学科2/161,仅低于Nature Reviews,学科研究类期刊全球第一,中国大陆11/514!
“iMetaOmics” 是“iMeta” 子刊,主编由中国科学院北京生命科学研究院赵方庆研究员和香港中文大学于君教授担任,是定位IF>10的高水平综合期刊,欢迎投稿!
iMeta主页:
http://www.imeta.science
姊妹刊iMetaOmics主页:
http://www.imeta.science/imetaomics/
出版社iMeta主页:
https://onlinelibrary.wiley.com/journal/2770596x
出版社iMetaOmics主页:
https://onlinelibrary.wiley.com/journal/29969514
iMeta投稿:
https://wiley.atyponrex.com/journal/IMT2
iMetaOmics投稿:
https://wiley.atyponrex.com/journal/IMO2
邮箱:
office@imeta.science






























































 6523
6523

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








