1、什么是亚细胞
细胞可以分成多个细胞器或者细胞区域,如细胞膜、细胞质、细胞核、线粒体、高尔基体、叶绿体、内质网等,这些细胞器被称为“亚细胞”。
蛋白质亚细胞位置示意图如下:

2、蛋白质亚细胞定位
把确定某种蛋白质或表达产物应在的亚细胞位置的过程称为“蛋白质亚细胞定位(Protein Subcellular Localization,PSL)
2.1蛋白质序列获取
可以在NCBI官网上进行下载https://www.ncbi.nlm.nih.gov/
首先,直接百度打开NCBI的网页,找到目标—protein

可以根据自己需求选择,这里我选了HIV-1, Human Protein Interaction Database
将页面拖到下方,看到URL的链接点击进去

然后就可以看到很多条信息,我们随便点击一条

点进去之后,找到fasta
点进去就可以看到序列,这样我们就获取到了我们所需的序列。(如果我们想要其fasta格式文件,首先复制粘贴下来,保存到文本文档中,将后缀名改为fasta就生成了fasta文件。)
2.2 WoLF PSORT——亚细胞定位
WoLF PSORT基于分选信号、氨基酸组成和功能motifs,例如DNA结合motifs,将蛋白质氨基酸序列转换为数值定位特征。转换之后,一个简单的k最近邻居分类器被用于预测。(2007年Paul Horton等人发表的一篇论文WoLF PSORT: protein localization predictor对其进行了详细介绍)
这里我们可以选择WoLF PSORT来进行我们数据的分析https://wolfpsort.hgc.jp/

根据你的需要选择你所要搜寻的种类,可选择文本粘贴和文件上传(fasta格式即可)。设置完成后即可运行程序。

等待一会出现下图——点击details
便出现结果如下——此图提供有关查询邻居的信息(WoLF PSORT训练数据中具有最相似定位特征的蛋白质)。为了方便用户,给出了百分比标识和每个邻居与查询对齐的链接。

通过在详细的结果页面上向下滚动,可以找到一个特性表,给出查询及其邻居的每个本地化特性的值。这些值被标准化为相对于WoLF PSORT训练数据的百分位数。蓝色显示的邻居值与查询值相差10%以内,而红色显示的邻居值与查询值相差20%以上。
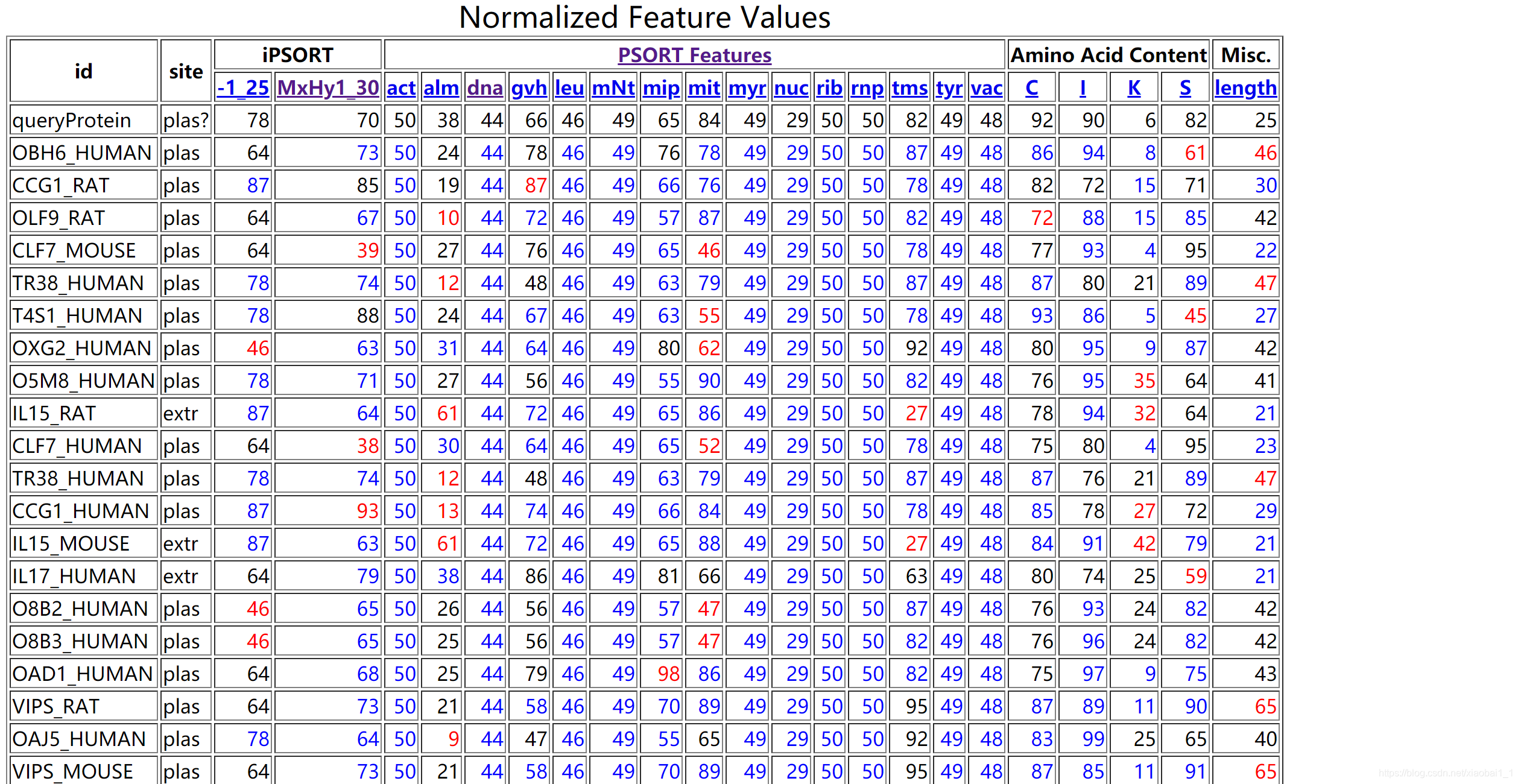
其中每一行代表一个基因(对于每个邻居基因,显示以下内容:UniProt ID、本地化站点、本地化功能与查询的距离、到查询的标识百分比、到其UniProt条目的链接、来自UniProt的亚细胞本地化行和其他可用的本地化信息。)

我们还可以点进去看具体信息——


UniProt
是 Universal Protein 的英文缩写,是信息最丰富、资源最广的蛋白质数据库。它由整合Swiss-Prot、 TrEMBL 和 PIR-PSD 三大数据库的数据而成。他的数据主要来自于基因组测序项目完成后,后续获得的蛋白质序列。它包含了大量来自文献的蛋白质的生物功能的信息。







 本文介绍了蛋白质亚细胞定位的概念,包括细胞的不同区域,如细胞膜、细胞核等。详细阐述了如何从NCBI获取蛋白质序列,并利用WoLF PSORT进行亚细胞定位预测。该过程涉及将蛋白质序列转换为数值特征,然后通过分类器预测其在细胞内的位置。
本文介绍了蛋白质亚细胞定位的概念,包括细胞的不同区域,如细胞膜、细胞核等。详细阐述了如何从NCBI获取蛋白质序列,并利用WoLF PSORT进行亚细胞定位预测。该过程涉及将蛋白质序列转换为数值特征,然后通过分类器预测其在细胞内的位置。
















 3万+
3万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








