更新:
複製了評論fromXiefocus: 建库的时候加入物种信息应该就好了吧,我是这样建库的,输出的时候选择-6格式,就能输出带有物种信息的比对表 diamond makedb --in db_nr/nr_plants/plants.nr.fa.gz --db db_nr/db_nr_plants/nr_plants --taxonmap db_nr/prot.accession2taxid.gz --taxonnodes db_nr/taxdump/nodes.dmp
原文:
需求:环境菌功能基因扩增子测序的OTU序列已经用diamond进行了NR全库的比对(blastx),还需得知其物种信息。
P.S.本人是没接触过扩增子比对相关内容,不保证该过程的合理性。
【流程主要参考这个,对于小白如我,该文很详细。本文也只是根据我的需求重新整理了这篇文章】一文完成nt库序列快速下载及blast结果注释物种 (qq.com)
【装所需文件主要参考这个】(20条消息) NR数据库的物种注释_songyi10的博客-CSDN博客
1. 安装所需软件(taxonkit和csvtk)
# 安装taxonkit
conda create -n taxonkit
conda activate taxonkit
conda install -c bioconda taxonkit
# 安装csvtk(也可以conda install -c bioconda安装,但我用的服务器突然镜像源有点问题就先直接装了(后来镜像源已解决))
# 进入自己放软件的文件夹 cd ./soft/csvtk
wget https://github.com/shenwei356/csvtk/releases/download/v0.25.0/csvtk_linux_amd64.tar.gz
mkdir -p $HOME/bin/; cp csvtk $HOME/bin/ 2. 下载NR数据库所附的物种信息和taxid信息
2.1 下载
下载NR物种相关信息和taxid信息的网站:
https://ftp.ncbi.nlm.nih.gov/pub/taxonomy/
https://ftp.ncbi.nlm.nih.gov/pub/taxonomy/accession2taxid
用wget下载文件(或者直接手动下载再复制进去,但挺大的,最好在服务器里下载 ),加-c可以断点续传。
# 我把他们全部放在数据库的文件夹
# cd ./db/nr
wget -c https://ftp.ncbi.nlm.nih.gov/pub/taxonomy/accession2taxid/prot.accession2taxid.gz
wget -c https://ftp.ncbi.nlm.nih.gov/pub/taxonomy/accession2taxid/prot.accession2taxid.gz.md5
wget -c https://ftp.ncbi.nlm.nih.gov/pub/taxonomy/taxdump.tar.gz
wget -c https://ftp.ncbi.nlm.nih.gov/pub/taxonomy/taxdump.tar.gz.md5
2.2 完整性检验
这里的两个.md5文件其实是用来做完整性检验的,如果下载那两个压缩包没有问题,不下载.md5文件且不做这步都没关系。
用md5sum和cat两个命令确认下下载的东西正常——如果两个结果一样,则文件完整,如下图(这个展示用的是另外的文件,不是我用的文件)。
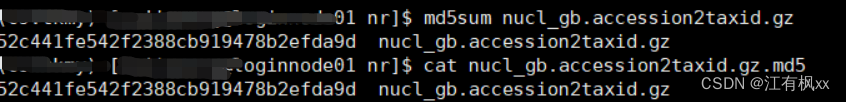
2.3 解压
tar -zxvf taxdump.tar.gz
gzip -d prot.accession2taxid.gz > prot.accession2taxid # 这个解压完41.79Ggzip解压可能会遇到“gzip: prot.accession2taxid already exists; do you wish to overwrite (y or n)”,输入y即可(但提交任务的话我不知道咋整),这样解压之后原本的.gz文件就消失了。
gzip -d和gunzip功能一样 。
解压出这些文件。
把names.dmp,nodes.dmp,delnodes.dmp,merged.dmp复制到taxonkit的默认路径下。根据其他博客的说明,names.dmp和nodes.dmp是必需的。
mkdir -p $HOME/.taxonkit
cp names.dmp nodes.dmp delnodes.dmp merged.dmp $HOME/.taxonkit复制(cp)完可以看到用户根目录的隐藏文件夹taxonkit下有这些文件(不用亲自去看啦,我就展示一下而已)。
![]()
3.提取细菌和古菌的taxid及其与物种的对应关系
运用软件:taxonkit
因为细菌和古菌的id号分别是2和2157,所以我只提--ids 2 ,2157。更详细的请去看我链接的那两篇文章。
taxid,顾名思义就是物种的id。
提取出来的taxid和taxonomy对应信息输出为一个文档prokaryotes_taxid_Ano.txt,之后可以手动(比如用Excel的vlookup或R语言的merge)与后面的生成的表格一起进行整理(5.中我还会再浅浅提及)。
# 建细菌和古菌的子库prokaryotes.taxid.txt
conda activate taxonkit
taxonkit list -j 2 --ids 2,2157 --indent "" > prokaryotes.taxid.txt
# 看看子库的基本信息(非数据处理过程)
wc -l prokaryotes.taxid.txt
head -n 5 prokaryotes.taxid.txt
# 提取taxid和taxonomy(界门纲目科属种)的对应信息到prokaryotes_taxid_Ano.txt
less prokaryotes.taxid.txt|taxonkit reformat -I 1 -r Unassigned -f "{k}\t{p}\t{c}\t{o}\t{f}\t{g}\t{s}\t{t}"| sed '1i\Taxid\tKingdom\tPhylum\tClass\tOrder\tFamily\tGenus\tSpecies\tStrain' > prokaryotes_taxid_Ano.txt
[更新] 其实并不需要这一步提取子库!!!!看下面第四个步骤的红字
获取物种分类信息的方法(TaxonKit/ete3/Biopython)-CSDN博客
4.taxid和subject的对应,并抽出自己比对结果的这部分taxid
把diamond比对后12行结果中的第二列(subject id)复制到一个单独的.txt文件phoDAAAA.txt(tab分割),且把它放在工作文件夹./db/nr(这不是一个好行为,只是为了方便权且这样做)。
运用软件:csvtk
如果用conda装的csvtk要记得激活一下csvtk的环境哦。
# 得到taxid和nr的genebank ID(accession.version)的对应表,即prokaryotes.acc.taxid2.txt
cat prot.accession2taxid |csvtk -t grep -f taxid -P prokaryotes.taxid.txt |csvtk -t cut -f accession.version,taxid > prokaryotes.acc.taxid2.txt
# 和自己的blast结果(subject)对应上,得到sseq.acc.taxid2.txt
# 相当于从上面得到的prokaryotes.acc.taxid2.txt里抽出自己要的子集。
# 在-P中输入自己比对得到的subject这列(phoDAAAA.txt)
cat prokaryotes.acc.taxid2.txt |csvtk -t grep -f accession.version -P phoDAAAA.txt |csvtk -t cut -f accession.version,taxid > sseq.acc.taxid2.txt
prokaryotes.acc.taxid2.txt文件形如这样
【更新】如果跳过了上一步的子库提取,可以将这里获得的sseq.acc.taxid2.txt进行这个操作,这样的话也可以简化第5步的数据整理:
进入taxonkit环境
运行下面的代码
cut -f 2 sseq.acc.taxid2.txt \
| taxonkit lineage \
| taxonkit reformat -F -P | cut -f 1,3 >Test_taxoid2list.tsv 得到的Test_taxoid2list.tsv 是taxid对应的物种注释(这就不需要自己vlookup或者merge了)
5. 后续自己进行信息的整理
下图是excel里用vlookup的图示。红字是对整个流程的一些总结。
R语言的merge函数也可以做类似操作,但少量数据就没必要了。
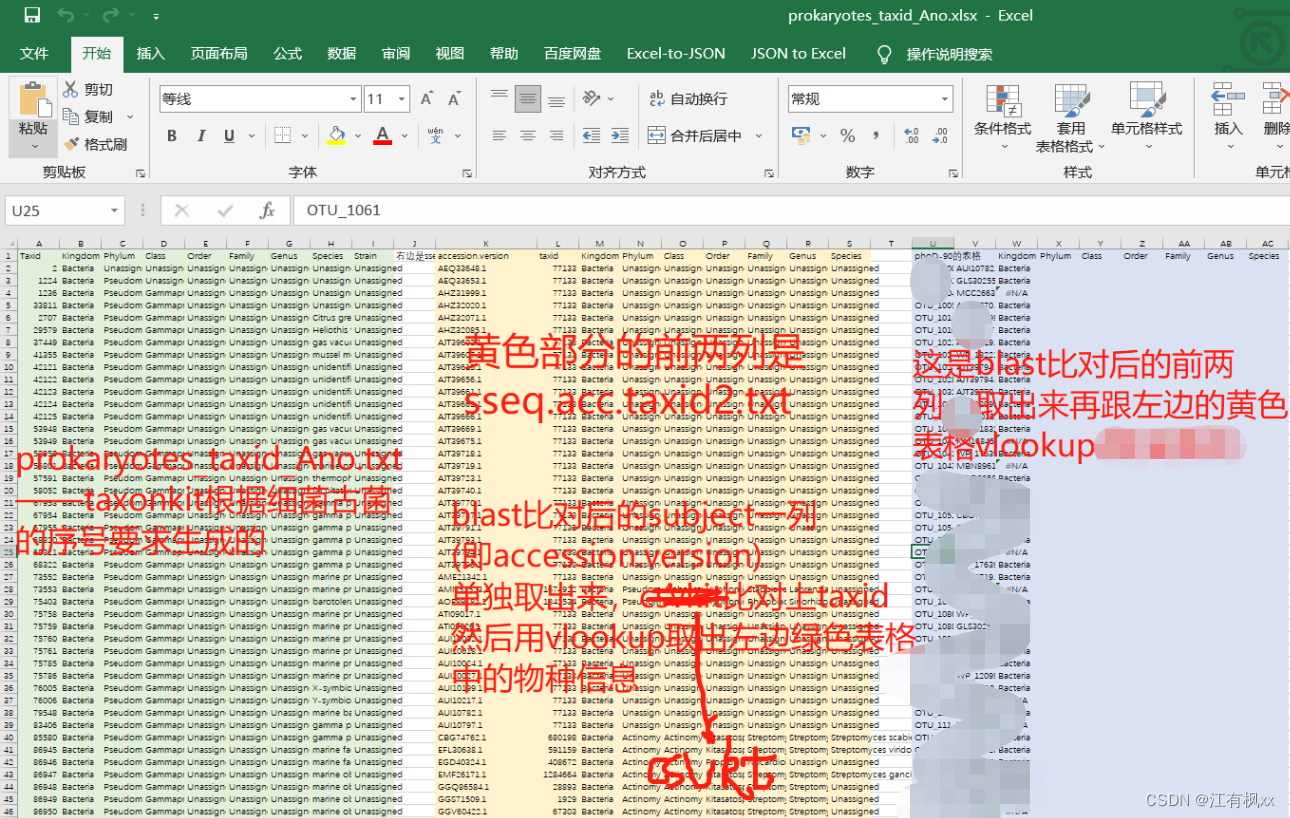
但这样的流程下来,还是有一些在NCBI查ID号就是NCBI里有信息的细菌,但这套流程注释不到的,不知道是哪里的问题。





















 9296
9296











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








