论文解读:《RELATION:A Deep Generative Model for Structure-Based De Novo Drug Design》
文章地址:https://pubs.acs.org/doi/full/10.1021/acs.jmedchem.2c00732
DOI:https://doi.org/10.1021/acs.jmedchem.2c00732
期刊:Journal of Medicinal Chemistry
2021年影响因子/JCR分区:8.039/Q2
出版日期:2022年6月17日
1.文章概述
基于深度学习的药物从头分子设计最近获得了相当大的关注。许多基于 DL 的生成模型已成功开发用于设计新分子,但其中大多数是以配体为中心的,并且目标结合口袋的 3D 几何形状在分子生成中的作用尚未得到充分利用。本文提出了一种新的基于 3D 的生成模型,称为 RELATION。在 RELATION 模型中,双向迁移学习(BiTL) 算法专门设计用于提取蛋白质-配体复合物的所需几何特征,并将其转移到潜在空间进行分子生成。应用药效团调节和基于对接的贝叶斯采样来有效地导航广阔的化学空间,以设计具有所需几何特性和药效团特征的分子。作为概念验证,RELATION 模型用于设计两个靶点 AKT1 和 CDK2 的抑制剂。计算结果表明,RELATION模型可以有效地生成具有良好结合亲和力和药效团特征的新型分子。
2.迁移学习相关知识点
2.1 迁移学习定义
迁移学习(Transfer Learning,TL):机器学习中的一个名词,是指一种学习对另一种学习的影响,或习得的经验对完成其他活动的影响。迁移广泛存在于各种知识、技能与社会规范的学习中。简单地说,迁移学习就是用在某个原始领域训练好的模型,去应用在未知的目标领域。
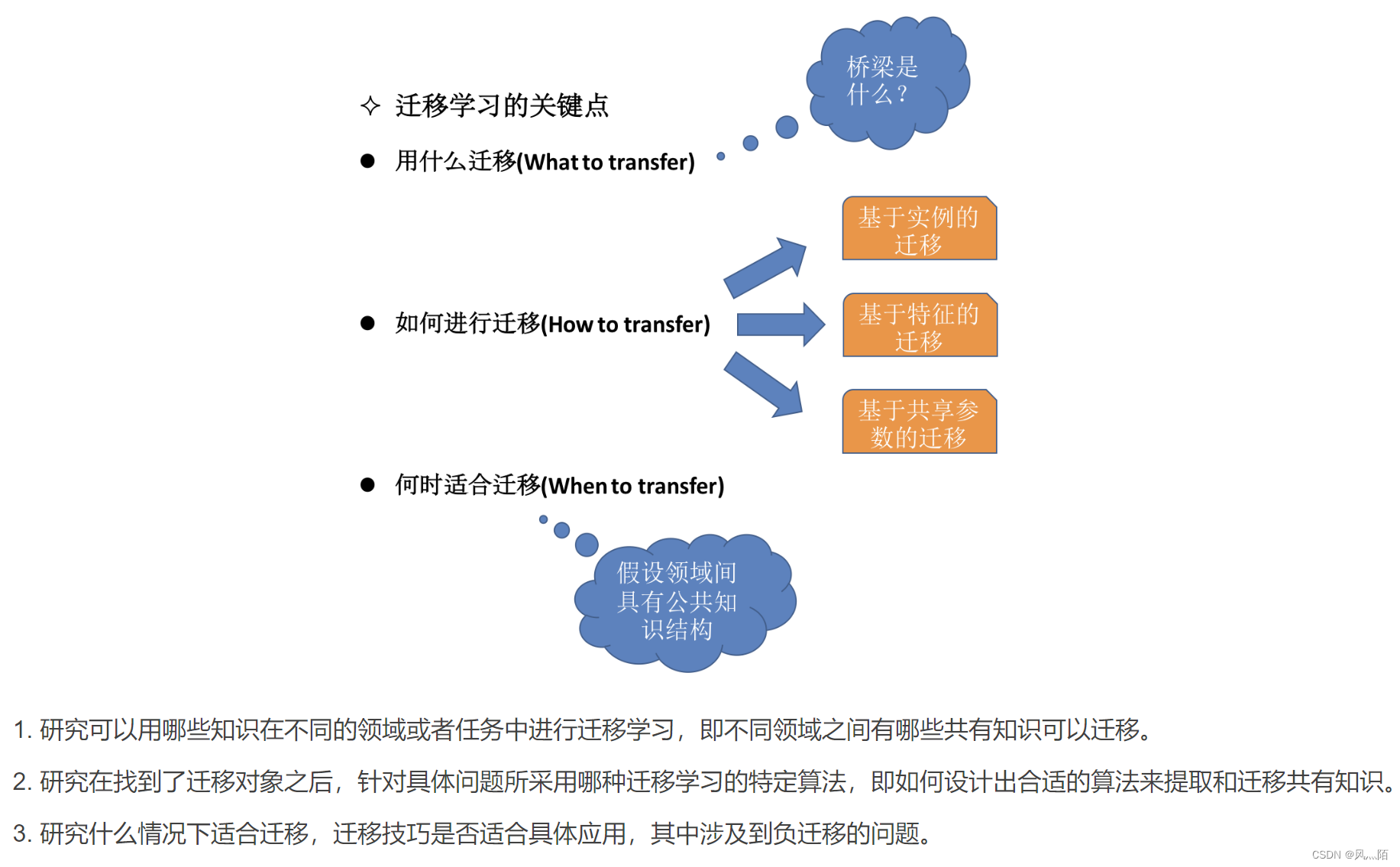
图参考:CSDN——迁移学习概述(Transfer Learning)
2.2 用法与不足
2.3 常见的迁移学习类型
详情请见:AI研习社——7种常见的迁移学习
- 领域自适应(Domain Adaptation)
- 在线迁移学习(Online transfer learning)
- 终身迁移学习(Lifelong transfer learning)
- 异构迁移学习(Heterogeneous Transfer Learning)
- 深度迁移学习(Deep Transfer Learning)
- 强化迁移学习(Reinforcement Transfer Learning)
- 对抗迁移学习(Adversarial transfer learning)
3.背景
药物发现的主要目标之一是鉴定具有所需药理特性的新型化学实体(先导化合物)。在药物发现早期鉴定出的候选药物,在后期临床试验中失败的风险很高(约95%),从而导致资源的浪费。高质量的先导化合物能够大大缩短药物探索的时间,提高成药的可能性。在先导化合物的设计过程中,要充分考虑候选分子的结构新颖性、生物活性、靶标选择性、化学可合成性、成药性和安全性等,这些性质直接影响药物开发的成败,因此先导化合物的发现一直是创新药物研发的主要瓶颈。通常,传统的从头药物设计方法可以分为两类:基于配体的和基于结构的。经典的基于配体的方法,需要一组经过实验验证的活性化合物作为起点,活性化合物共享的共同特征可以随后被利用来指导新型候选药物的设计。基于结构的方法,通过递增地添加、删除、插入或替换嵌入目标结合口袋中的化学支架的片段来创造新的小分子。 随着计算机硬件、软件和算法的飞速发展,高通量筛选、虚拟筛选和药物从头设计等计算机辅助药物设计技术开始取代传统方法,并大大缩短了先导物发现的时间和成本。从头药物设计目的是生成具有新颖结构的潜在候选药物。
全新药物设计与虚拟筛选技术不同,不依赖已有的化学数据库,可以通过不同的生成算法对类药空间进行更加深入的探索和发掘。传统的全新药物设计方法通常将遗传算法结合到药物从头设计中,尝试通过进化策略来优化生成的化合物结构。然而,传统的药物从头设计方法无法兼顾生成分子的新颖性与理想属性。基于深度学习(DL)的一些先进的生成算法已被用于新药设计,因为DL能够更高效地处理数据,对化合物属性深度特征的提取能力更强。主要有以下方法:编码-解码器(Encoder-Decoder,Enc-Dec)、循环神经网络(Recurrent Neural Network, RNN)、生成对抗网络(Generative Adversarial Networks,GAN)和强化学习(Reinforcement Learning,RL)。但大多数基于DL的生成算法都是以配体为中心的。因此,分子被表示为 1维(D) 的SMILES串或 2D 的分子图。1D 或 2D 表示的缺点是 3D 分子几何结构对配体结合的关键影响不能被生成模型捕获,不足以完成基于结构的药物从头设计中的特定目标任务。也有一些方法将配体或/和蛋白质结合的 3D 分子几何结构整合到基于 DL 的药物生成架构中,但这部分模型没有解决从头药物设计中的关键点,如配体结合模式和生成分子的药效团特征。
作者提出了RELATION(REceptor-LigAnd interacTION),这是一种基于Enc-Dec 的从头药物设计生成模型。与现有的DL方法不同,模型以具有原子物理化学性质的配体−受体复合体的三维网格构象为输入,因此,它在生成具有良好的药效团特征和结合模式的特定靶点的分子方面明显更高效。采用结构域分离网络(domain separation networks,DSN)进行双向迁移学习,来促进配体和配体−蛋白复合物之间的信息交换。为了不断提高模型的有效性,药效团的约束和基于对接分数的抽样分别由条件生成和对接分数抽样获得。最后,利用该模型设计了AKT1(protein kinase B alpha)和CDK2(cyclin-dependent kinases 2)的潜在抑制剂。
4.数据
RELATION模型的训练使用了源域和目标域两种数据集。
其中源域的数据集来源于ZINC数据库,除碳、氮、氧、硫、氟、氯、溴和氢之外,还含有带电原子或原子的分子被去除,选择分子量范围在200到600,logP(通过RDKit中的Crippen模型计算)在−2到6之间的分子,共包含1,200,000个小分子;从BindingDB、ChEMBL和PDB数据库中收集了407种AKT1抑制剂和1017种CDK2抑制剂(IC50< 50 nM),然后将两个靶点的抑制剂对接到靶标蛋白,只保留配体周围5 Å的原子作为蛋白配体复合物数据集。随后将源域数据集和目标域数据集放入7.57.57.5 Å3的网格中,并将源域数据集和目标域数据集的质心与立方体框的质心对齐,重原子的位置以1 Å作为分辨率,每个原子由19个物理化学性质描述。最后,源和目标数据集中的每个分子都由一个 4D 张量表示,该张量由其坐标(张量的前三个维度)和特征向量定义。为了检测旋转对 RELATION 模型的影响,作者构建了 10 个不同方向的 AKT1 源-目标数据集,其中 3D 网格中的每个数据点从旋转列表中随机旋转(3D 坐标9 次旋转,每个面对角线旋转6次,每个空间对角线旋转8次)。如果正确训练了RELATION 模型,它应该生成一个相似的分子集,而不用管源-目标数据集中差异的方向。

如图 7 所示,使用 LigandScout 4.4 从 AKT1(PDB ID:4GV1)和 CDK2(PDB ID:4KD1)的晶体结构中提取药效团特征,将 CVAE 架构引入药效团的 RELATION 模型约束生成。 CVAE中的药效团特征设置为“Relative Pharmacophore-Fit”,省略特征的最大数量设置为“1”。Relative Pharmacophore-Fit 分数 (rel.SFCR) 可以由下列公式定义,其中 rmsFP 是匹配特征对距离的均方根 (rms),NMFR 是几何匹配特征对的数量。

5.方法
5.1 模型架构

基于 VAE 的 RELATION 模型主要由两部分组成,如图 8 所示。第一部分是3D卷积编码器,包括私有编码器和共享编码器,第二部分是caption-LSTM解码器。训练数据转换为4D张量后,进入编码器通过八个隐藏层。第一层包含64个滤波器,然后在每个奇数层上加倍,最后一层包含512个滤波器。每个偶数层后面都有一个额外的池化层,内核大小、步幅和填充为2,以执行下采样。利用ReLU激活函数对3D-CNN模型进行训练,生成并合并一个1024维的嵌入向量。在最终输出层之前,采用三层LSTM解码器可以将隐藏层内的高维向量转化为SMILE分子式,其词汇量输入大小为39,隐藏大小为1024。
作者还试图通过将GAN框架引入架构来探索基于AAE的关系模型的有效性,在基于 AAE 的 RELATION 模型中,添加了一个额外的判别器,输入数据通过共享和私有编码器转换为潜码H,然后由解码器网络解码。另一方面,判别器将预测给定向量是由AAE生成的还是从N(0,1)中采样的向量。
5.2 损失函数

5.3 训练情况

用于对代理模型 f̂(x) 进行建模,并选择预期的改进作为获取函数 α。黑盒目标函数设置为 AutoDock Vina63 对接分数和支持向量回归 (SVR) 构建的 QSAR 函数的预测
SVR QSAR 函数是使用基于 Scikit-learn 的 RBF 内核构建的。 Pearson相关系数(R2)和交叉验证系数(RCV2)作为模型构建的评价标准。
从 BindingDB 收集的 657 种 AKT1 非抑制剂和 1714 种 CDK2 非抑制剂 (50 nM ≤ IC50 ≤ 10000 nM) 合并为两个抑制剂组。用作因变量的 pIC50 值被标准化为 0-1 的范围。使用薛定谔优化所有分子,然后使用 PaDEL-Descriptor 程序计算 797 个描述符。每个抑制剂组被随机分为训练集(90%)和测试集(10%)。训练集的 10 倍交叉验证用于评估构建的 SVR 模型。 GridSearchCV(网格搜索和交叉验证)算法用于优化参数 C(cost)、g(gamma)和 ε(insensitive loss function)。对于 AKT1-SVR 模型,训练集的 RCV2 和测试集的 R2 分别为 0.837 和 0.812。对于 CDK2-SVR 模型,训练集的 RCV2 和测试集的 R2 分别为 0.810 和 0.748。
整个 RELATION 模型使用 Adam 优化器进行优化,起始学习率为 10-4,训练 150 个 epoch 直到收敛。
5.4 评估标准


6.结果
6.1 生成分子的性质和质量

从表1中可以看到,可以发现具有双向TL的RELATION(AAE)和RELATION(VAE)模型比其他模型产生了更高的有效性、唯一性和内部多样性分数。FCD(Fréchet ChemNet距离)值可用于评估生成分子与参考数据集之间化学和生物性质的相似性。显然,TL模型产生的FCD值比非TL模型相对较低,表明使用TL技术时生成分子的化学和生物性质与现有抑制剂更相似。

根据图1中的非TL列可以看出,与现有抑制剂相比,VAE和对抗自编码器(AAE)生成的分子分布在完全不同的空间,当使用单向TL算法重新训练模型时,生成的分子分布与现有抑制剂之间的重叠明显增加。如图2中的双向TL列所示,RELATION(VAE)和RELATION(AAE)生成的分子几乎完全与现有抑制剂重叠,表明通过在VAE和AAE架构上使用双向TL,生成的分子和现有抑制剂覆盖了相似的化学空间。这些结果也与表1所示的数据(FCD值)一致。因此,随着双向TL算法和几何构象的应用,作者提出的RELATION模型可以捕获训练数据的潜在特征,RELATION模型在生成分子的有效性、唯一性、新颖性和多样性等方面的性能优于其他现有的基于3D的生成模型。
6.2 输入返校概念股对于结果稳定性的影响

作者使用了10个具有不同方向的AKT1源数据集-目标数据集(在生成分子的质量和属性部分中提到)来训练关系(AAE)模型。最终结果可以发现生成的分子集的质量没有表现出明显的变化。
6.3 RELATION 和基于药效团的 RELATION 的比较

作者进一步将条件VAE(CVAE)中的药效团特征引入到RELATION的训练中,以使生成过程更适合目标特异性任务。对比的RELATION是指基于AAE的RELATION(因为它在FCD指标方面的表现优于基于VAE的RELATION)。生成分子的药效团分数(rel.SFCR)的分布如图2所示。对于AKT1和CDK2,RELATIONpha(基于药效团的RELATION模型)生成的分子比原始RELATION模型生成的分子具有更高的药效团分数。这表明通过将药效团特征引入RELATION,生成的分子可以增强与预设药效团模型的匹配。
为了提高RELATIONpha模型的采样效率,并在显著较大的化学空间中生成一组具有良好对接构象的有效分子,作者还在模型中进入了贝叶斯优化(Bayesian Optimization,BO)的采样,如图3所示,生成分子的药效团分数得到了提高,其中BOdock的性能略优于BOqsar。BOdock生成的分子的对接分数与原始RELATION模型生成的分子相比有显著提高,但BOqsar生成的分子的QSAR分数仅略有变化。还发现,BOdock生成的分子表现出与现有抑制剂相似的药效团和对接分数分布。这些结果表明,BOdock产生的分子更有可能同时具有良好的药效团和对接特征,因此,BOdock可能是靶向特异性任务的更好选择。
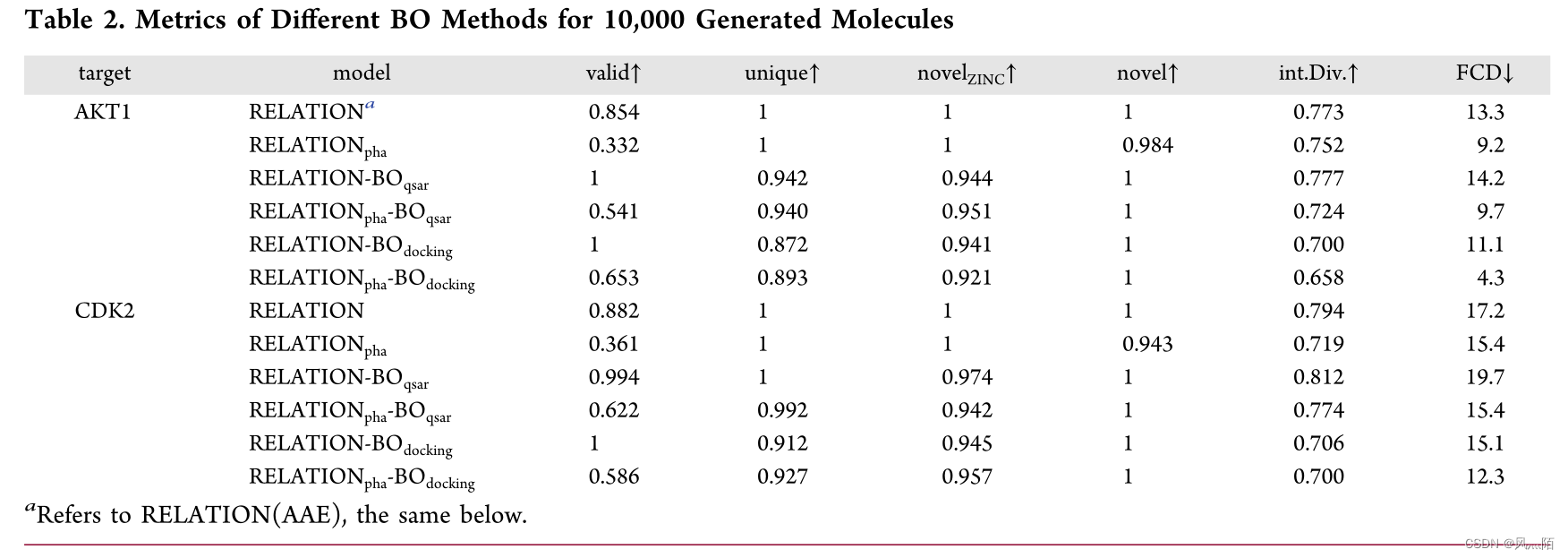
RELATION 和 RELATIONpha 的性能总结在表 2 中。根据 FCD 度量,作者发现 RELATIONpha 生成的分子的物理化学性质与现有抑制剂的物理化学性质更相似,这是由于将药效团特征引入了RELATION模型。然而,观察到由 RELATIONpha 生成的分子的有效性、新颖性和多样性下降并不奇怪。特别是,有效性从 ∼80% 下降到 ∼30%,这表明潜在空间中的采样对于 RELATIONpha 是非常低效的。这种无效性对于属性受限的 CVAE 架构是不可避免的。 整体低成功率可能是由于药效团性质和潜点之间的强相关性,潜点的离散性导致无效分子在从潜在向量到分子的解码过程。
6.4 RELATION模型的贝叶斯优化

以整个 ZINC 数据集作为潜在分子点的 BO 过程的采样是耗时的(为 BOdocking 生成 10,000 个分子约为 70 小时)。考虑到 Bemis-Murcko 支架仅占 ZINC 数据集中结构的一小部分(25%),作者试图通过缩小潜在分子点来提高采样过程的速度并提高生成分子的新颖性。从 Bemis-Murcko 支架组中随机选择 5%、10% 和 20% 的潜在分子点,然后对模型进行重新训练。使用整个 Bemis-Murcko 支架组 (25%) 和随机选择的 ZINC 数据集 (15%) 确定 40% 的潜在分子点。根据图 3 所示的新模型的性能,可以观察到,当 ZINC 潜分子点的大小在 5-20% 范围内时,生成分子的新颖性略有下降,但在大小增加到 40%,则急剧下降。因此可以推断,在潜在空间中引入重复数据确实会使BO方法倾向于对这些重复数据进行采样,导致新颖性不足。同时,图 3 中的有效性数据表明,当仅使用 5% 的数据时,生成的分子的有效性几乎达到最大值。


上述模拟数据表明,基于ZINC数据集的一小部分的BO采样可以产生与基于整个ZINC的数据集相似的结果。为了确定采样分子的质量与采样速度之间的平衡,我们进一步比较了不同采样尺度下生成的分子的质量,以选择适当数量的潜在分子点。如图 4 和 S3 所示,基于 BO 的 RELATIONpha 模型生成的分子(橙色,整个 ZINC 设置为潜在点X)比 RELATIONpha 生成的分子(蓝色)具有更有利的对接分数,并且更类似于现有的抑制剂。还可以观察到,仅使用 ZINC 数据集的 20% 作为潜在点㼿 可以产生与使用整个 ZINC 数据集几乎相同的性能。因此,选择 20% 的 ZINC 数据集作为 BO 中使用的潜在分子点的标准X用于以下研究。
6.5 BO采样过程对接结果
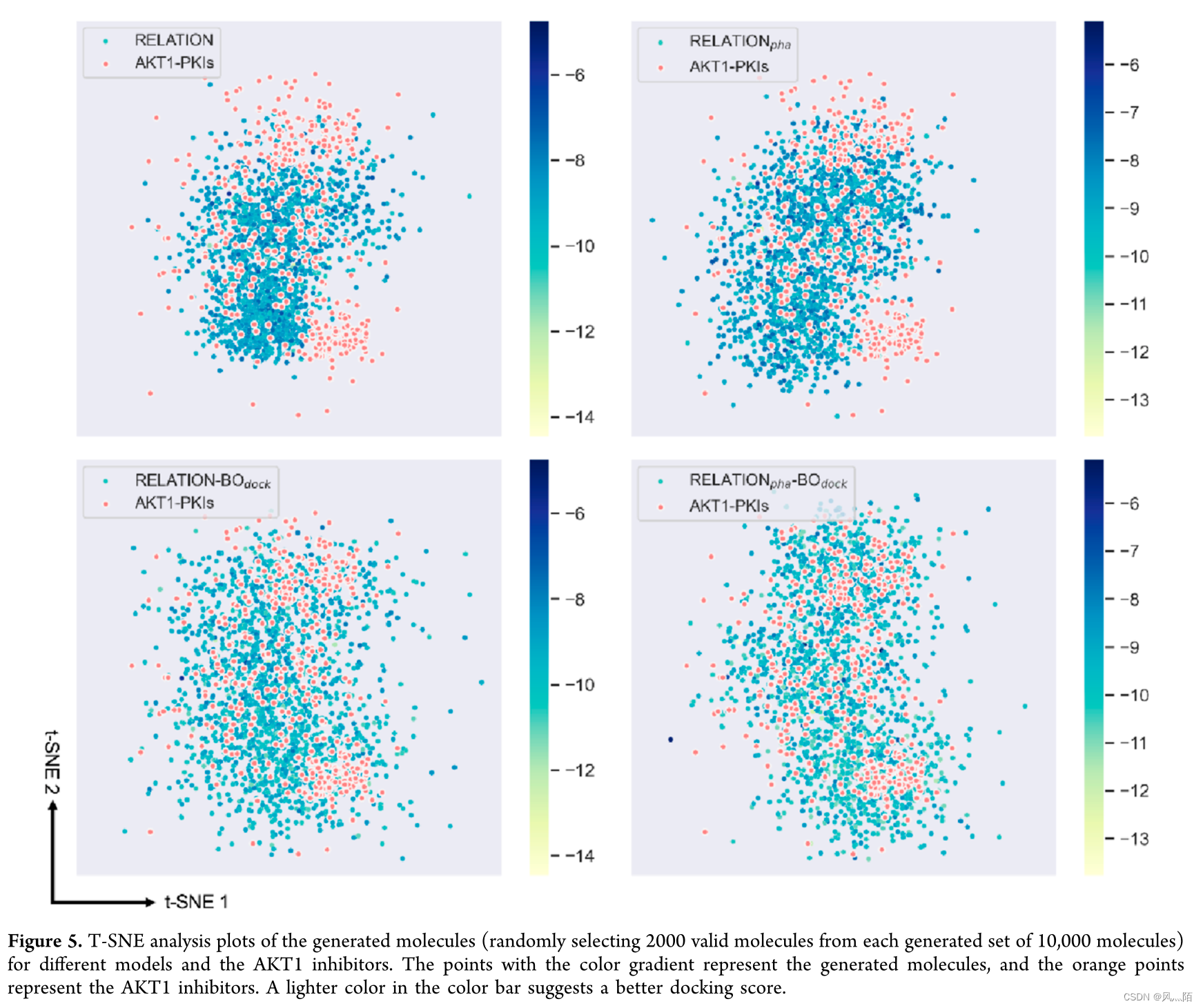

为了进一步研究基于BO采样的RELATION模型的性能,作者将不同模型生成的有效分子与AKT1和CDK2抑制剂再次进行了T-SNE分析。如图5和S6所示,RELATION和RELATIONpha模型不能有效地探索AKT1和CDK2抑制剂的化学空间(红圈中标记的点)。随着通BO-采样方式的引入,生成的分子在化学空间中的分布比原始RELATION更加分散,说明生成的分子与AKT1和CDK2抑制剂的化学空间更为相似。此外,根据点的颜色梯度,使用BO采样的RELATION模型生成的分子比原始RELATION模型生成的分子的对接得分更优。

如图6所示,可以发现生成的分子与AKT1抑制剂构象相同,朝向铰链区、酸孔和环区的方向相同,右侧的药效团特征也与理想的AKT1抑制剂一致。同时,对接和药效团评分结果表明,随着BO采样的引入,RELATION和RELATIONpha都能产生具有良好对接分数的分子,但BO-RELATIONpha产生的分子具有更高的药效团匹配分数。而且,通过将药效团特征引入RELATION架构,模型可以根据预设的药效团特征进行条件生成。
7.结论
本文提出的RELATION模型,可以生成新颖、有效、多样且亲和力高的分子。其中引入的双向TL在模拟现有抑制剂的理化性质方面比其他方法有更大的优势;为了能够创建具有所需特征的新分子,引入了药效团约束和基于BO的采样来优化RELATION,优化后的模型生成的分子与现有抑制剂具有更好的结合亲和力和更高的相似性,并具有更高的药效团匹配分数。总体来看,本文提出的RELATION模型在这些目标特异性任务中有效性较高。是一个基于结构的从头药物设计的有力工具。
参考
- https://mp.weixin.qq.com/s/peTD7iODMJCE2VNJ8-zmbw
- https://blog.csdn.net/dakenz/article/details/85954548
- https://zhuanlan.zhihu.com/p/481307870
- https://www.yanxishe.com/blogDetail/9561

























 8464
8464











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








